在使用机器学习进行训练前,往往需要大量数据及其标签。而对数据进行标注需要花费人力,因此未标注的数据容易获得,同时这些数据中也隐含了对分类有用的信息。
半监督学习(semi-supervised learning)可以结合已标注数据以及未标注的数据,以实现更准确的分类。
本文结合sklearn实现,介绍Label Spreading的具体过程。
1.Label Spreading
1.1 简介
Learning with Local and Global Consistency中提到,半监督学习满足两个一致性的先验假设,即:
- 1)附近的点倾向于拥有相同label,即Local Consistency;
- 2)同一结构(如同一cluster)内的点可能拥有相同label,即Global Consistency。
如果仅使用有标签的数据进行有监督学习,则有可能只满足第一个假设,Local Consistency。
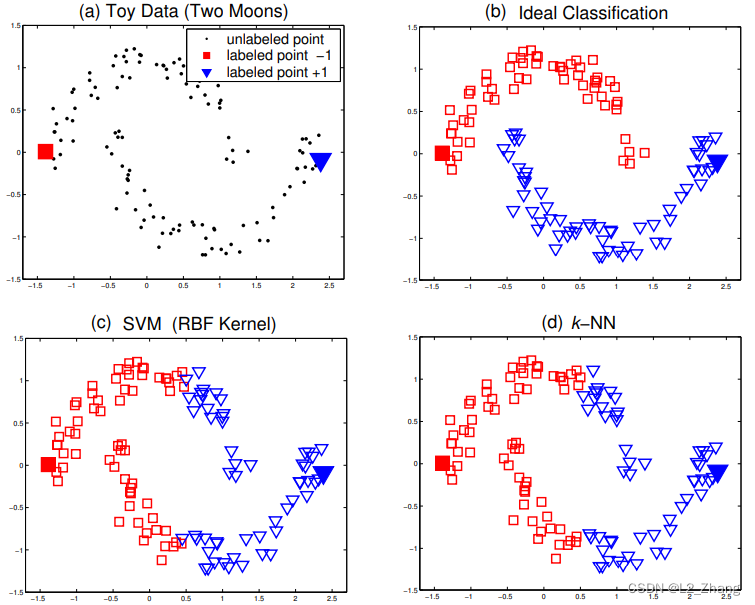
文中也举了一个例子,比如某个数据集,数据分布是2个月牙形,数据集中仅有2个数据标注过(2个类别各1个)。从满足Local 以及Global Consistency角度,我们知道理想的分类应该如图b)。而实际上当我们使用SVM或者k-NN(k取1),它们的结果都不好,只利用到了Local Consistency。
1.2 具体实现
设有一组数据 x 1 , x 2 , . . . , x n x_1,x_2,...,x_n x1,x2,...,xn,分为 c c c类,其中 x i ( i ≤ l ) x_i(i\le l) xi(i≤l)有label,剩下的数据点 x i ( l + 1 ≤ i ≤ n ) x_i(l+1\le i\le n) xi(l+1≤i≤n)没有标签。
-
1)构造矩阵 Y n ∗ c Y_{n*c} Yn∗c:当 x i x_i xi为第 j j j类时 Y i j = 1 Y_{ij}=1 Yij=1,否则为 Y i j = 0 Y_{ij}=0 Yij=0;
-
2)矩阵 F 0 = Y F_0=Y F0=Y;
-
3)构造邻接矩阵/关联矩阵(affinity_matrix) W n ∗ n W_{n*n} Wn∗n对角阵degree matrix D n ∗ n D_{n*n} Dn∗n,
W i j = exp ( − ∥ x i − x j ∥ 2 / 2 σ 2 ) \qquad W_{ij}=\exp({-\|{x_i-x_j}\|}^2/{2\sigma^2)} Wij=exp(−∥xi−xj∥2/2σ2);如果 i = j i=j i=j, W i j = 0 W_{ij}=0 Wij=0;
D i i = ∑ j W i j \qquad D_{ii}=\sum_jW_{ij} Dii=∑jWij;
S = D − 1 2 W D − 1 2 \qquad S=D^{-{\frac{1}{2}}} WD^{-{\frac{1}{2}}} S=D−21WD−21
-
迭代n_iters次:
F t + 1 = α S F t + ( 1 − α ) Y \qquad F_{t+1}= \alpha S F_{t}+(1- \alpha)Y Ft+1=αSFt+(1−α)Y; -
输出结果:
y i = arg max F i \qquad y_{i}= \argmax F_{i} yi=argmaxFi。
可以看出, α \alpha α、 S S S和 Y Y Y是固定不变的,每次迭代只更新 F F F。
另外,label spreading 迭代之前的过程是和谱聚类的部分内容是一致的,可参考一文GET Kmeans、DBSCAN、GMM、谱聚类Spectral clustering 算法。
1.3 代码及示例
以下代码均改编自skleran的官方实现。
1.3.1 构建数据集
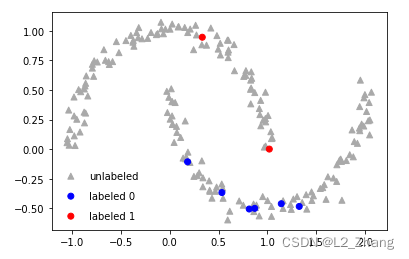
这里构建了一个双月形的数据集,设定标注过的数据只是极个别。
from sklearn.metrics.pairwise import euclidean_distances
from sklearn.neighbors import NearestNeighbors
from sklearn.base import BaseEstimator, ClassifierMixin
from scipy.sparse import csgraph
import numpy as np
from abc import ABCMeta, abstractmethod
from sklearn import datasets
import matplotlib.pyplot as plt
np.random.seed(123)
n_samples = 200
noisy_moons = datasets.make_moons(n_samples=n_samples, noise=0.05)
features = noisy_moons[0]
labels = noisy_moons[1]
c_s = ['r', 'b', 'darkgrey']
rng = np.random.RandomState(42)
# labels = labels[:]
random_unlabeled_points = rng.rand(len(labels)) < 0.95
labels = np.copy(labels)
labels[random_unlabeled_points] = -1
mk = ["^", "o"]
markers = np.array([mk[item] for item in labels])
colors = np.array([c_s[item] for item in labels])
plt.scatter(features[labels == -1][:, 0], features[labels == -1][:, 1],
c=colors[labels == -1], marker="^", label="unlabeled")
plt.scatter(features[labels == 1][:, 0], features[labels == 1]
[:, 1], c=colors[labels == 1], marker="o", label="labeled 0")
plt.scatter(features[labels == 0][:, 0], features[labels == 0]
[:, 1], c=colors[labels == 0], marker="o", label="labeled 1")
plt.legend(scatterpoints=1, frameon=False,
labelspacing=1, loc='lower left')
plt.show()
1.3.2 使用label spreading半监督学习
def rbf_kernel(X, Y=None, gamma=None):
if gamma is None:
gamma = 1.0 / X.shape[1]
K = euclidean_distances(X, Y, squared=True)
K *= -gamma
np.exp(K, K) # exponentiate K in-place
return K
class LabelSpreading:
def __init__(self, kernel="rbf", gamma=20,
alpha=0.99, max_iter=20, tol=1e-3,):
self.max_iter = max_iter
self.tol = tol
# kernel parameters
self.kernel = kernel
self.gamma = gamma
# clamping factor
self.alpha = alpha
def _get_kernel(self, X, y=None):
if y is None:
return rbf_kernel(X, X, gamma=self.gamma)
else:
return rbf_kernel(X, y, gamma=self.gamma)
def _build_graph(self):
"""Graph matrix for Label Spreading computes the graph laplacian"""
n_samples = self.X_.shape[0]
affinity_matrix = self._get_kernel(self.X_)
laplacian = csgraph.laplacian(affinity_matrix, normed=True)
laplacian = -laplacian
laplacian.flat[:: n_samples + 1] = 0.0 # set diag to 0.0
return laplacian
def predict(self, X):
probas = self.predict_proba(X)
return self.classes_[np.argmax(probas, axis=1)].ravel()
def predict_proba(self, X_2d):
weight_matrices = self._get_kernel(self.X_, X_2d)
weight_matrices = weight_matrices.T
probabilities = weight_matrices @ self.label_distributions_
normalizer = np.atleast_2d(np.sum(probabilities, axis=1)).T
probabilities /= normalizer
return probabilities
def fit(self, X, y):
self.X_ = X
# actual graph construction (implementations should override this)
graph_matrix = self._build_graph()
# label construction
# construct a categorical distribution for classification only
classes = np.unique(y)
classes = classes[classes != -1]
self.classes_ = classes
n_samples, n_classes = len(y), len(classes)
alpha = self.alpha
y = np.asarray(y)
unlabeled = y == -1
# initialize distributions
self.label_distributions_ = np.zeros((n_samples, n_classes))
for label in classes:
self.label_distributions_[y == label, classes == label] = 1
y_static = np.copy(self.label_distributions_)
y_static *= 1 - alpha
l_previous = np.zeros((self.X_.shape[0], n_classes))
unlabeled = unlabeled[:, np.newaxis]
for self.n_iter_ in range(self.max_iter):
if np.abs(self.label_distributions_ - l_previous).sum() < self.tol:
break
l_previous = self.label_distributions_
self.label_distributions_ = graph_matrix @ self.label_distributions_
# clamp
self.label_distributions_ = (
np.multiply(alpha, self.label_distributions_) + y_static
)
transduction = self.classes_[
np.argmax(self.label_distributions_, axis=1)]
self.transduction_ = transduction.ravel()
print(self.n_iter_)
colors = [c_s[item] for item in self.transduction_]
plt.scatter(self.X_[:, 0], self.X_[:, 1], c=colors)
plt.show()
return self
label_prop_model = LabelSpreading()
label_prop_model.fit(features[:], labels)
label spreading效果确实比较犀利:
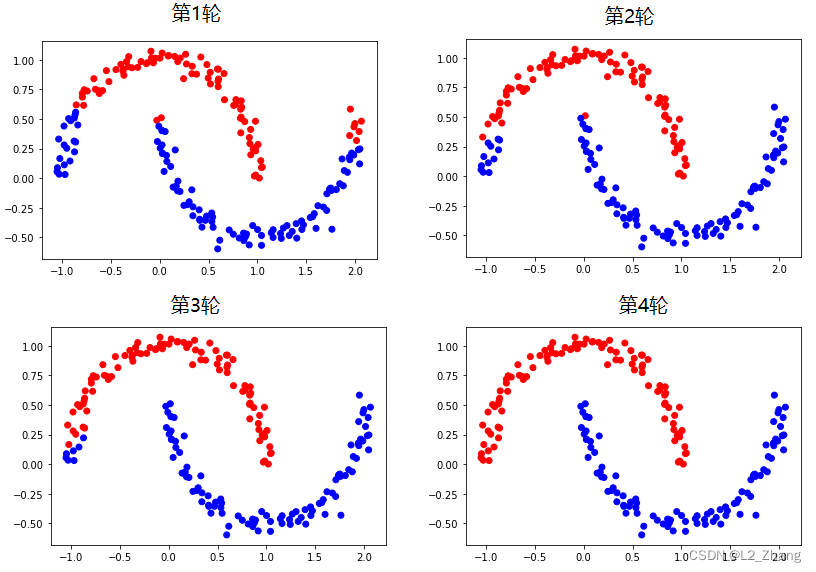
这里需要注意的是,
α
\alpha
α值设定对有的数据影响蛮大的,论文中推荐使用0.99.
参考文献
[1] Learning with Local and Global Consistency
[2] sklearn.semi_supervised.LabelSpreading
























 1万+
1万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










