会议回顾
胡琳艳,苗智超课题组
2025年1月17日,应Guangzhou RNA Club邀请,中国科学院生物物理研究所王艳丽教授作了题为 “The RNA-guided arms race between bacteria and phages” 的线上学术报告。王艳丽教授是国家“杰出青年科学基金”获得者、长江学者,长期从事CRISPR-Cas系统及其调控机制的研究,主要聚焦以下四个方向:(1) 第一类CRISPR-Cas系统的作用机理;(2) 第二类CRISPR-Cas系统的作用机理;(3) Anti-CRISPR蛋白对CRISPR活性的抑制机制;(4)小分子介导的基因沉默的结构生物学研究。
会议内容
在本次报告中,王艳丽教授围绕CRISPR-Cas 免疫系统的作用原理及其与噬菌体的对抗展开,重点介绍了 CRISPR-Cas 的分类、作用机制,以及细菌与噬菌体之间的进化博弈,特别是噬菌体如何通过 Anti-CRISPR(ACR)蛋白规避 CRISPR-Cas 系统的攻击,为 CRISPR-Cas 相关调控研究提供了新的视角。

图1: CRISPR-Cas系统及其进化军备竞赛
一、CRISPR-Cas 免疫系统的基础
1. CRISPR-Cas 系统概述
CRISPR-Cas 系统是一种 RNA 介导的获得性免疫系统,广泛存在于细菌和古细菌中 (图1)。它能够识别并降解入侵的外源 DNA,以抵御噬菌体感染。除此之外,CRISPR-Cas 由于其高度的靶向性,被发展为强大的基因编辑工具,在生命科学、医学和农业等领域发挥重要作用。
2. CRISPR-Cas 的作用机制
CRISPR-Cas 系统通过“记忆 - 识别 - 降解”的方式对抗噬菌体入侵,其作用过程可分为三个阶段 (图2):
适应 (Adaptation): 细菌在噬菌体首次入侵时,会截取一小段噬菌体 DNA 片段,并将其作为间隔序列 (spacer)整合到宿主 CRISPR 基因座中,相当于存储入侵者的“身份信息”。
表达 (Expression): 这些存储的 DNA 片段会被转录生成前体 crRNA (pre-crRNA),再经过加工形成成熟的 crRNA,携带间隔序列信息。
干扰 (Interference): 成熟 crRNA 结合 Cas 蛋白,形成效应复合物,一旦识别到匹配的噬菌体 DNA,就会触发核酸内切酶活性,靶向降解入侵 DNA,从而阻止噬菌体复制。

图2: CRISPR-Cas 系统的三大防御步骤
3. CRISPR-Cas 的分类
根据 Cas 蛋白的组成,CRISPR-Cas 系统被划分为两大类 (Class 1 和 Class 2) (图3):
第一大类 (Class 1)由多个 Cas 蛋白共同组成,与 crRNA 结合形成大型蛋白-RNA 复合物,包括 Type I、Type III 和 Type IV。其中 Type I 是最早被发现的 CRISPR-Cas 系统,也是自然界中分布最广泛的类型,约 90% 的 CRISPR-Cas 系统属于该类,主要依赖 Cas3 进行 DNA 降解。
第二大类 (Class 2)由单个大型 Cas 蛋白负责 crRNA 结合和 DNA 识别切割,结构更简洁,包括 Type II、Type V 和 Type VI。其中 Type II 是 CRISPR 研究中的重要类别,其代表性蛋白 Cas9 已被广泛应用于基因编辑技术,推动了生命科学和医学领域的发展。

图3: CRISPR-Cas 系统的分类及其类型特征
二、不同 CRISPR-Cas 系统的作用机制
1. Type I CRISPR-Cas (Class 1)
Type I CRISPR-Cas (Class 1)主要依赖 Cas3 进行 DNA 降解。该系统由多个 Cas 蛋白与 crRNA 形成大型复合物,首先识别带有 PAM (Protospacer Adjacent Motif)序列的靶 DNA (图4)。识别后,会招募 Cas3,Cas3 能够沿 DNA 移动并持续降解大片段 DNA (图5)。这种降解模式不同于 Type II (Cas9)系统的精准切割。

图4: Type I CRISPR-Cas 系统的工作机制

图5: Cascade 复合体招募 Cas3 切割目标 DNA
2. Type II CRISPR-Cas (Class 2)
相比之下,Type II CRISPR-Cas (Class 2)主要依赖 Cas9 进行 DNA 切割。Cas9 介导的免疫过程中 (图6, 图7),首先结合 sgRNA,形成蛋白-RNA 复合物。随后识别目标 DNA 并形成 R-loop 结构。最终,HNH 结构域和 RuvC 结构域分别切割互补链和非互补链,完成 DNA 断裂。

图6: Cas9 结构域及 DNA 切割机制

图7: Cas9 识别并切割 DNA 的动态过程
三、细菌与噬菌体的“军备竞赛”
尽管 CRISPR-Cas 赋予细菌强大的免疫能力,但噬菌体作为进化速度极快的病毒,也在不断演化出逃避免疫识别或直接抑制 CRISPR 活性的策略,使得细菌与噬菌体之间形成了一场持续的“军备竞赛”。在这一竞争过程中,噬菌体不断优化自身基因组,以逃脱 CRISPR-Cas 免疫系统的监测,而细菌则不断优化 CRISPR-Cas 机制,以维持其防御能力。噬菌体的主要对抗策略包括两种:
(1)突变免疫逃逸:噬菌体通过基因组突变,改变其 DNA 序列,使其不再被 CRISPR-Cas 识别,从而成功逃避降解。这种策略依赖于噬菌体高突变率,能够在短时间内适应细菌的免疫防御。
(2)Anti-CRISPR (ACR) 蛋白抑制:噬菌体进化出 Anti-CRISPR(ACR)蛋白,能够直接抑制 CRISPR-Cas 复合物的功能,阻止其识别、靶向或降解噬菌体 DNA。ACR 蛋白在噬菌体与宿主的长时间博弈中逐渐演化而来,成为噬菌体抵抗 CRISPR-Cas 免疫的关键武器。
四、噬菌体的 Anti-CRISPR (ACR)机制
ACR 蛋白是噬菌体对抗 CRISPR-Cas 免疫系统的重要策略,它们通过不同方式阻碍 CRISPR-Cas 的活性,针对不同类型的 CRISPR-Cas 体系,ACR 蛋白的抑制策略也有所不同。目前,研究表明,ACR 主要通过以下方式抑制 Type I(Class 1)和 Type II(Class 2)CRISPR-Cas 系统。
1.ACR 对 Type I CRISPR-Cas 的抑制机制
Type I CRISPR-Cas 系统主要依赖 Cascade 复合物(由多个 Cas 蛋白与 crRNA 组成)识别靶 DNA,并通过 Cas3 进行 DNA 降解。ACR 蛋白的主要抑制策略是干扰 Cascade 复合物的组装或阻断 Cas3 的招募,从而削弱 CRISPR-Cas 的免疫能力。
(1)AcrIF25: 破坏 Cascade 复合物的稳定性,使 CRISPR-Cas 失去识别与降解功能 (图8)。
在 Type I CRISPR-Cas 系统中,Cascade 复合物由多个 Cas 蛋白 (包括 Cas7)与 crRNA 共同组成,能够精确靶向噬菌体 DNA,并招募 Cas3 进行降解。而 AcrIF25 通过特异性结合 Cas7,破坏 Cascade 复合物的结构,使其无法稳定结合 crRNA 和靶 DNA。这种结构的不稳定性导致 crRNA 无法精准匹配目标序列,进而影响 Cas3 的招募,使 CRISPR-Cas 复合物 丧失降解噬菌体 DNA 的能力,最终让噬菌体成功规避宿主免疫系统。

图8: AcrIF25 与 Cas7 结合,影响 CRISPR-Cas 系统功能
(2)AcrIF3: 通过阻断 Cas3 的招募,抑制 CRISPR-Cas 介导的 DNA 降解 (图9)。
在 Type I CRISPR-Cas 免疫过程中,Cascade 复合物在识别到靶 DNA 后,需要招募 Cas3 作为核酸酶执行 DNA 降解。而 AcrIF3 通过直接结合 Cas3,阻止其与 Cascade 复合物相互作用,使 Cas3 无法被正确招募。由于 Cas3 无法执行降解任务,即便 Cascade 复合物仍然能够识别噬菌体 DNA,宿主也无法清除入侵的外源基因组,从而使噬菌体 DNA 得以复制,成功逃避免疫攻击。

图9: AcrF3 阻止 Cas3 被 Cascade 复合体招募
2.ACR 对 Type II CRISPR-Cas 的抑制机制
Type II CRISPR-Cas 系统依赖 Cas9 进行 DNA 识别和切割,ACR 蛋白的抑制策略主要针对 sgRNA 结合、DNA 结合位点竞争、R-loop 结构形成及 Cas9 的构象变化等关键环节。
(1)AcrIIC2: 结合 Cas9 的 Bridge Helix(BH)结构域,阻止 sgRNA 结合,使 CRISPR-Cas 丧失 DNA 识别能力 (图10)。
Cas9 需要依赖 sgRNA 结合并引导 DNA 识别,其中 BH 结构域(Bridge Helix)是 Cas9 结合 sgRNA 的关键 α 螺旋结构。AcrIIC2 通过结合 Cas9 的 BH 结构域,形成二聚体,占据 sgRNA 结合位点,使 Cas9 无法加载 sgRNA,进而阻止 DNA 识别。这种抑制作用直接作用于 CRISPR-Cas 免疫过程的最早阶段,使 Cas9 在免疫反应的初始阶段就处于完全非活性状态,最终使细菌失去对噬菌体 DNA 的防御能力 (图10)

图10: AcrIIC2 结合 BH 阻碍 Cas9 激活
(2)AcrIIC5: 通过竞争 Cas9 的 DNA 结合位点,使 Cas9 误结合错误靶点,阻止其识别真实的目标 DNA (图11)。
在正常情况下,Cas9 通过识别 PAM(Protospacer Adjacent Motif)序列进行 DNA 结合,而 AcrIIC5 由于表面带有类似双链 DNA(dsDNA)的酸性区域,能够模拟 DNA 结构,与 Cas9 结合,使其误认为 AcrIIC5 是目标 DNA。被误导的 Cas9 无法识别真正的噬菌体 DNA,导致 CRISPR 免疫系统无法启动降解程序,从而使噬菌体成功逃避攻击。

图11: AcrIIC5 通过模拟 DNA 阻止 Cas9 识别靶 DNA
(3)AcrIIC4: 通过阻碍 R-loop 结构的形成,使 Cas9 处于非活性状态,无法执行 DNA 切割 (图12, 图13)。
在正常的 CRISPR 免疫过程中,Cas9 识别 DNA 后,sgRNA 会与靶 DNA 形成 R-loop 结构,这一结构的稳定形成是 Cas9 介导 DNA 切割的关键步骤。而 AcrIIC4 通过嵌入 Cas9 REC1 和 REC2 结构域之间的缝隙,阻止 Cas9 结构域的运动,使 Cas9 无法完成构象变化。由于 Cas9 结构受到抑制,R-loop 结构无法正确形成,Cas9 也无法完成 DNA 断裂,最终噬菌体 DNA 得以存活并复制。
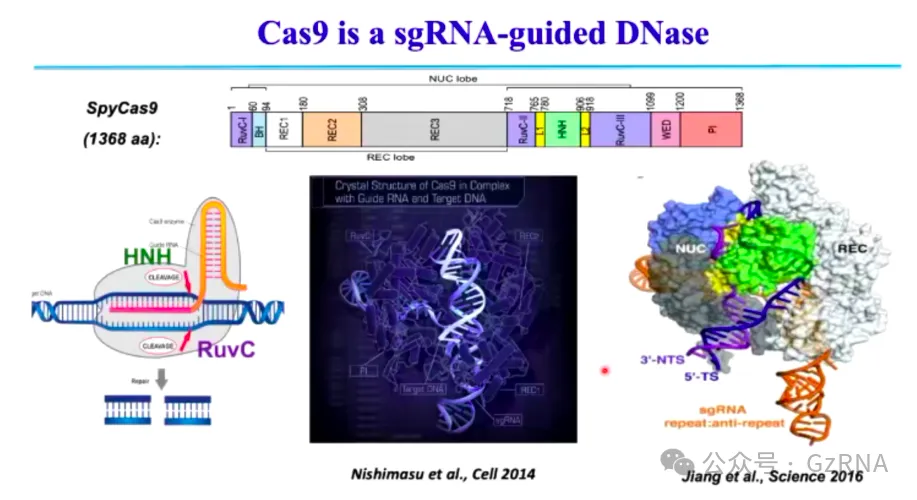

图12: AcrIIC4 限制 REC2 运动,阻止 R-loop 形成

图13: AcrIIC4 嵌入 REC1 和 REC2 之间抑制 Cas9
(4)AcrIIC3: 结合 Cas9 的 HNH 结构域和 R2 结构域,锁定 Cas9 在非活性构象,使其无法切割 DNA (图14)。
Cas9 在执行 DNA 切割时,HNH 结构域需要进行大幅度旋转,从非活性状态转变为活性状态,才能执行精确的 DNA 切割。而 AcrIIC3 通过结合 Cas9 的 HNH 结构域和 R2 结构域,形成稳定的二聚体,使 Cas9 结构域无法发生必要的构象变化。这种分子锁定机制使 Cas9 即便已经结合 sgRNA 和 DNA 仍无法激活核酸酶活性,从而阻止 DNA 降解,使噬菌体 DNA 免受 CRISPR-Cas 免疫系统的攻击。

图14: 不同 ACR 蛋白抑制 Cas9 的多种机制
总结
CRISPR-Cas 系统在细菌和古细菌中作为重要的免疫防御工具,其多样化的作用机制赋予了它强大的适应性。然而,噬菌体也在不断进化,通过突变逃逸和 Anti-CRISPR 机制对抗 CRISPR-Cas,形成了生物学上的“军备竞赛”。这些研究不仅加深了我们对 CRISPR-Cas 免疫系统的理解,也为基因编辑技术的改进和 CRISPR-Cas 相关调控策略的开发提供了新的研究方向。
精彩问答
问题1:噬菌体的抑制机制为何要组装好后又拆除?这种机制是否用于维持平衡?
答:噬菌体编码的蛋白,如 F25,能够抑制 CRISPR-Cas 系统,从而抵抗宿主的免疫机制。这种机制类似于病毒与宿主之间的对抗关系,如新冠病毒与人体免疫系统的互相适应。
问题2:抑制 CRISPR-Cas 的因子主要是来自噬菌体的基因组吗?
答:目前研究发现,大多数抑制因子都是噬菌体编码的蛋白,但也有一些 RNA 分子可以模拟 CRISPR-Cas 结构并进行抑制。
问题3:anti-CRISPR 机制在不同阶段的作用强度如何?在哪个阶段抑制效果最好?
答:最有效的抑制方式是干扰 CRISPR-Cas 复合物与 DNA 的结合。这一机制已被多个不同的 anti-CRISPR 蛋白所利用,尽管它们的具体作用机制各不相同。
问题4:在人类和哺乳动物细胞中进行 CRISPR 基因编辑时,是否存在类似的 anti-CRISPR 机制?
答:哺乳动物细胞自身并不具有天然的 anti-CRISPR 机制,但可以通过人为设计来调控 CRISPR 编辑,例如设计调控 CRISPR 活性的蛋白或 RNA 分子,从而实现对基因编辑的精确控制。
问题5:AI 是否可以用于预测新的 anti-CRISPR 蛋白?目前对 anti-CRISPR 蛋白的研究是否还不完整?
答:目前已知的 anti-CRISPR 蛋白种类仍然较少。自 2016 年首次发现以来,研究进展迅速,但仍有许多未知的机制。AI 可以用于预测更多可能的 anti-CRISPR 蛋白,并帮助发现新的作用模式。
问题6:细菌自身是否存在针对 anti-CRISPR 的调控机制?
答:细菌确实存在调控 anti-CRISPR 的机制。例如,一些蛋白可以结合 anti-CRISPR 蛋白的启动子,抑制其表达,从而调控 CRISPR-Cas 系统的活性。这可能有助于维持宿主与噬菌体的平衡。
问题7:CRISPR-Cas 系统在解析 MEME 与 Cas9 复合物的结构时,是否能观察到完整的 HNH 结构?
答:由于研究采用的是晶体结构解析技术,因此能够以高分辨率解析 HNH 结构域的每个原子,包括结合的金属离子,所有氨基酸侧链的密度都吻合良好。
问题8:这种 HNH 结构是如何捕捉到的?是在样品制备过程中获得的稳定状态,还是通过构象分类发现的瞬时状态?
答:研究人员在制备过程中采用了突变小蛋白,并结合特定的实验条件,从而成功捕捉到了 HNH 结构的活性状态,虽然其中有一定的运气成分,但实验设计在稳定性控制方面发挥了重要作用。
问题9:anti-CRISPR 蛋白的不同类型是否存在进化关系?它们的结构域是否具有保守性?
答:anti-CRISPR 蛋白具有极高的多样性,不同种类之间的结构域并没有明显的保守性。这种多样性类似于病毒的进化模式,即生物越低等,其基因和蛋白的多样性就越高。
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








