一、数据预处理
(一)、数据质量控制主要包括:检查原始数据质量,包括测序深度、序列长度、测序质量等参数,保证同一分析的数据拥有相似的测序参数,便于后续指标计算和统计分析。思影将提供各样品和组间的数据质量报告,并为后续的分析提供合适的建议。
(二)、测序数据的降噪和分类
主要包括:
1、Demultiplexing sequences 根据已知的引物barcode序列将混样测序结果分成不同的样品。
2、DAD2 denoising and quality filtering 去除末端质量较差的数据并对每个样品进行降噪,得到每个样品的特征序列用于进行菌群分类。
3、OTU classifier analysis 基于朴素贝叶斯分类器对各个样品的菌群进行分类。
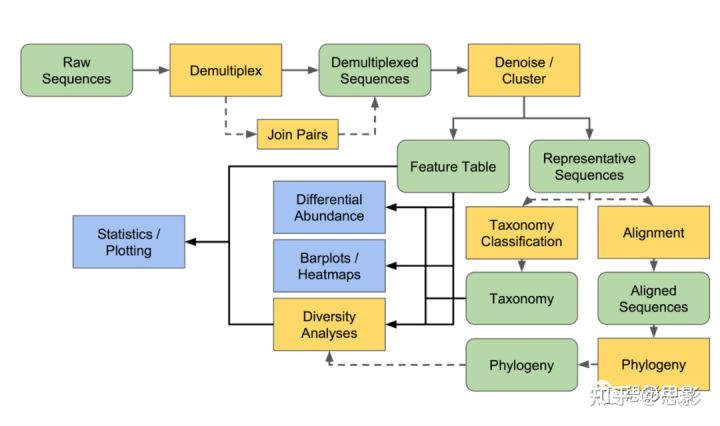
二、相关指标计算及统计
1、差异OTU数量及变化规律:火山图

横轴为相对丰度变化的差异倍数,纵轴为相对丰度取Log2对数值;红色点为显著上调的OTU,绿色为显著下调的OTU,灰色为不显著变化的OTU;除上下调外,大小代表相对丰度的对数值,点的形状代表物种的门分类信息。
2、样本菌群分布和分类进化树
运用系统发育树方法对各个样本中菌群多样性进行分析,得到样品菌群分类进化树。

3、Alpha多样性指标:指通过单样本的多样性分析反映样品内的微生物群落的丰富度和多样性。
主要包括:
测序深度指数(Observed spieces和Good’s coverage)
菌群丰度(Chao1和ACE)
菌群多样性指数(shannon指数和simpson指数) Faith’s Phylogenetic Diversity Pielou’s Evenness
4、Beta多样性指标
主要包括:
Jaccard distance(群落差异的定性度量,即只考虑种类,不考虑丰度);
Bray-Curtis distance(群落差异的定量度量);
unweighted UniFrac distance(非加权UniFrac距离(包含特征之间的系统发育关系的群落差异定性度量));
weighted UniFrac distance(加权UniFrac距离(包含特征之间的系统发育关系的群落差异定量度量));
5、组间差异分析(OUT(97%)或者ASV(99%))
主要包括:
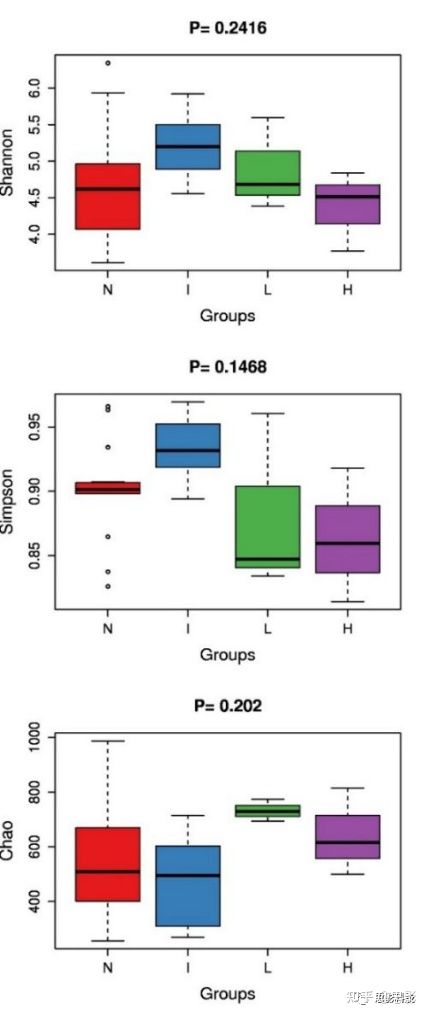
5.1、Alpha多样性样本间差异分析:
5.2、Beta多样性5.2.1 组间Bray-Curtis相异度:

5.2.2、NMDS非度量多维尺度分析:

5.2.3、PCA:基于欧式距离;

5.2.4、PCoA分析:即主坐标分析,也是一种非约束性的数据降维分析方法,可用来研究样本群落组成的相似性或差异性,与PCA分析类似;主要区别在于,PCA基于欧氏距离,PCoA基于除欧氏距离以外的其它距离,通过降维找出影响样本群落组成差异的潜在主成分。

5.2.5、菌群与环境因子相关性分析:Mantel test分析,例如:人体微生物区与某疾病程度的相关性;不同药物组合处理疾病后,微生物组成结构与病情改善之间的相关性。

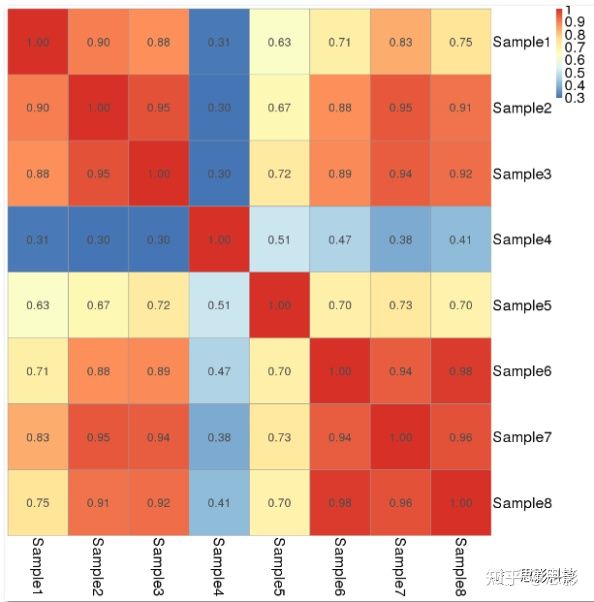
5.2.6、样本间相关性分析
5.2.7、组间物种丰度热图:

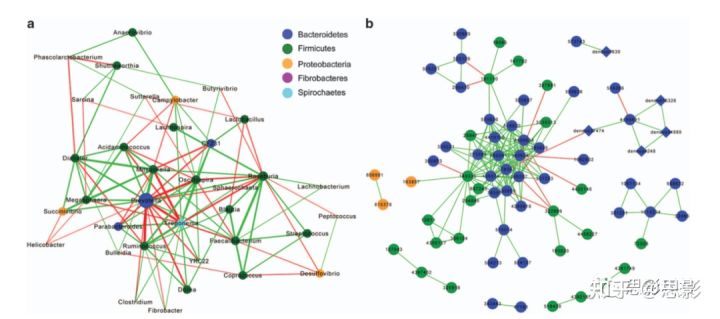
5.2.8、network网络分析:结果输出:
5.2.9、LDA(linear discriminant analysis)差异分析(Lefse分析)
Lefse分析:组间菌群差异,找出各组间差异的微生物种类。其中显著差异的logarithmic LDA score设为2。
目的:寻找与生物学意义关联的biomarker
分三步:1、组间属性丰度比较;2、样本间属性丰度比较;3、分组并定义相关程度

5.2.10、Meta-stats分析:组间群落显著性差异分析,根据得到的群落丰度数据,运用严格的统计学方法可以检测两组微生物群落中表现出丰度差异的分类,进行稀有频率数据的多重假设检验和假发现率(FDR)分析可以评估观察到的差异的显著性。分析可选择门、纲、目、科及属等不同分类学水平。
应用范围:
转录组、16S、宏基因组等;只能两组样本之间比较(非参数T检验、卡方检验);分析可以没有生物学重复(卡方检验代替分参数T检验)
作用:差异物种分析;差异功能分析(数据不一定符合正态分布,参数检验不适合)
5.2.11、STAMP分析:可以实现不同平台下兼容性分析数据。
6、定制化分析
思影可根据客户需求,提供定制化服务,如脑肠轴分析:与脑影像数据(结构或功能指标)、脑电、行为、量表数据的联合分析。

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








