本文转自:http://blog.csdn.net/qq_27469517/article/details/53482563
整个第四章都是数据预处理。
4.1是数据清洗。就是处理无关数据,缺失或者异常数据等等。
具体看书,就不赘述了,还是上代码实践。
书上给的代码是有问题的!
-
- import pandas as pd
- from scipy.interpolate import lagrange
-
- inputfile = 'data/catering_sale.xls'
- outputfile = 'tmp/sales.xls'
-
- data = pd.read_excel(inputfile)
- data[u'销量'][(data[u'销量'] < 400) | (data[u'销量'] > 5000)] = None
-
-
-
- def ployinterp_column(s, n, k=5):
- y = s[list(range(n-k, n)) + list(range(n+1, n+1+k))]
- y = y[y.notnull()]
- return lagrange(y.index, list(y))(n)
-
-
- for i in data.columns:
- for j in range(len(data)):
- if (data[i].isnull())[j]:
- data[i][j] = ployinterp_column(data[i], j)
-
- data.to_excel(outputfile)
究其原因,应该是
- data[u'销量'][(data[u'销量'] < 400) | (data[u'销量'] > 5000)] = None
这句话有问题。
改正方法主要是 .loc 函数进行修改。
.loc 函数主要是选定指定列操作,参见 http://blog.csdn.net/chixujohnny/article/details/51095817
参考下面这个链接
http://blog.csdn.net/o1101574955/article/details/51627401
给出了修改版:
-
-
- import pandas as pd
- from scipy.interpolate import lagrange
-
- inputfile = 'data/catering_sale.xls'
- outputfile = 'tmp/sales.xls'
-
- data = pd.read_excel(inputfile)
-
- row_indexs = (data[u'销量'] < 400) | (data[u'销量'] > 5000)
- data.loc[row_indexs,u'销量'] = None
-
-
-
- def ployinterp_column(s, n, k=5):
- y = s[list(range(n-k, n)) + list(range(n+1, n+1+k))]
- y = y[y.notnull()]
- return lagrange(y.index, list(y))(n)
-
-
- for i in data.columns:
- for j in range(len(data)):
- if (data[i].isnull())[j]:
-
- data.loc[j,i] = ployinterp_column(data[i], j)
- data.to_excel(outputfile)
这时候我在 http://blog.csdn.net/aq_cainiao_aq/article/details/53257136 也看见一篇,我没试过,可以参考。
def那段代码应该要结合拉格朗日插值法具体看,现在简单分析一下:
s为列向量,n为被插值的位置,k为取前后的数据个数,默认为5。
返回的拉格朗日函数有两个值,y.index应该是插值位置,list(y) 就是结果吧。
可惜没找到关于 lagrange 函数的说明,只能猜。
现在的问题是,日期全变成了 ######,想想怎么修改呢?
其实日期是没问题的,但是后面多了时分秒,太长了就变成了######。我不想要这个东西。
去百度搜索了很久,http://blog.csdn.net/dm_vincent/article/details/48696857 参考了一下这个,但是感觉没看懂。
不过呢,插值算是处理好了。
===================================================================================================
4.3讲的是数据变换
主要是数据的规范化处理,把数据转换成“适当的”形式。
代码就是狗屎,我自己补充了一下,给的代码不仅没有 print ,还tm少了一个 import,真的服了
-
-
- import pandas as pd
- import numpy as np
-
- datafile = 'data/normalization_data.xls'
- data = pd.read_excel(datafile, header = None)
-
- print (data - data.min())/(data.max() - data.min())
- print (data - data.mean())/data.std()
- print data/10**np.ceil(np.log10(data.abs().max()))

就是如此。
==================================================================================================
4.3.3讲的是连续属性离散化。 但是这代码都是什么鬼???
-
-
- import pandas as pd
-
- datafile = 'data/discretization_data.xls'
- data = pd.read_excel(datafile)
- data = data[u'肝气郁结证型系数'].copy()
- k = 4
-
- d1 = pd.cut(data, k, labels = range(k))
-
-
-
- w = [1.0*i/k for i in range(k+1)]
- w = data.describe(percentiles = w)[4:4+k+1]
- w[0] = w[0]*(1-1e-10)
- d2 = pd.cut(data, w, labels = range(k))
-
- from sklearn.cluster import KMeans
- kmodel = KMeans(n_clusters = k, n_jobs = 4)
- kmodel.fit(data.reshape((len(data), 1)))
- c = pd.DataFrame(kmodel.cluster_centers_).sort(0)
- w = pd.rolling_mean(c, 2).iloc[1:]
- w = [0] + list(w[0]) + [data.max()]
- d3 = pd.cut(data, w, labels = range(k))
-
- def cluster_plot(d, k):
- import matplotlib.pyplot as plt
- plt.rcParams['font.sans-serif'] = ['SimHei']
- plt.rcParams['axes.unicode_minus'] = False
-
- plt.figure(figsize = (8, 3))
- for j in range(0, k):
- plt.plot(data[d==j], [j for i in d[d==j]], 'o')
-
- plt.ylim(-0.5, k-0.5)
- return plt
-
- cluster_plot(d1, k).show()
-
- cluster_plot(d2, k).show()
- cluster_plot(d3, k).show()
运行无限报错。回头再来试试。
以下内容转自http://blog.csdn.net/xuyaoqiaoyaoge/article/details/52678307
2.连续属性离散化
首先是确定分成几个区间,每个区间的范围多大,然后是将每个区间用符号或者整数值来表示。
1.等宽法
相同宽度的区间
2.等频法
将相同数量的记录放入每个区间
3.基于聚类的方法
先聚类,用K-Means,然后对簇进行处理,每个簇一个标记。
总之,区间的数量自己决定。
import pandas as pd
datafile = 'E:/PythonMaterial/chapter4/demo/data/discretization_data.xls'
data = pd.read_excel(datafile)
data = data[u'肝气郁结证型系数'].copy()
k = 4
d1 = pd.cut(data, k, labels = range(k))
print pd.value_counts(d1)
d2=pd.qcut(data,4,labels = range(k))
print pd.value_counts(d2)
w=[0,0.1,0.2,0.3,0.4,0.5,0.6,0.7,0.8,0.9,1]
d3 = pd.qcut(data, w, labels = range(10))
print pd.value_counts(d3)
w=[0,0.1,0.5,0.9,1]
d4 = pd.qcut(data, w, labels = range(4))#labels是可以替换的,如:labels=['hao','zhong','yiban','huai']
print pd.value_counts(d4)
from sklearn.cluster import KMeans
kmodel = KMeans(n_clusters = k, n_jobs = 1)
kmodel.fit(data.reshape((len(data), 1)))
c = pd.DataFrame(kmodel.cluster_centers_).sort(0)
w = pd.rolling_mean(c, 2).iloc[1:]
w = [0] + list(w[0]) + [data.max()]
d3 = pd.cut(data, w, labels = range(k))
def cluster_plot(d, k):
import matplotlib.pyplot as plt
plt.rcParams['font.sans-serif'] = ['SimHei']
plt.rcParams['axes.unicode_minus'] = False
plt.figure(figsize = (8, 3))
for j in range(0, k):
plt.plot(data[d==j], [j for i in d[d==j]], 'o')
plt.ylim(-0.5, k-0.5)
return plt
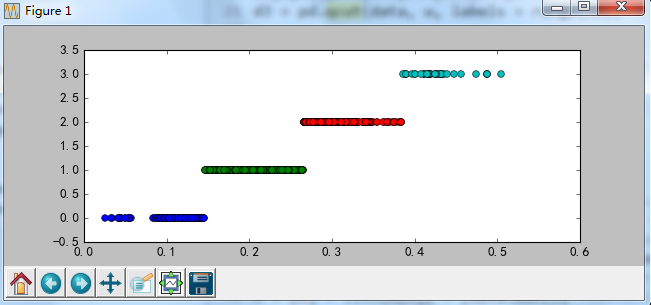
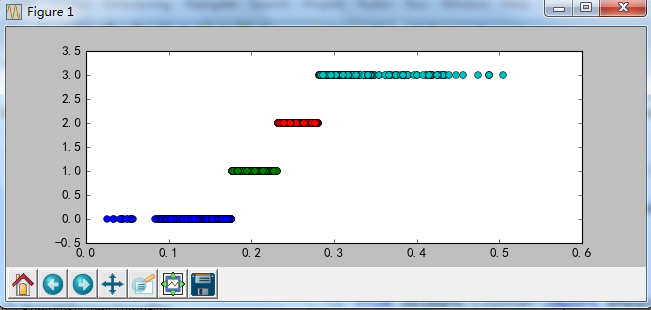
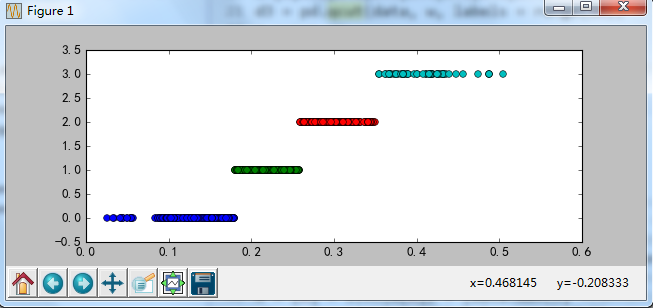
cluster_plot(d1, k).show()
cluster_plot(d2, k).show()
cluster_plot(d3, k).show()
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 27
- 28
- 29
- 30
- 31
- 32
- 33
- 34
- 35
- 36
- 37
- 38
- 39
- 40
- 41
- 42
- 43
- 44
- 45
- 46
- 47
- 48
- 49
- 50
- 51
- 52
- 53
- 54
- 55
1 508
2 275
0 112
3 35
Name: 肝气郁结证型系数, dtype: int64
1 234
3 233
0 233
2 230
Name: 肝气郁结证型系数, dtype: int64
6 97
0 97
4 95
2 94
8 93
9 92
1 92
5 91
7 90
3 89
Name: 肝气郁结证型系数, dtype: int64
2 371
1 370
0 97
3 92
Name: 肝气郁结证型系数, dtype: int64
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26
- 1
- 2
- 3
- 4
- 5
- 6
- 7
- 8
- 9
- 10
- 11
- 12
- 13
- 14
- 15
- 16
- 17
- 18
- 19
- 20
- 21
- 22
- 23
- 24
- 25
- 26


























 1900
1900

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








