目录
自上次实验安装完vscode和anaconda并配置好环境后,我们就可以开始进行机器学习的实验了。
今天我们要开始第一次尝试写机器学习的算法,本次实验选取较为简单的k-近邻算法作为入门的机器学习算法。
1. 安装实验所需要的包
开始实验前先安装实验所需的包
我们需要numpy包,其支持大量的维度数组与矩阵运算,此外也针对数组运算提供大量的数学函数库。
因为我们使用的是anaconda的虚拟环境,因此安装python包需要先进入conda环境。
conda activate 虚拟环境名报错的话去掉conda
安装numpy包
pip install numpy如果下载很慢的话可以切换下载源
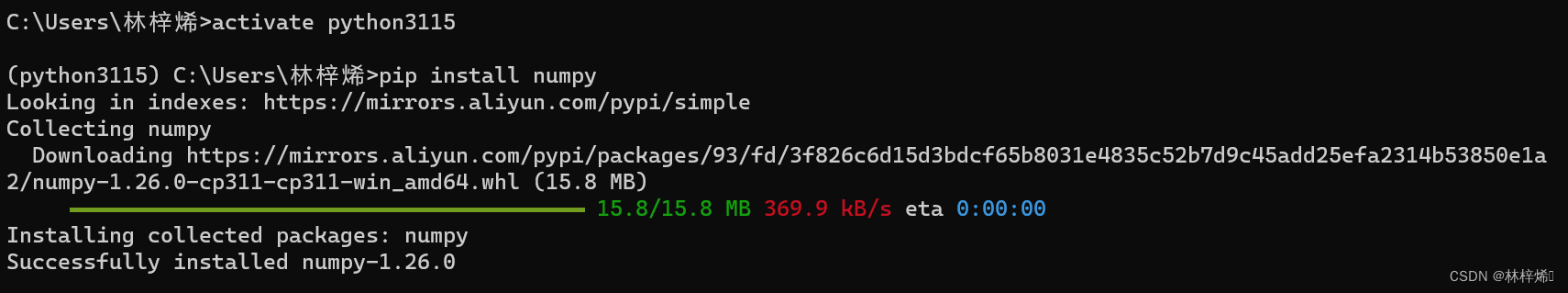
安装完成后在vscode中导入包测试是否会报错,没有报错则说明安装成功
2. 简易k-近邻算法
我们先写一个简易的k-近邻算法,不考虑算法的准确率,只作为入门的上手训练。
这里我使用了书上简单的例子,预测电影的类型。
数据集如下:
不同列以\t分隔
California 3 104 爱情片
He's Not Really into Dudes 2 100 爱情片
Beautiful Woman 1 81 爱情片
Kevin Longblade 101 10 动作片
Robo Slayer 3000 99 5 动作片
amped II 98 2 动作片
首先给出算法的伪代码
(1)读取文件中的特征和对应的标签数据到数据集中。
(2)计算当前点到数据集中其他所有点的距离。
(3)选取k个与当前点距离最小的点。
(4)计算k个点中各类别出现的频率。
(5)返回出现频率最高的类别,该类别即为预测的分类。
代码实现如下:
# _*_ coding:utf-8 _*_
import numpy as np
import matplotlib
# 读取文件,把文件中的数据存入矩阵中
# param: fileName: 文件名
def file_matrix(fileName, feature_num):
# 打开数据文件读取数据
f = open(fileName, 'r', encoding='utf-8')
lines = f.readlines()
# 创建n行2列的以0填充的二维数组存放数据,作为特征矩阵(n为数据的行数)
data_matrix = np.zeros((len(lines), feature_num))
# 数据标签
labels = []
index = 0
# 遍历文件中每行的数据
for line in lines:
# data_line = []
# 将读取的数据进行切割(以'\t'为分隔符),第一列内容不需要,直接切片除去数组中第一个元素,
# 切片前需去除换行符(使用strip())
line_split = line.strip().split('\t')[1 :]
# 将分割完的数据中的前两列存放入data_set中,作为数据集
data_matrix[index, :] = line_split[: -1] # a[i, :]为第i行的所有元素,a[: -1]为取数组的除最后一个外的所有元素
# 将最后一列的内容也就是电影标签保存进标签数组
labels.append(line_split[-1])
index += 1
f.close()
return data_matrix, labels
def classify(inX, data_matrix, labels, k):
# a = np.array([18., 90.])
# 计算距离,这里使用欧式距离公式,使用特征矩阵进行计算
# 首先将待求解数据进行扩展,使其形状与数据集的特征矩阵相同
# tile()函数将数据数组横向复制1次,纵向复制n次(n为数据集行数)
# 然后将待求解数据矩阵与数据集相减后再乘方
sq_dis = (np.tile(inX, (len(data_matrix), 1)) - data_matrix) ** 2
# 将得到的矩阵的每一行进行求和再开方即可得到待求数据到其他所有点的距离
# sum(axis=1)为对矩阵在1轴上(列)求和,即对每一行求和,axis=哪个轴,哪个轴就消失
dis_mat = sq_dis.sum(axis=1) ** 0.5
# 创建近邻标签字典,key:类型,value:个数
near_type = {}
# 选取k个距离最近的点,统计这些点所属的标签,并累计各个标签的个数(个数按标签存放入字典中)
for i in range(k):
# 注意这里的dis_mat与原本的labels是对应的,所以不能改变dis_mat的顺序,
# 而是使用np.argsort()函数获取其排序后的索引从而访问差距最小的k个样本的标签,其顺序是升序
# 如[2,1,3],它的argsort的索引就是[1,0,2],
# 遍历时数组时将索引 “i” 替换为 “np.argsort(a)[i]” 得到的就是排序过后的数组
if(labels[np.argsort(dis_mat)[i]] not in near_type.keys()): # 不存在这个键就新增
near_type[labels[np.argsort(dis_mat)[i]]] = 1
else: # 存在这个键就将该键的值加1
near_type[labels[np.argsort(dis_mat)[i]]] += 1
return max(near_type, key=near_type.get), near_type
a = np.array([18., 90.])
data_matrix, labels = file_matrix('knn测试数据.txt', 2)
type1, near_type = classify(a, data_matrix, labels, 3)
print('k个点中各类别出现的频率:\n', near_type)
# 输出个数最多的标签即为待求数据预测结果
print('预测结果:\n', type1 )
运行结果:
k为3:
k个点中各类别出现的频率:
{'爱情片': 3}
预测结果:
爱情片
k为5:
k个点中各类别出现的频率:
{'爱情片': 3, '动作片': 2}
预测结果:
爱情片
3. 更换复杂数据集进行预测
现在我们换一个更复杂的数据集进行预测
下面是数据集(仅截取部分数据)
40920 8.326976 0.953952 largeDoses
14488 7.153469 1.673904 smallDoses
26052 1.441871 0.805124 didntLike
75136 13.147394 0.428964 didntLike
38344 1.669788 0.134296 didntLike
72993 10.141740 1.032955 didntLike
35948 6.830792 1.213192 largeDoses
42666 13.276369 0.543880 largeDoses
67497 8.631577 0.749278 didntLike
35483 12.273169 1.508053 largeDoses
50242 3.723498 0.831917 didntLike
63275 8.385879 1.669485 didntLike
5569 4.875435 0.728658 smallDoses
51052 4.680098 0.625224 didntLike
77372 15.299570 0.331351 didntLike
43673 1.889461 0.191283 didntLike
61364 7.516754 1.269164 didntLike
69673 14.239195 0.261333 didntLike
15669 0.000000 1.250185 smallDoses
28488 10.528555 1.304844 largeDoses
6487 3.540265 0.822483 smallDoses
上面的数据集中前三列为样本特征,最后一列为样本标签
根据此数据集将读取数据的函数稍作修改(此数据集第一列是有用数据)
def file_matrix(fileName, feature_num):
# 打开数据文件读取数据
f = open(fileName, 'r', encoding='utf-8')
lines = f.readlines()
# 创建n行2列的以0填充的二维数组存放数据,作为特征矩阵(n为数据的行数)
data_matrix = np.zeros((len(lines), feature_num))
# 数据标签
labels = []
index = 0
# 遍历文件中每行的数据
for line in lines:
# data_line = []
# 将读取的数据进行切割(以'\t'为分隔符)
# 切片前需去除换行符(使用strip())
line_split = line.strip().split('\t')
# 将分割完的数据中的前两列存放入data_set中,作为数据集
data_matrix[index, :] = line_split[: feature_num] # a[i, :]为第i行的所有元素,a[: n]为取数组中的从0到n-1的所有元素
# 将最后一列的内容也就是电影标签保存进标签数组
labels.append(line_split[-1])
index += 1
f.close()
return data_matrix, labels给出点[40000, 1.3, 0.95],k选择10,预测其结果
k个点中各类别出现的频率:
{'largeDoses': 7, 'didntLike': 3}
预测结果:
largeDoses
使用点[40000, 10.3, 10.95],k同上,预测其结果
k个点中各类别出现的频率:
{'largeDoses': 7, 'didntLike': 3}
预测结果:
largeDoses
这里就出现了一个问题,第一个点和第二个点的第二、第三个点差异很大,但预测的结果却相同。导致这个问题的原因是三个特征值的数量级差距较大,第一个特征比第二、第三特征的差距过大,在与其他点进行比较时,第一个特征的差距也就会比第二、第三特征多得多,导致第二、第三特征几乎被“忽略”掉了,因此我们需要对数据集进行处理,使三个特征对结果的影响相同。
这里我们采用的处理方法是将数值进行归一化,将数值转化为[0,1]区间或[-1, 1]区间内的值,因为这里使用的数据集中的数据都为正数,因此将数值归一化到[0,1]区间,使用下面的公式实现:
newValue = (oldValue - min) / (max - min)
根据上述公式给出归一化函数代码:
def autoNorm(data_matrix):
# 计算每列数据的最大最小值和数值的范围
# min(axis=0)是在1轴上(列)的所有值的最小值,得出的结果是各个列的最小值,max同理
# 将得到最大最小值的结果扩展至n行3列(n为数据集样本个数),以便后续的操作
minValues = np.tile(data_matrix.min(axis=0), (len(data_matrix), 1))
maxValues = np.tile(data_matrix.max(axis=0), (len(data_matrix), 1))
value_range = maxValues - minValues
norm_data = (data_matrix - minValues) / value_range
return norm_data归一化后的数据集:
[[0.44832535 0.39805139 0.56233353]
[0.15873259 0.34195467 0.98724416]
[0.28542943 0.06892523 0.47449629]
...
[0.29115949 0.50910294 0.51079493]
[0.52711097 0.43665451 0.4290048 ]
[0.47940793 0.3768091 0.78571804]]
接着使用归一化后的数据从新进行预测,注意不要忘了归一化预测的样本
样本:[40000, 8., 0.9]
预测结果:
[0.44832535 0.39805139 0.56233353] largeDoses [0.0368758]
[0.40775476 0.37047534 0.51225093] largeDoses [0.03748485]
[0.4036462 0.42206758 0.52792823] largeDoses [0.0526834]
[0.36902479 0.36946169 0.54258685] largeDoses [0.07145473]
[0.425931 0.42194812 0.59088471] largeDoses [0.07322149]
[0.45335422 0.43408057 0.58356159] largeDoses [0.07558712]
[0.37070108 0.39140821 0.5687926 ] largeDoses [0.07816664]
[0.35813439 0.41804059 0.50531852] largeDoses [0.09121539]
[0.47698662 0.39455214 0.44119524] largeDoses [0.09809094]
[0.35250293 0.32415569 0.52183921] largeDoses [0.10402663]
k个点中各类别出现的频率: {'largeDoses': 10}
预测结果: largeDoses
样本:[40000, 6., 1.2]
预测结果
[0.39385141 0.32652986 0.71533516] largeDoses [0.06007227]
[0.46214105 0.3248929 0.65570265] largeDoses [0.06862165]
[0.48649655 0.34545836 0.74020412] largeDoses [0.08266456]
[0.46428845 0.3020898 0.62409723] didntLike [0.08874556]
[0.40945296 0.20113996 0.76052919] didntLike [0.10476754]
[0.36053378 0.33809675 0.65879113] largeDoses [0.1051011]
[0.53807807 0.33644474 0.71423563] largeDoses [0.11168814]
[0.51470862 0.32152774 0.63045833] largeDoses [0.11399351]
[0.36871802 0.31502142 0.79791792] didntLike [0.11745695]
[0.36401784 0.38052298 0.71921155] largeDoses [0.12011169]
k个点中各类别出现的频率: {'largeDoses': 7, 'didntLike': 3}
预测结果: largeDoses
4. 测试数据集
至此,我们已经掌握了knn算法的简易实现,但是这样的算法是没有实际用处的,原因在于我们并没有告诉该算法什么时候它是对的,什么时候是错的,也就是我们并不能知道它的错误率,它所得出的结果可靠性很低。
为了测试算法的预测效果,我们需要给它大量已知答案的数据,也就是测试数据,通过大量测试数据的,我们可以得出它预测的错误率,通过不断得调整参数,我们可以将它的错误率降到一个我们可以接受的范围,然后再使用该算法进行预测,所得出的结果可靠性就很高了。
现在我们开始写测试算法
伪代码如下:
(1)从文件中读取数据集
(2)归一化数据
(3)确定测试集数量
(4)遍历测试集:
4.1. 将测试集中抽取的样本数据传入分类器进行预测分类
4.2. 将预测结果和实际结果进行比较,判断正误,并统计错误个数
(5)计算错误率
(6)返回错误率
代码实现:
# 测试数据,从数据集中抽取一定量的数据作为测试集,测试算法的错误率
# params:file_name:文件名,feature_num:样本特征个数
# return:err_rate: 错误率(百分比)
def datingClassTest(file_name, feature_num):
# 测试集占数据集的比例,这个比例一般较小
hoRatio = 0.1
# 读取数据集
data_matrix, labels = file_matrix(file_name, feature_num)
# 归一化数据
norm_data = autoNorm(data_matrix)
# 测试集数量为数据集总数乘以测试集所占比例,因为hoRatio为float型,得出的结果也为float,需要强制类型转换为int
num_test_samples = int(len(data_matrix) * hoRatio)
# 统计预测错误个数,用于计算错误率
err_count = 0.
k = 3
# 进行测试数据,从数据集中抽取测试集,使用测试集运行分类算法,再将预测结果与实际值比较,计算出错误率
for i in range(num_test_samples):
# 使用测试数据运行分类算法,得出预测标签
# 这里的测试数据是单个样本,即数据集中的第i行,训练集是除测试集的所有行
# norm_data[i, :]表示第i行的所有元素,norm_data[num_test_samples : len(norm_data), :]表示[num_test_samples, len(norm_data))区间的所有行
predicted_label, near_type = classify(norm_data[i, :], norm_data[num_test_samples : len(norm_data), :], labels[num_test_samples : len(norm_data)], k)
# 输出预测值与实际值
print('预测标签为: ', predicted_label, ', 实际标签为: ', labels[i])
# 如果预测值与实际值相同,则预测结果正确;否则预测错误,err_count + 1
if(predicted_label != labels[i]):
err_count += 1
err_rate = err_count / num_test_samples * 100
return err_rate预测结果(一下仅为部分结果):
预测标签为: largeDoses , 实际标签为: largeDoses
预测标签为: smallDoses , 实际标签为: smallDoses
预测标签为: didntLike , 实际标签为: didntLike
预测标签为: didntLike , 实际标签为: didntLike
预测标签为: didntLike , 实际标签为: didntLike
预测标签为: didntLike , 实际标签为: didntLike
预测标签为: largeDoses , 实际标签为: largeDoses
预测标签为: largeDoses , 实际标签为: largeDoses
预测标签为: didntLike , 实际标签为: didntLike
预测标签为: largeDoses , 实际标签为: largeDoses
预测标签为: didntLike , 实际标签为: didntLike
预测标签为: didntLike , 实际标签为: didntLike
预测标签为: smallDoses , 实际标签为: smallDoses
预测错误率为: 5.0 %
将k改为4,错误率为
预测错误率为: 4.0 %
在不改变测试集比例的情况下,这个结果已经是最好的了,如果再降低一点测试集的比例,可以得到1.25%的错误率甚至是0%,不过这显然没有什么意义,测试集样本数太少,很难准确的评估模型的性能。
现在我们已经完全掌握了knn算法,接下来让我们来选择一些其他的数据集来运行我们的代码。
5. 更换数据集测试与预测
我们读取红酒分类数据集进行测试。
14.23,1.71,2.43,15.6,127,2.8,3.06,0.28,2.29,5.64,1.04,3.92,1065,0
13.2,1.78,2.14,11.2,100,2.65,2.76,0.26,1.28,4.38,1.05,3.4,1050,0
13.16,2.36,2.67,18.6,101,2.8,3.24,0.3,2.81,5.68,1.03,3.17,1185,0
14.37,1.95,2.5,16.8,113,3.85,3.49,0.24,2.18,7.8,0.86,3.45,1480,0
13.24,2.59,2.87,21,118,2.8,2.69,0.39,1.82,4.32,1.04,2.93,735,0
14.2,1.76,2.45,15.2,112,3.27,3.39,0.34,1.97,6.75,1.05,2.85,1450,0
14.39,1.87,2.45,14.6,96,2.5,2.52,0.3,1.98,5.25,1.02,3.58,1290,0
14.06,2.15,2.61,17.6,121,2.6,2.51,0.31,1.25,5.05,1.06,3.58,1295,0
14.83,1.64,2.17,14,97,2.8,2.98,0.29,1.98,5.2,1.08,2.85,1045,0
13.86,1.35,2.27,16,98,2.98,3.15,0.22,1.85,7.22,1.01,3.55,1045,0
14.1,2.16,2.3,18,105,2.95,3.32,0.22,2.38,5.75,1.25,3.17,1510,0
14.12,1.48,2.32,16.8,95,2.2,2.43,0.26,1.57,5,1.17,2.82,1280,0
13.75,1.73,2.41,16,89,2.6,2.76,0.29,1.81,5.6,1.15,2.9,1320,0
14.75,1.73,2.39,11.4,91,3.1,3.69,0.43,2.81,5.4,1.25,2.73,1150,0
14.38,1.87,2.38,12,102,3.3,3.64,0.29,2.96,7.5,1.2,3,1547,0
13.63,1.81,2.7,17.2,112,2.85,2.91,0.3,1.46,7.3,1.28,2.88,1310,0
其包含13个特征:
Alcohol Malic Ash Alcalinity of ash Magnesium Total phenols Flavanoids
Nonflavanoid phenols Proanthocyanins Color intensity Hue ofdiluted wines
Proline
标签为红酒的种类
首先先测试
# _*_ coding:utf-8 _*_
import numpy as np
# 读取文件,把文件中的数据存入矩阵中
# params: fileName: 文件名,feature_num:样本特征个数
# return:data_matrix:特征矩阵,labels样本标签
def file_matrix(fileName, feature_num, start_line, separator):
# 打开数据文件读取数据
f = open(fileName, 'r', encoding='utf-8')
lines = f.readlines()
# print(lines)
# 创建n行2列的以0填充的二维数组存放数据,作为特征矩阵(n为数据的行数)
data_matrix = np.zeros((len(lines), feature_num))
# 数据标签
labels = []
index = 0
# 遍历文件中每行的数据
for line in lines[start_line:]:
# data_line = []
# 将读取的数据进行切割(以'\t'为分隔符),第一列内容不需要,直接切片除去数组中第一个元素,
# 切片前需去除换行符(使用strip())
line_split = line.strip().split(separator)
# 将分割完的数据中的前两列存放入data_set中,作为数据集
data_matrix[index, :] = line_split[: feature_num] # a[i, :]为第i行的所有元素,a[: n]为取数组中的从0到feature_num-1的所有元素
# 将最后一列的内容也就是电影标签保存进标签数组
labels.append(line_split[-1])
index += 1
f.close()
print(data_matrix)
print(len(labels))
return data_matrix, labels
# 分类数据
# params:inX:预测样本,data_matrix:特征矩阵,labels样本标签,k:近邻点个数
# return:预测标签,near_type:k个点中各类别的频率
def classify(inX, data_matrix, labels, k):
# a = np.array([18., 90.])
# 计算距离,这里使用欧式距离公式,使用特征矩阵进行计算
# 首先将待求解数据进行扩展,使其形状与数据集的特征矩阵相同
# tile()函数将数据数组横向复制1次,纵向复制n次(n为数据集行数)
# 然后将待求解数据矩阵与数据集相减后再乘方
sq_dis = (np.tile(inX, (len(data_matrix), 1)) - data_matrix) ** 2
# print('sq_dis:\n', sq_dis ** 0.5)
# 将得到的矩阵的每一行进行求和再开方即可得到待求数据到其他所有点的距离
# sum(axis=1)为对矩阵在1轴上(列)求和,即对每一行求和,axis=哪个轴,哪个轴就消失
# dis_mat = sq_dis.sum(axis=1) ** 0.5
dis_mat = np.sum(sq_dis, axis=1) ** 0.5
# 创建近邻标签字典,key:类型,value:个数
near_type = {}
# 选取k个距离最近的点,统计这些点所属的标签,并累计各个标签的个数(个数按标签存放入字典中)
for i in range(k):
# 注意这里的dis_mat与原本的labels是对应的,所以不能改变dis_mat的顺序,
# 而是使用np.argsort()函数获取其排序后的索引从而访问差距最小的k个样本的标签,其顺序是升序
# 如[2,1,3],它的argsort的索引就是[1,0,2],
# 遍历时数组时将索引 “i” 替换为 “np.argsort(a)[i]” 得到的就是排序过后的数组
# if(labels[np.argsort(dis_mat)[i]] not in near_type.keys()): # 不存在这个键就新增
# near_type[labels[np.argsort(dis_mat)[i]]] = 1
# else: # 存在这个键就将该键的值加1
# near_type[labels[np.argsort(dis_mat)[i]]] += 1
if(labels[np.argsort(dis_mat)[i]] not in near_type.keys()): # 不存在这个键就新增
near_type[labels[np.argsort(dis_mat)[i]]] = 1
else: # 存在这个键就将该键的值加1
near_type[labels[np.argsort(dis_mat)[i]]] += 1
# print(data_matrix[np.argsort(dis_mat)[i]], labels[np.argsort(dis_mat)[i]], dis_mat[[np.argsort(dis_mat)[i]]])
# print()
return max(near_type, key=near_type.get), near_type
# 归一化数据
# params:data_matrix:特征矩阵
# return:norm_data:归一化后的数据集
def autoNorm(data_matrix):
# 计算每列数据的最大最小值和数值的范围
# min(axis=0)是在1轴上(列)的所有值的最小值,得出的结果是各个列的最小值,max同理
# 将得到最大最小值的结果扩展至n行3列(n为数据集样本个数),以便后续的操作
minValues = np.tile(data_matrix.min(axis=0), (len(data_matrix), 1))
maxValues = np.tile(data_matrix.max(axis=0), (len(data_matrix), 1))
value_range = maxValues - minValues
norm_data = (data_matrix - minValues) / value_range
return norm_data
def autoNormInx(inX, data_matrix):
# 计算每列数据的最大最小值和数值的范围
# min(axis=0)是在1轴上(列)的所有值的最小值,得出的结果是各个列的最小值,max同理
# 将得到最大最小值的结果扩展至n行3列(n为数据集样本个数),以便后续的操作
minValues = data_matrix.min(axis=0)
maxValues = data_matrix.max(axis=0)
value_range = maxValues - minValues
norm_data = (inX - minValues) / value_range
return norm_data
# 测试数据,从数据集中抽取一定量的数据作为测试集,测试算法的错误率
# params:file_name:文件名,feature_num:样本特征个数
# return:err_rate: 错误率(百分比)
def datingClassTest(file_name, feature_num, start_line, separator):
# 测试集占数据集的比例,这个比例一般较小
hoRatio = 0.1
# 读取数据集
data_matrix, labels = file_matrix(file_name, feature_num, start_line, separator)
# 归一化数据
norm_data = autoNorm(data_matrix)
print(norm_data)
# 测试集数量为数据集总数乘以测试集所占比例,因为hoRatio为float型,得出的结果也为float,需要强制类型转换为int
num_test_samples = int(len(data_matrix) * hoRatio)
# 统计预测错误个数,用于计算错误率
err_count = 0.
k = 3
# 进行测试数据,从数据集中抽取测试集,使用测试集运行分类算法,再将预测结果与实际值比较,计算出错误率
for i in range(num_test_samples):
# 使用测试数据运行分类算法,得出预测标签
# 这里的测试数据是单个样本,即数据集中的第i行,训练集是除测试集的所有行
# norm_data[i, :]表示第i行的所有元素,norm_data[num_test_samples : len(norm_data), :]表示[num_test_samples, len(norm_data))区间的所有行
predicted_label, near_type = classify(norm_data[i, :], norm_data[num_test_samples : len(norm_data), :], labels[num_test_samples : len(norm_data)], k)
# 输出预测值与实际值
print('预测标签为: ', predicted_label, ', 实际标签为: ', labels[i])
# 如果预测值与实际值相同,则预测结果正确;否则预测错误,err_count + 1
if(predicted_label != labels[i]):
err_count += 1
err_rate = err_count / num_test_samples * 100
return err_rate
err_rate = datingClassTest('wine_data.csv', 13, 0, ',')
print('预测错误率为:', err_rate, '%')
输出结果
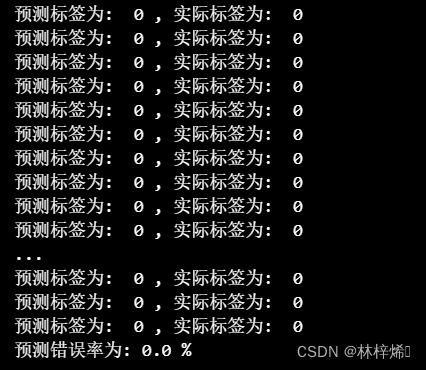
可以看出测试集测试的预测错误率居然达到了0.0%,0%的错误率基本是不可能的,那么就可能是哪里出了问题,我试着调高了一下测试集的比例,当我将比例调高到0.21的时候,发现它能得到一个比较正常的错误率2.7%
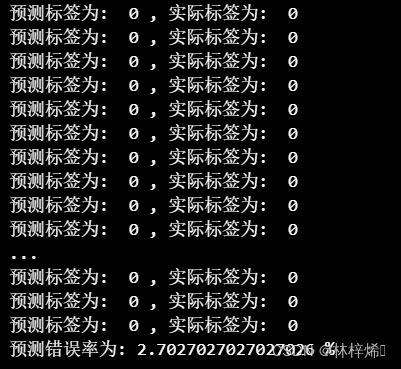
那么先前预测错误率达到0%则有可能是因为测试集太少,不能涵盖足够的样本,其得到的错误率可靠性也就很低。
我查看了一下数据集,发现数据集标签是有序的,也就是数据集前n行都是标签为0的样本,接下来是1,它的样本顺序是没有打乱的,在我们进行测试的时候,选择较小的测试集比例,则其基本只能测试一个种类的样本的错误率,而不能涵盖全部种类,调高测试集比例自然能够解决这个问题,不过这样仍然有点缺陷,我们可以选择将样本的顺序打乱,使其各种种类分布不再有序。
在file_matrix函数中lines = f.readlines()语句的后面加上random.shuffle(lines)可以打乱其顺序。
打乱后我们再测试一次
这里测试集比例设为0.1

可以看出错误率正常了
接着我们自己输入一组数据来预测其类型
写一个接收输入的函数
def classifyPerson():
alcohol = float(input("please input Alcohol: "))
malic_acid = float(input("please input Malic acid: "))
ash = float(input("please input Ash: "))
alcalinity_of_ash = float(input("please input Alcalinity of ash: "))
magnesium = float(input("please input Magnesium: "))
total_phenols = float(input("please input Total phenols: "))
flavanoids = float(input("please input Flavanoids: "))
nonflavanoid_phenols = float(input("please input Nonflavanoid phenols: "))
proanthocyanins = float(input("please input proanthocyanins: "))
color_intensity = float(input("please input color_intensity: "))
hue = float(input("please input hue: "))
ofdiluted_wines = float(input("please input ofdiluted_wines: "))
proline = float(input("please input proline: "))
inX = np.array([alcohol, malic_acid, ash, alcalinity_of_ash, magnesium, total_phenols,
flavanoids, nonflavanoid_phenols, proanthocyanins, color_intensity,
hue, ofdiluted_wines, proline])
data_matrix, labels = file_matrix('wine_data.csv', 13, 0, ',')
print(inX)
inX = autoNormInx(inX, data_matrix)
print('the wine kind probably is: ', classify(inX, data_matrix, labels, 3)[0])数据:[ 12. 1.81 2.3 18.4 80.7 1.58 1.45 0.51 1.63 2.36 1.06 2.24 478. ]
预测结果:
the wine kind probably is: 1
与其接近的数据为
![]()






















 1032
1032











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








