基础操作
#获取当前脚本所在位置
getwd()
#更改脚本位置
setwd(dir = "C:/Users/LG/Desktop/R") #注意斜杠是正斜线
#查看当前目录下存在的文件
dir()x<-5 #赋值给局部变量
y<<-3 #赋值给全局变量
x <- sum(1,2,3,4,5)
y <- 5
rm(x,y) #删除对象x和y
history(25) #显示前25条操作记录data() #查看内置的数据集
?plot #寻找关于plot的帮助ctrl + L 清空屏幕
#导入tidyr包(一次性操作)
install.packages('tidyr')
#加载包(每次都要载入)
library(tidyr)
#获取datasets包中的所有示例数据集、获取所有包的数据集、获取特定包的数据集
data(births2006.smpl)数据
R中的数据类型:数值型,字符串型,逻辑型,日期型
1.对象类型:向量、矩阵、数组、列表、数据框
#查看dataframe中的每个字段的数据类型
str(df)
向量vectors,存储数值型、字符串型、逻辑型数据的一维数组
向量内必须都是同个类型的
#定义向量
x <- c(1,2,3,4,5)
y <- c("one","two","three")
z <- c(TRUE,T,F)
z <- c(1:100) #生成1到100的等差为1的数列
seq (from=1,to=100,by=2) #生成1到100的等差值为2的数列
seq (from=1,to=100,length.out=10) #生成1到100的等差的10个值的数列
rep(2,5) #生成拥有5个2的数列a <- c(1,2,"one") #输出"1","2","one"
length(a) #统计向量a里的元素个数(每个字段里几行)
a[2] #访问向量a中的第2个元素(从1开始而不是0)
a[-2] #访问向量a中除了第2个元素外的其它所有元素
a[c(1,2)] #输出a中对应位置的元素列表lists它可以在其中包含许多不同类型的元素,如向量,函数甚至其中的另一个列表。
矩阵matrix是二维的
矩阵内也必须都是同个类型的
#创建矩阵
x <- 1:20
m <- matrix(x,4,5,byrow=T) #4行5列,T表示按行排列,F按列排列
#矩阵索引命名
rnames <- c("R1","R2","R3","R4")
cnames <- c("C1","C2","C3","C4","C5")
dimnames(m) <- list(rnames,cnames)数组arrays是三维的
#创建数组
x <- 1:20
dim(x) <- c(2,2,5)#矩阵与数组的索引
m[2,3] #第2行第3列的数据
m[2,c(2,3,4)] #第2行的第2、3、4列的数据
m[2,] #访问第2行所有
m[,2] #访问第2列所有
m["R1","C2"] #通过索引查找数据帧Data Frames
#创建dataframe
BMI <- data.frame(
gender = c("Male", "Male","Female"),
height = c(152, 171.5, 165),
weight = c(81,93, 78),
Age = c(42,38,26)
)
print(BMI)
# gender height weight Age
#1 Male 152.0 81 42
#2 Male 171.5 93 38
#3 Female 165.0 78 26 2.对象类型2:因子
在R中,名义型变量和有序型变量称为因子
例如:颜色就是factor,蓝绿青黄就是level
#创建因子
f <- factor(c("red","white","blue","red"))
#[1] red white blue red
#Levels: blue red white
table()函数:

3.缺失值
R中缺失值用NA表示
#创建带有缺失值的向量
a <- c(NA,1:20)
#跳过缺失值计算总值或均值(否则计算出来的也是NA),注意均值的分母也改变了
sum(a,na.rm=TRUE)
mean(a,na.rm=TRUE)
#测试每个值是否为NA,输出TURE或FALSE
is.na(a)
处理缺失值:
install.packages("VIM")
library(VIM)
#去掉NA值(必须重新赋值,只运行这个函数不会改变原数据集)
a <- na.omit(a)
#去掉带有NA的整行,在sleep(系统自带数据集)中
na.omit(sleep)NA是存在的值,但是缺失
NaN是不存在的
Inf是无穷大或无穷小
4.字符串
#获取字符个数(空格也算)
nchar("hello world")
month.name
#[1] "January" "February" "March" "April" "May" "June" "July"
#[8] "August" "September" "October" "November" "December"
#nchar返回了向量中每个元素的字符的个数,结果还是向量
nchar(month.name)
#[1] 7 8 5 5 3 4 4 6 9 7 8 8
#注意区分length和nchar
length(month.name)
#[1] 12paste("we","love","code")
#[1] "we love code"
paste("we","love","code",sep="-")
#[1] "we-love-code"读写文件
读文件:1.该文件在脚本所在目录:直接读文件名;2.不在当前目录下:需要绝对路径
#读入txt文件的数据
x <- read.table("pp.txt")
x <- read.table("c:/Users/pp.txt",sep=',',header=TURE,skip=5) #第1行是表头,跳过前5行
head(x,n=10) #显示头10行
tail(x) #显示尾几行,默认6
#读入csv文件的数据
y <- read.csv("pp.csv")#读取xlsx文件
install.packages('readxl')
library(readxl)
x <- read_excel("PSI.xlsx")数据处理
1.数据转换
#查询是否是dataframe格式
is.data.frame(state.x77)
#把matrix强制转换为dataframe
df <- as.data.frame(state.x77)#转化字符串、逻辑型等为数值型
df$age <- as.numeric(df$age)2.数据筛选
#数据筛选
x <- read_excel("PSI.xlsx")
x1 <- x[which(x$continent==7,)] #筛选出continent这个字段中为7的数据
x2 <- x[ which(x$ID>50 & x$ID<100,) ] #筛选出ID字段中大于50且小于100的数据3.数据抽取
#从向量中抽取
x <- 1:100
sample(x,30) #在x内随机取样30个元素
sample(x,60,replace=T) #有放回的抽样
#从数据框中抽取
y <- read_excel("PSI.xlsx")
y[ sample(y$ID,30,replace=F), ]4.删除数据
y <- read_excel("PSI.xlsx")
y[ , -1:-5 ] #删除第1到第5列
y$ID <- NULL #删除ID列5.合并dataframe
cbind(x,y) #两个数据框暴力一左一右合并(要求有相同的行)
rbind(x,y) #上下合并(要求相同列)6.重复值处理
duplicated(df) #判断向量或数据框中哪些是重复值
df[ duplicated(df), ] #取出重复部分
df[ !duplicated(df), ] #取出非重复部分
unique(df) #把重复去除7.转置
fd <- t(df) #实现转置
letters
#[1] "a" "b" "c" "d" "e" "f" "g" "h" "i" "j" "k" "l" "m" "n" "o" "p" "q" "r" "s" "t" "u" #"v" "w" "x" "y" "z"
rev(letters)
#[1] "z" "y" "x" "w" "v" "u" "t" "s" "r" "q" "p" "o" "n" "m" "l" "k" "j" "i" "h" "g" "f" #"e" "d" "c" "b" "a"8.对行列数据进行处理
transform(df,height=height*100) #直接修改某一列使其所有数据*100
transform(df,h2=height*100) #多生成了一列#sort排序(只能用于向量)
sort(x)
#对数据框进行排序方法1
df[ order(df$ID), ] #按这一列进行升序排序
df[ order(-df$ID), ] #降序排序
df[ order(df$ID,df$height), ] #ID相同则按height排rs <- rowSums(WorldPhones) #对该dataframe每行进行加总#FUN可选:sum,mean,var,log.....
#margin=1对象为每行,2对象为每列,c(1,2)为对所有行和列
apply(WorldPhones,MARGIN=1,FUN=mean)9.数据中心化和标准化
消除因为数据不同的计量单位或者刻度使数据不能直接的比较的问题
中心化:各项数据减去数据集的均值
标准化:中心化后再除以数据集的标准差
scale(df,center=T,scale=F) #只做中心化处理
scale(df,center=T,scale=T) #做标准化处理预处理
merge(df1,df2,by="ID") #根据"ID"字段对应值来合并
length(ID) #查看这个字段有几行(几个元素)install.packages('modeest')
library(modeest)
#most frequent value
mfv(join_df_full$SO2)2.tidyr
tidy data:
1.Each variable forms a column
2.Each observation forms a row
3.Each type of observational unit forms a table
install.packages(c("tidyr","dplyr"))
library(tidyr)df <- data.frame(x=c(NA,"a.b","a.d","b.c"))
# x
#1 <NA>
#2 a.b
#3 a.d
#4 b.c
df1 <- separate(df,col=x,into=c("A","B"),sep=,)
# A B
#1 <NA> <NA>
#2 a b
#3 a d
#4 b c
unite(df1,col="new",A,B,sep="-")
# new
#1 NA-NA
#2 a-b
#3 a-d
#4 b-cpivot_longer()
让输出的数据框更“长”
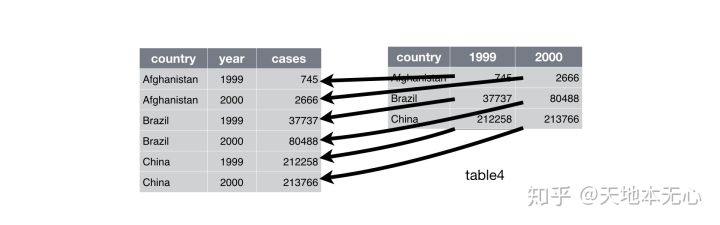
# Sepal.Length Sepal.Width Petal.Length Petal.Width Species
#1 5.1 3.5 1.4 0.2 setosa
#51 7.0 3.2 4.7 1.4 versicolor
#101 6.3 3.3 6.0 2.5 virginica
pivot_longer(data = mini_iris,
cols = c(Sepal.Length, Sepal.Width, Petal.Length, Petal.Width),
names_to = "flower_attr",
values_to = "attr_value")
# A tibble: 12 x 3
# Species flower_attr value
# <fct> <chr> <dbl>
# 1 setosa Sepal.Length 5.1
# 2 setosa Petal.Length 1.4
# 3 setosa Sepal.Width 3.5
# 4 setosa Petal.Width 0.2
# 5 versicolor Sepal.Length 7
# 6 versicolor Petal.Length 4.7
# 7 versicolor Sepal.Width 3.2
# 8 versicolor Petal.Width 1.4
# 9 virginica Sepal.Length 6.3
#10 virginica Petal.Length 6
#11 virginica Sepal.Width 3.3
#12 virginica Petal.Width 2.5pivot_wider()
将行里面的数据又还到列里面去, 让数据框看起来“变得更宽”

#执行如下代码,则将上题数据还原回去了
flatted_data <- pivot_longer(data = mini_iris,
cols = c(Sepal.Length, Sepal.Width, Petal.Length, Petal.Width),
names_to = "flower_attr",
values_to = "attr_value")
3.dplyr
#根据数据框内某字段进行排序
arrange(df,ID)
arrange(df,desc(ID)) #降序排#内连接
join_df <- inner_join(psi_df_tidy,so2_df,by=c("Date","Region"))
#缺失值drop掉
join_df <- drop_na(join_df)
#查看该数据框总体情况
summary(join_df)链式操作符%>%
将一个函数的输出传递给下一个函数作为输入
快捷键:ctrl+shift+M
各种函数
1.统计量
install.packages("pastecs")
library(pastecs)
stat.desc(df) #计算数据框中的一些基本统计量
stat.desc(df,norm=T) #增加一些描述性统计量2.分组统计
#根据so2_df数据集中的Region字段的不同值,分成几个不同的数据集,效果如下
split(so2_df,so2_df$Region) 
独立性与相关性分析
1.独立性检验
卡方独立性检验
head(Arthritis,5) #r自带数据集
# ID Treatment Sex Age Improved
#1 57 Treated Male 27 Some
#2 46 Treated Male 29 None
#3 77 Treated Male 30 None
#4 17 Treated Male 32 Marked
#5 36 Treated Male 46 Marked
df <- table(Arthritis$Treatment,Arthritis$Improved) #顺序没关系
df
# None Some Marked
# Placebo 29 7 7
# Treated 13 7 21
chisq.test(df)
# Pearson's Chi-squared test
#
#data: df
#X-squared = 13.055, df = 2, p-value = 0.001463患者接受的治疗和改善水平看上去存在某种关系( p<0.01 )
这里的p值表示从总体中抽取的样本行变量与列变量时相互独立的概率,由于p的概率值很小,所以我们拒绝了治疗类型和治疗结果相互独立的原假设。
其它独立性检验
样本量小时,推荐使用Fisher检验
fisher.test(df)mantelhaen.test(df)2.相关性检验
相关性检验指标:Pearson系数、Spearman系数、Kendall系数
显著性水平(P值),回答的问题是他们之间是否有关系,说明得到的结果是不是偶然因素导致的(具有统计学意义);相关系数回答的问题是相关程度强弱。
当P<0.01或P<0.05,则为说明水平显著。
一般相关系数在0.7以上说明关系非常紧密;0.4~0.7之间说明关系紧密;0.2~0.4说明关系一般。
假如说我得到”P<0.05,相关系数 R=0.279”,意味着二者之间确实(P<0.05)存在相关关系,而相关性为0.279。
cor()
#相关系数计算的方法(默认Pearson)
cor(state.x77) #r自带数据集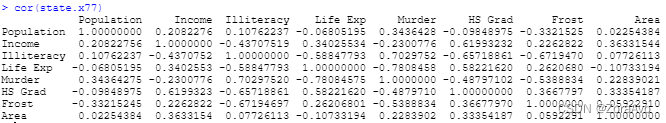
cor(state.x77,method = 'kendall')
cor(state.x77,method = 'spearman')cor.test()
cor()只给出相关系数一个值,cor.test()给出相关系数,p值等
cor.test(state.x77[,3],state.x77[,5])

corr.test()
install.packages('psych')
library(psych)
corr.test(state.x77) 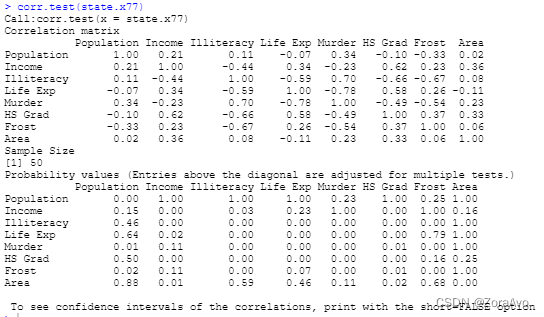
3.协方差与偏相关
协方差
协方差用来衡量两个变量的总体误差,刻画两个随机变量 x、y之间的相关性。
如果两个变量的变化趋势一致,也就是说如果其中一个大于自身的期望值,另外一个也大于自身的期望值,那么两个变量之间的协方差就是正值。如果两个变量的变化趋势相反,则反之。
协方差反映的东西和相关系数是差不多的,之所以还要再去定义相关系数是因为协方差存在量纲的影响。
cov(state.x77)偏相关
当两个变量同时与第三个变量相关时,将第三个变量的影响剔除,只分析另外两个变量之间相关程度的过程。
p值是针对原假设H0:假设两变量无线性相关。一般假设检验的显著性水平为0.05,如果p值小于0.05,说明两变量有线性相关的关系。如果大于0.05,则一般认为无线性相关关系,至于相关的程度则要看相关系数R值,R越大,说明越相关。越小,则相关程度越低。
install.packages('ggm')
library(ggm)
pcor(c(1,5,2,3,6),cov(state.x77))
#[1] 0.34627244.t检验
主要用于样本含量较小(例如n < 30),总体标准差σ未知的正态分布。通过比较不同数据的均值,研究两组数据之间是否存在显著差异。
install.packages("MASS")
library(MASS)
t.test(Prob~so,data=UScrime)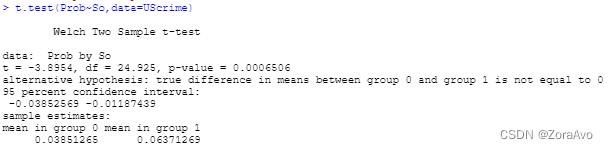
绘图
plot()
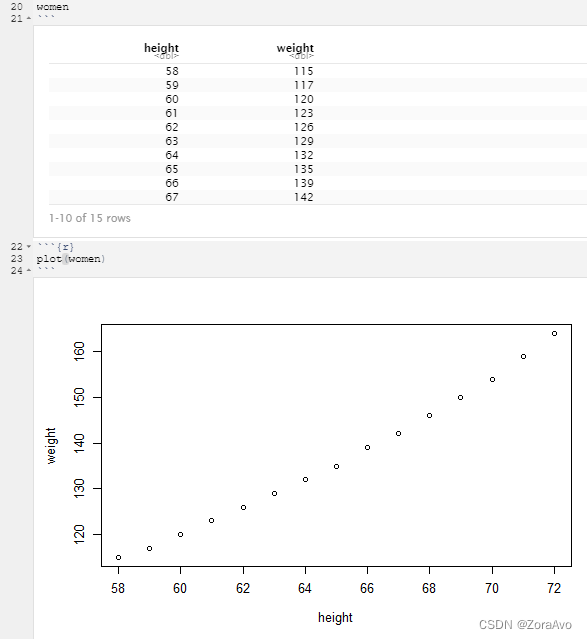
例子:针对join_df_full这个数据集
install.packages('ggplot2')
library(ggplot2)ggplot(join_df_full)+geom_bar(aes(x=Region),stat='count')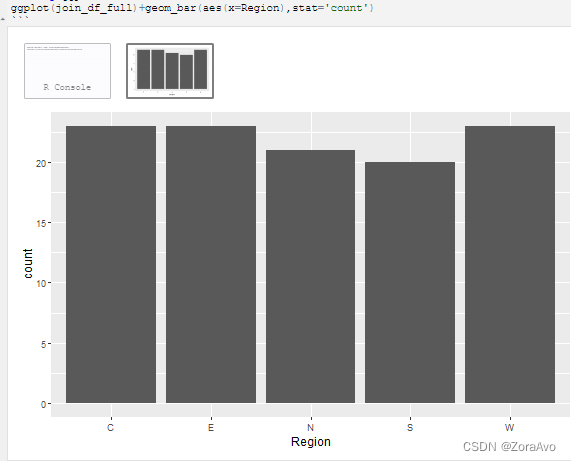
ggplot(join_df_full)+geom_histogram(aes(x=SO2),bins=10 ) 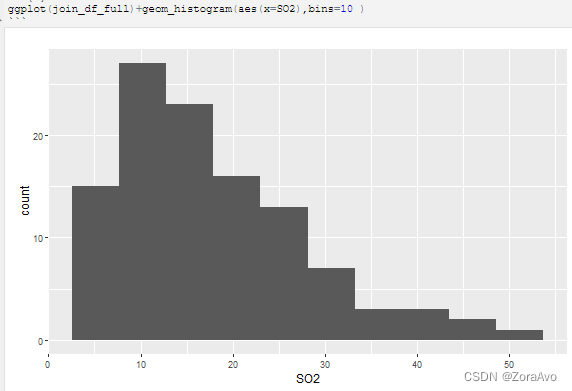
ggplot(join_df_full)+geom_dotplot(aes(x=Region,y=PM10),binaxis='y',stackdir = 'center',stackratio = 1) #stackratio=0就是同样的数据的点会重叠 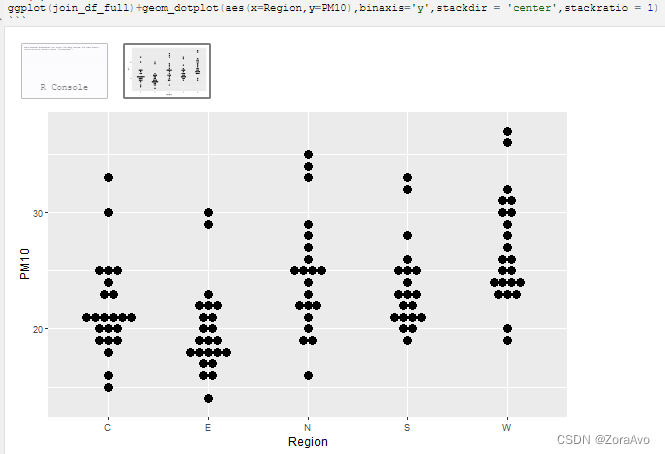
ggplot(join_df_full)+geom_boxplot(aes(x=Region,y=PM10))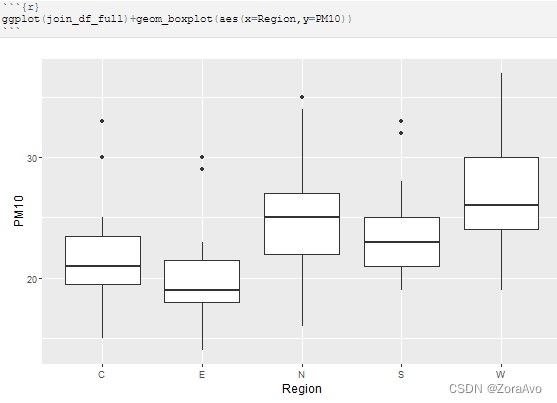
ggplot(join_df_full)+geom_point(aes(x=SO2,y=PSI)) 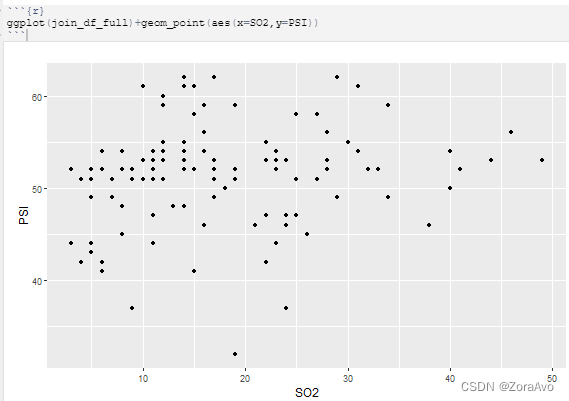
线性回归

1.普通最小二乘法
残差越接近0,模型越精确。残差其实就是真实值和预测值间的差值。
x <- lm(formula = weight~height,women) #women为自带数据集
根据上述结果:weight=3.45*height-87.52
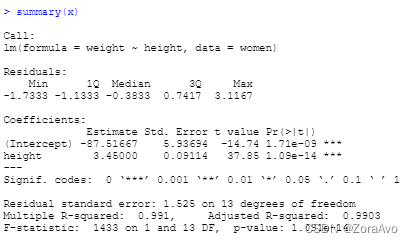
p值越小,越具有统计学意义

画出拟合图:
plot(women$height,women$weight)
abline(x)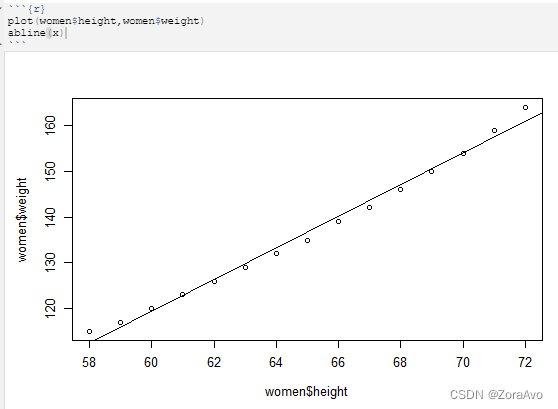
2.多元线性回归
sta <- as.data.frame(state.x77)
x <- lm(Murder ~ Population+Illiteracy+Income+Frost,data=sta)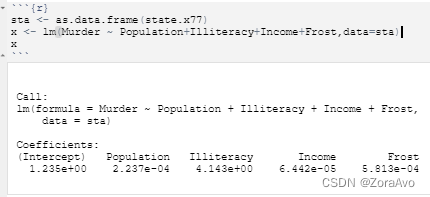
比较哪个模型(含有不同影响因素变量的模型)的拟合度更高:
结果AIC值越小,说明越越少的参数就能获得足够的拟合度
sta1 <- lm(Murder ~ Population+Illiteracy+Income+Frost,data=sta)
sta2 <- lm(Murder ~ Population+Illiteracy,data=sta)
AIC(sta1,sta2)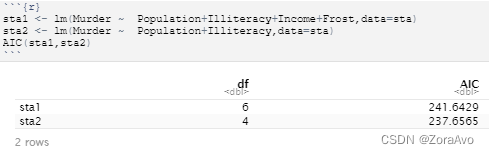
说明sta2的模型更优
3.广义线性回归
用glm() 进行广义线性回归的拟合
泊松回归
install.packages('robust')
library(robust)
data(breslow.dat,package='robust') #library并不是完全加载,data()进一步加载
attach(breslow.dat) #锁住该数据集,就不用aaa$bbb了,直接bbb
x <- glm(sumY ~ Base + Trt + Age, data = breslow.dat, family = poisson(link = 'log'))
summary(x) 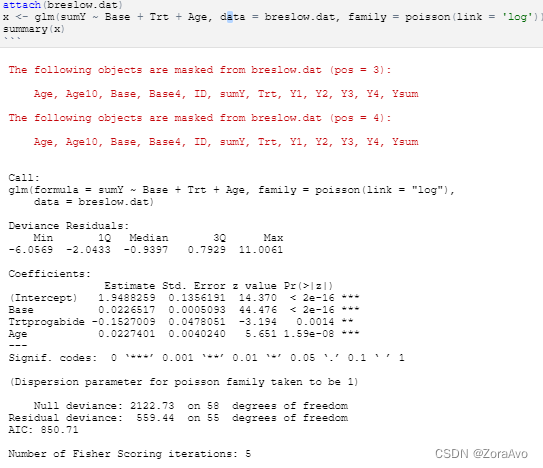
Logistic回归
预测二值型结果变量
方差分析
单因素方差分析
R中默认:当用如下表达式对数据进行建模
Y ~ A + B +A:B ,则A为主效应,B根据A调整,A:B交互性再根据A和B做调整
aov():
#利用自带数据集cholesterol举例
x <- aov(cholesterol$response ~ cholesterol$trt, data = cholesterol)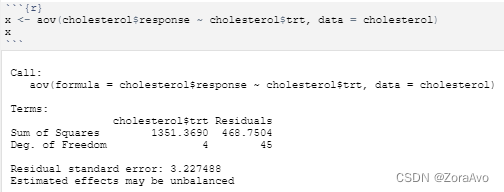
双因素方差分析
协方差分析
多因素方差分析
功效分析
在给定置信度的情况下,判断所需样本量,也可以计算某样本量内能检测到给定效应值的概率
1.
library(pwr)
pwr.f2.test(u=3,sig.level=0.05,power=0.9,f2=0.0769)
结果表明,假定显著性水平为0.05,在功效水平0.9下,至少有185个受试者才行
2.
pwr.anova.test(k=2,f=0.25,sig.level=0.05,power=0.9)

主成分分析与因子分析
1.主成分分析
主成分分析是一种降维算法,它能将多个的大量的指标转换为少数几个主成分,这些主成分是原始变量的线性组合,且彼此之间互不相关,其能反映出原始数据的大部分信息。
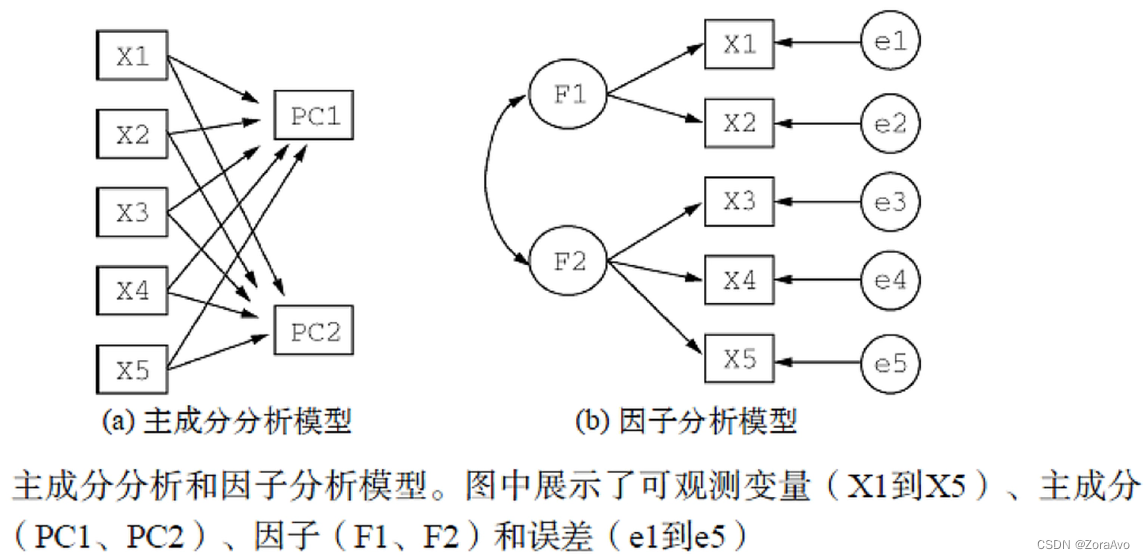
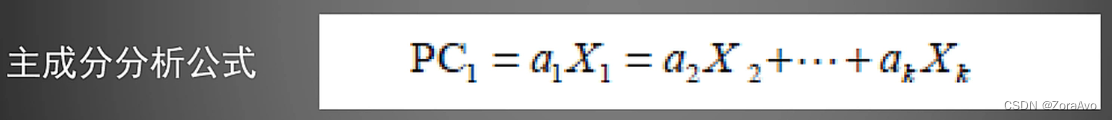
举例:

主成分分析与因子分析步骤:
1、数据预处理;(计算前确保数据中没有缺失值)
2、选择分析模型;
3、判断要选择的主成分/因子数目;(通过碎石图)4、选择主成分/因子;
5、旋转主成分/因子;6、解释结果;
碎石图:
原则是:要保留的主成分的个数的特征值要大于1且大于平行分析的特征值。
#绘制碎石图
#fa="pc"表示现实主成分的特征值
library(psych)
fa.parallel(USJudgeRatings,fa='pc',n.iter=100)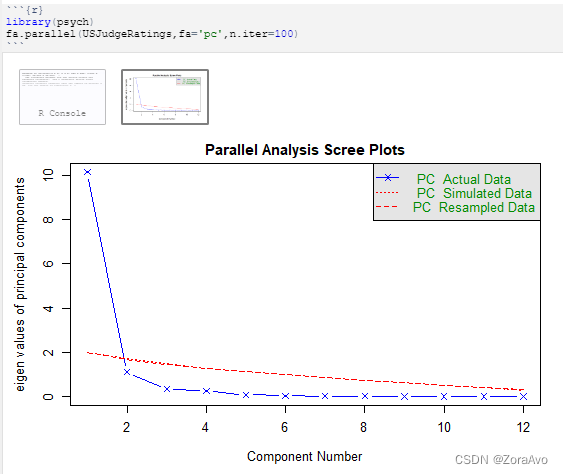
碎石图(直线与x符号)的特征值(x)大于1,同时大于100次模拟的平行分析(虚线),因此可得保留一个主成分即可。
主成分分析
#nfactors指主成分个数
#rotate指旋转的方法
#scores指是否需要计算主成分得分
pc <- principal(USJudgeRatings,nfactors = 1,rotate = 'none',scores = F)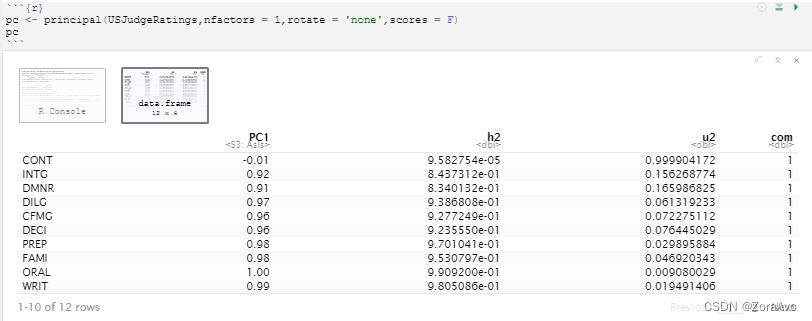
PC1包含了成分载荷,指观测变量与主成分的相关系数
h2栏指成分公因子方差,即主成分对每个因子变量的方差解释度
u2栏指主成分唯一性,即方差无法对主成分解释的比例
#获得每个变量的得分
pc <- principal(USJudgeRatings,nfactors = 1,rotate = 'none',scores = T)
pc$scores2.因子分析

公共因子是虚拟的
购物篮分析
apriori()是关联规则挖掘算法
install.packages('arules')
library(arules)
data("Groceries")
result <- apriori(Groceries,parameter = list(support=0.01,confidence=0.5))
#最小支持度是0.01
#最小置信度为0.5
result
inspect(result)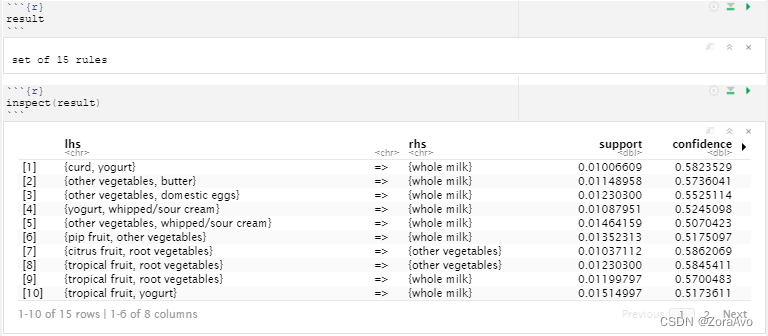
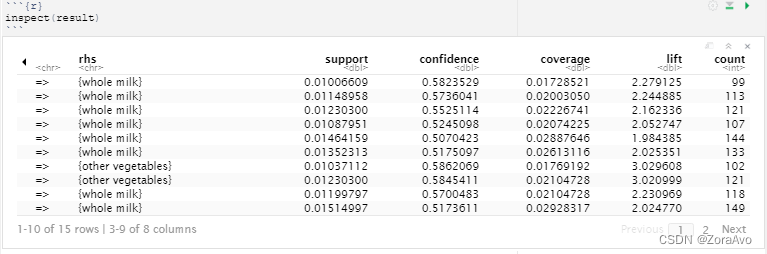
表示例如,如果有curd和yogurt就会有whole milk。lift类似相关性,系数越大相关度越大。






















 3363
3363











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








