pyg自带数据集代入测试(二)
写在前面:
由于要先复习一下框架中模型是如何运行的并且使用框架中的数据集就不需要做额外的处理了,所以今天的主要工作就是将pyg中提供的框架参数进行复现。接下来是具体步骤以及注意事项:
1. 根据框架内的基本代码复现一下基本的数据集的结构:
import torch
from torch_geometric.data import Data
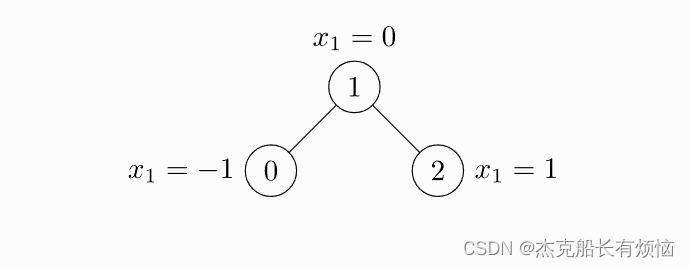
edge_index = torch.tensor([[0, 1, 1, 2],
[1, 0, 2, 1]], dtype=torch.long)
x = torch.tensor([[-1], [0], [1]], dtype=torch.float)
data = Data(x=x, edge_index=edge_index)
print(data)
>>> Data(edge_index=[2, 4], x=[3, 1])
#返回属性名列表
print(data.keys)
>>> ['x', 'edge_index']
for key, item in data:
print(f'{key} found in data')
>>> x found in data
>>> edge_index found in data
print(data['x'])
>>> tensor([[-1.0],
[0.0],
[1.0]])
'edge_attr' in data
>>> False
# 相当于x.shape[0]
data.num_nodes
>>> 3
# 相当于 edge_index.shape[1]
data.num_edges
>>> 4
data.num_node_features
>>> 1
#是否存在孤立点
data.has_isolated_nodes()
>>> False
#是否存在自环
data.has_self_loops()
>>> False
#是否是有向图
data.is_directed()
>>> False
# 将数据移动到GPU中进行训练
device = torch.device('cuda')
data = data.to(device)
接下来用一个真实的数据集Cora来进行测试,具体代码如下:
这里插一个题外话,要是数据集下载不下来的话,可以点进程序中的类里面看下,里面都会附一个下载链接,一般是在github上,希望能帮到看我这篇博客的人。
from torch_geometric.datasets import Planetoid
# 路径这里写道数据集的根目录就行
dataset = Planetoid(root='E:\data', name='Cora')
>>> Cora()
# 只包含一个图
len(dataset)
>>> 1
# 这个图里面有七个分类
dataset.num_classes
>>> 7
# 节点的特征维度是1433
dataset.num_node_features
>>> 1433
2. 模型测试
先加载一下数据集
from torch_geometric.datasets import Planetoid
dataset = Planetoid(root='/tmp/Cora', name='Cora')
>>> Cora()
接下来我们实现两层GCN
import torch
import torch.nn.functional as F
from torch_geometric.nn import GCNConv
class GCN(torch.nn.Module):
def __init__(self):
super().__init__()
self.conv1 = GCNConv(dataset.num_node_features, 16)
self.conv2 = GCNConv(16, dataset.num_classes)
def forward(self, data):
x, edge_index = data.x, data.edge_index
x = self.conv1(x, edge_index)
x = F.relu(x)
x = F.dropout(x, training=self.training)
x = self.conv2(x, edge_index)
return F.log_softmax(x, dim=1)
# 从上面的代码不难看出,在__init__()中定义了网络层,在forword中实现了前向传播过程,激活函数使用relu。
3. 训练模型:
device = torch.device('cuda' if torch.cuda.is_available() else 'cpu')
model = GCN().to(device)
data = dataset[0].to(device)
optimizer = torch.optim.Adam(model.parameters(), lr=0.01, weight_decay=5e-4)
model.train()
for epoch in range(200):
optimizer.zero_grad()
out = model(data)
loss = F.nll_loss(out[data.train_mask], data.y[data.train_mask])
loss.backward()
optimizer.step()
4. 最后我们评估一下模型
model.eval()
pred = model(data).argmax(dim=1)
correct = (pred[data.test_mask] == data.y[data.test_mask]).sum()
acc = int(correct) / int(data.test_mask.sum())
print(f'Accuracy: {acc:.4f}')
>>> Accuracy: 0.8150
附录:
PPI:
(生物化学结构) 网络是蛋白质相互作用(Protein-Protein Interaction,PPI)网络的简称,在GCN中主要用于节点分类任务,PPI是指两种或以上的蛋白质结合的过程,通常旨在执行其生化功能。一般地,如果两个蛋白质共同参与一个生命过程或者协同完成某一功能,都被看作这两个蛋白质之间存在相互作用。多个蛋白质之间的复杂的相互作用关系可以用PPI网络来描述。
PPI数据集共24张图,每张图对应不同的人体组织,平均每张图有2371个节点,共56944个节点818716条边,每个节点特征长度为50,其中包含位置基因集,基序集和免疫学特征。基因本体基作为label(总共121个),label不是one-hot编码。
Cora:
该数据集共2708个样本点,每个样本点都是一篇科学论文,所有样本点被分为8个类别,类别分别是1)基于案例;2)遗传算法;3)神经网络;4)概率方法;5)强化学习;6)规则学习;7)理论
每篇论文都由一个1433维的词向量表示,所以,每个样本点具有1433个特征。词向量的每个元素都对应一个词,且该元素只有0或1两个取值。取0表示该元素对应的词不在论文中,取1表示在论文中。所有的词来源于一个具有1433个词的字典。
每篇论文都至少引用了一篇其他论文,或者被其他论文引用,也就是样本点之间存在联系,没有任何一个样本点与其他样本点完全没联系。如果将样本点看做图中的点,则这是一个连通的图,不存在孤立点。
词向量的每个元素都对应一个词,且该元素只有0或1两个取值。取0表示该元素对应的词不在论文中,取1表示在论文中。所有的词来源于一个具有1433个词的字典。
每篇论文都至少引用了一篇其他论文,或者被其他论文引用,也就是样本点之间存在联系,没有任何一个样本点与其他样本点完全没联系。如果将样本点看做图中的点,则这是一个连通的图,不存在孤立点。

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








