应小伙伴的需求,我来做一个脑电源模型构建的教程。在这个教程中,我假设大家都已经知道源模型是什么了。
目录
1. 引言
需要明确一点,在正问题建模中源模型构建指的是构建源网格,其目标是为脑电正问题建模提供几何结构表达。因此,所构建的源模型应该至少包括两个内容:一个是描述源位置的坐标点(顶点);一个是描述灰质皮层表面的顶点连接关系的单元。假设一个源模型存在M个顶点,由于坐标点由 [x,y,z] 三个值确定,因此顶点的维度为M*3或3*M;然后,三点确定一个平面,因此一个单元至少应该包含三个顶点,这就是我们所说的三角形单元,也有多边形单元,最常见的就是四边形单元。下面我们介绍fieldtrip的源模型构建。
fieldtrip的源模型构建是基于Freesurfer完成的,顶点维度为M*3,在源模型中标记为”pos“,单元为三角形单元,在源模型中标记为”tri“。下面为一个常见源模型中所包含的数据。

其余数据:"brainstructure"中仅包含两个数据“1”和“2”,“1”表示左脑,“2”表示右脑;“baianstructurelabel”包含两个cell—{'CORTEX_LEFT' }、{'CORTEX_RIGHT'},也就是“brainstructure”中数字“1”和“2”对应的标签;inside表示颅内坐标点的编号,最终生成的脑电正问题模型仅包含inside中逻辑值为1的内容。
2. 方法
官方教程已经给出了很详细的构建步骤:Creating a sourcemodel for source reconstruction of MEG or EEG data - FieldTrip toolbox
下面我写一下我自己做的过程,其中一些具体的代码问题我已经太熟悉了可能没有办法捕捉到当时做的细节,但是我会在最后附上我整个头模型、源模型和电极模型构建的代码。
源模型构架包括三部分内容:Freesurfer处理前期准备+Freesurfer进行源模搭建+Freesurfer处理后数据读入及坐标变换。
2.1 Freesurfer处理前准备
处理前需要准备好:ctf坐标系下的“.mgz”文件,acpc坐标系下的“.mgz”文件,和两个坐标系转换矩阵。坐标系的转换如下链接:
fieldtrip学习——1.坐标系介绍(ctf坐标系和acpc坐标系简介)-CSDN博客
Freesurfer处理前准备:保存好4个文件
| 文件名 | 描述 | 使用函数 |
| Sub_transform_vox2ctf.mat | 体素到ctf坐标系的转换矩阵 | ft_volumerealign |
| Sub_transform_vox2acpc.mat | 体素到acpc坐标系的转换矩阵 | ft_volumerealign |
| Subctf.mgz | ctf坐标系下.mgz格式的头模型 | ft_volumewrite |
| Sub.mgz | acpc坐标系下.mgz格式的头模型 | ft_volumewrite |
"Sub"为你设置的被试名;Freesurfer需要保证MRI维度为256*256*256,因此“Sub_transform_vox2ctf.mat”和“Sub_transform_vox2acpc.mat”应该是在重采样为256*256*256的转换矩阵。下面分别给出mgz文件保存的代码和体素到acpc坐标系下的转换代码:
ctf坐标系下.mgz格式头模型的保存

体素转换到acpc坐标系

2.2 Freesurfer进行源模搭建
在进行如下步骤之前需要在Linux系统上搭建好Freesurfer平台,具体平台搭建方法可参考网上的教程,我当时踩了很多坑,最终觉得沉下心一点一点扒官网的教程其实是最快的。下面是官网链接:https://surfer.nmr.mgh.harvard.edu/fswiki/DownloadAndInstall
当我们已经安装好Freesurfer,那我们可以直接进行以下操作就好啦!(注意:下面“~”是你自己的文件夹路径,不要直接复制)
- 将刚刚产生的2个.mgz文件(Sub.mgz, Subctf.mgz)复制到Ubuntu的~/Subjects文件夹下面。
- Ctrl+Alt+T打开终端
- 将将一下命令一句一句复制进终端进行操作
| 设置环境变量 | export FREESURFER_HOME=/usr/local/freesurfer | 无脑复制 |
| export SUBJECTS_DIR=~/Subjects | 无脑复制 | |
| export SUBJECTNAME=Sub(修改名字后更改) | 一般为被试名字 | |
| 转成Freesurfer认识的文件名 | mksubjdirs $SUBJECTS_DIR/$SUBJECTNAME | 无脑复制 |
| cp $SUBJECTS_DIR/$SUBJECTNAME.mgz $SUBJECTS_DIR/$SUBJECTNAME/mri/$SUBJECTNAME.mgz | 无脑复制 | |
| cd $SUBJECTS_DIR/$SUBJECTNAME/mri | 无脑复制 | |
| mri_convert -c -oc 0 0 0 $SUBJECTNAME.mgz orig.mgz | 无脑复制 | |
| cp orig.mgz orig/001.mgz | 无脑复制 | |
| 源模重建 | recon-all -autorecon1 -subjid $SUBJECTNAME(大概3小时) | 无脑复制 |
| recon-all -autorecon2 -subjid $SUBJECTNAME(大概6小时) | 无脑复制 | |
| recon-all -autorecon3 -subjid $SUBJECTNAME(大概3小时) | 无脑复制 | |
| 创建workbench文件夹 | cd(进入用户路径下) | 无脑复制 |
| cd ~/1--Experiments/Code/Functions/fieldtrip-20201229/bin/ (这个是我的) | 无脑复制 | |
| ./ft_postfreesurferscript.sh ~/Subjects SUBJECTNAME ~/template | 无脑复制 |
- 最后将~/Subjects/Sub文件夹下的workbench文件复制到U盘,转到Windows系统的matlab操作即可。
2.3 Freesurfer处理后数据读入及坐标变换
- 数据读入:读入左右脑皮层数据
cd ~\workbench
subjectname = ‘Sub’;
filename = fullfile([subjectname,'.L.midthickness.8k_fs_LR.surf.gii']);
sourcemodel = ft_read_headshape({filename, strrep(filename, '.L.', '.R.')}); %加- 坐标转换:将读入数据从acpc坐标系转换到ctf坐标系
load(fullfile([subjectname,'_transform_vox2acpc']));
load(fullfile([subjectname,'_transform_vox2ctf']));
transform_acpc2ctf = transform_vox2ctf/transform_vox2acpc; %求坐标系转换矩阵
sourcemodel = ft_transform_geometry(transform_acpc2ctf, sourcemodel); %坐标系转换- 构建sourcemodel结构体
sourcemodel.inside = sourcemodel.atlasroi>0;
sourcemodel = rmfield(sourcemodel, 'atlasroi');
save(fullfile(sprintf('%s_sourcemodel',subjectname)), 'sourcemodel');3. 检查
完成建模,必须要检查模型的正确性。源模型的检查,主要是要检查源模型和头模型是否匹配。包括:源模型的前后是否正确,源模型是否正确包含在颅骨以内。如下为检查代码:
figure
hold on
%% 绘制头组织
ts = 3; % 头皮组织编号,也可以是颅骨组织
figure
mesh2 =[];
mesh2.hex = headmodel.hex(headmodel.tissue==ts,:); %mesh2.hex(1:size(mesh2.hex),:);
mesh2.pos = headmodel.pos;
mesh2.tissue = headmodel.tissue(headmodel.tissue==ts,:); %mesh.tissue(1:size(mesh2.hex),:);
mesh_ed = mesh2edge(mesh2);
patch('Faces',mesh_ed.poly,'Vertices',mesh_ed.pos,...
'FaceAlpha',.5,'LineStyle','none',...
'FaceColor',[1 1 1],'FaceLighting','gouraud');
xlabel('coronal');
ylabel('sagital');
zlabel('axial')
camlight;
axis on;
%% 绘制源模型
inside = sourcemodel;
outside = sourcemodel;
inside.pos = sourcemodel.pos(sourcemodel.inside, :);
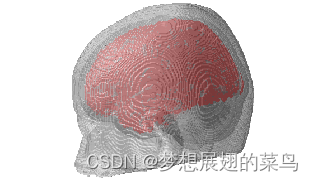
ft_plot_mesh(inside, 'vertexsize', 20, 'vertexcolor', 'red');以下是我的一个头源模型图(注意这个图片只提供参考,不可以去水印使用)
4. 头、源、电极模型构建总体代码
%% FEM头模型(head volume conduction model)的搭建+源模型(source model)的搭建
clear;
clc;
close all;
warning off
cd E:\0--Experiments\~ % cd到数据文件夹,看需要使用,不需要可注释掉
ft_defaults
%% %%%%%%%%%%%%%%%%%%%%%%%%%%%% 头模 %%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%%
% 1. read mri
subjectname = 'Sub';
SubMR=ft_read_mri('~'); % 第一张DICOM文件名
SubMR = ft_determine_coordsys(SubMR,'interactive','yes'); %查看坐标是否对齐,主要看XYZ轴方向是否对
% 2. realign
% x轴:原点指向鼻根
% y轴:原点指向左耳
% z轴:原点指向头顶
cfg = [];
cfg.coordsys = 'ctf';
cfg.method = 'interactive';
SubMR = ft_volumerealign(cfg,SubMR); %将头模型手动对齐ctf坐标,自己选择鼻根、左耳、头顶
SubMR = ft_determine_coordsys(SubMR,'interactive','yes'); %查看坐标是否对齐,主要看XYZ轴方向是否对
% 3. reslice
cfg = [];
cfg.dim = SubMR.dim;
cfg.resolution = 1; %像素为1mm
cfg.dim = [256 256 256]; %大小为256*256*256
mri_rlc1 = ft_volumereslice(cfg,SubMR); %头模型切割成1*1*1的网格
transform_vox2ctf = mri_rlc1.transform;
save(sprintf('%s_transform_vox2ctf',subjectname), 'transform_vox2ctf'); %保存坐标转换矩阵(头模型对齐到ctf坐标系)
% 4. segment
cfg = [];
cfg.output = {'gray','white','csf','skull','scalp'}; %头模型分割为灰质、白质、脑脊液、颅骨、头皮6部分
segmentedmri = ft_volumesegment(cfg,mri_rlc1);
% 5.prepare mesh
cfg = [];
% cfg.shift = 0.3; %将网格变形,是图形变得平滑,可删去
cfg.method = 'hexahedral'; %将MRI切割成六面体
mesh = ft_prepare_mesh(cfg,segmentedmri);
% 6. headmodel
cfg = [];
cfg.method = 'simbio'; %使用simbio进行FEM模型搭建
cfg.conductivity = [0.33 0.14 1.79 0.01 0.43]; %电导率设置,与上面 mesh.tissuelabel 设置顺序应一致
headmodel = ft_prepare_headmodel(cfg,mesh);
save Sub_headmodel headmodel
% 7. align the electrode
elec = ft_read_sens('D:\0--Experiments\Codes\Function\fieldtrip-20220321\template\electrode\standard_alphabetic.elc'); %电极模板路径
cfg = [];
cfg.method = 'interactive';
cfg.elec = elec;
cfg.headshape = headmodel;
elec_aligned = ft_electroderealign(cfg); %将eeg电极对齐到ctf坐标系下
save elec_aligned elec_aligned;
%% %%%%%%%%%%%%%%%%%%%%% 源模--before Freesurfer %%%%%%%%%%%%%%%%%%%%
subjectname='Sub';
% 1. 将解剖数据保存为mgz格式,以备Freesurfer用(ctf坐标系下):保存到当前路径
cfg = [];
cfg.filename = sprintf('%sctf.mgz',subjectname);
cfg.filetype = 'mgz';
cfg.datatype = 'double';
cfg.parameter = 'anatomy';
ft_volumewrite(cfg,mri_rlc1);
% 2. coregister to coordinate system according to the ‘acpc’ convention
% 转换到acpc坐标系下:x轴朝前;y轴朝右;z轴朝上
cfg = [];
cfg.method = 'interactive';
cfg.coordsys = 'acpc';
cfg.datatype = 'double';
mri_acpc = ft_volumerealign(cfg, mri_rlc1); % 头模型对齐到acpc坐标系下
mri_acpc = ft_determine_coordsys(mri_acpc,'interactive','yes'); %检查坐标系是否设置正确
transform_vox2acpc = mri_acpc.transform;
save(sprintf('%s_transform_vox2acpc',subjectname), 'transform_vox2acpc'); %保存acpc转换矩阵
% 3. 将坐标变换后解剖数据保存到磁盘
cfg = [];
cfg.filename = sprintf('%s.mgz',subjectname);
cfg.filetype = 'mgz';
cfg.datatype = 'double';
cfg.parameter = 'anatomy';
ft_volumewrite(cfg, mri_acpc); %将acpc坐标系下的头模型转换为.mgz格式
%% %%%%%%%%%%%%%%%%%%%%%%%% after Fresurfer %%%%%%%%%%%%%%%%%%%%%%%%
% 1. load headshape(以备后面与坐标转换一起将头模型转换到ctf坐标系下)
% 该步进workbench文件夹
cd I:\0--DataSet\Sub\workbench
subjectname = 'Sub';
filename = fullfile([subjectname,'.L.midthickness.8k_fs_LR.surf.gii']);
sourcemodel = ft_read_headshape({filename, strrep(filename, '.L.', '.R.')}); %加载Freesurfer处理后的源模型
% 2. compute transformation
%该步进自己的model文件夹
load(fullfile([subjectname,'_transform_vox2acpc']));
load(fullfile([subjectname,'_transform_vox2ctf']));
% 3. 将源模型转换到ctf坐标系下面
transform_acpc2ctf = transform_vox2ctf/transform_vox2acpc; %求坐标系转换矩阵
sourcemodel = ft_transform_geometry(transform_acpc2ctf, sourcemodel); %将源模型转换到ctf坐标系下面
sourcemodel.inside = sourcemodel.atlasroi>0;
sourcemodel = rmfield(sourcemodel, 'atlasroi');
save(fullfile(sprintf('%s_sourcemodel',subjectname)), 'sourcemodel');
%% %%%%%%%%%%%%%%%%%%%%%% 头、源电极模型检查 %%%%%%%%%%%%%%%%%%%%%%%
figure
hold on
ts = 3;
figure
mesh2 =[];
mesh2.hex = headmodel.hex(headmodel.tissue==ts,:); %mesh2.hex(1:size(mesh2.hex),:);
mesh2.pos = headmodel.pos;
mesh2.tissue = headmodel.tissue(headmodel.tissue==ts,:);
mesh_ed = mesh2edge(mesh2);
patch('Faces',mesh_ed.poly,'Vertices',mesh_ed.pos,...
'FaceAlpha',.5,'LineStyle','none',...
'FaceColor',[1 1 1],'FaceLighting','gouraud');
xlabel('coronal');
ylabel('sagital');
zlabel('axial')
camlight;
axis on;
ft_plot_sens(elec_aligned, 'style', '*g');
inside = sourcemodel;
outside = sourcemodel;
inside.pos = sourcemodel.pos(sourcemodel.inside, :);
outside.pos = sourcemodel.pos(~sourcemodel.inside, :);
ft_plot_mesh(inside, 'vertexsize', 20, 'vertexcolor', 'red');
ft_plot_mesh(outside, 'vertexsize', 20)
ft_plot_headmodel(headmodel, 'facealpha', 0.1)
view(125, 10)5. 相关链接
坐标转换矩阵构建:
fieldtrip学习——1.坐标系介绍(ctf坐标系和acpc坐标系简介)-CSDN博客
fieldtrip的官方教程:
Creating a sourcemodel for source reconstruction of MEG or EEG data - FieldTrip toolbox
Freesurfer官网链接:
https://surfer.nmr.mgh.harvard.edu/fswiki/DownloadAndInstall























 7201
7201











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








