论文阅读 | MAISI: 医学成像的合成数据生成新突破 | MAISI: Medical AI for Synthetic Imaging
研究背景
在现代医疗保健中,医学影像分析至关重要,它为患者的诊断、治疗计划和监测提供了关键见解。然而,医学影像分析面临着诸多挑战,如数据稀缺、标注成本高和隐私问题等。这些问题限制了机器学习模型在该领域的有效发展。合成数据生成作为一种新兴方向,通过创建人工但逼真的医学图像,可以增强现有数据集,减少对真实患者数据的依赖,并提供一种成本效益高的替代手动数据标注的方法。

论文方法
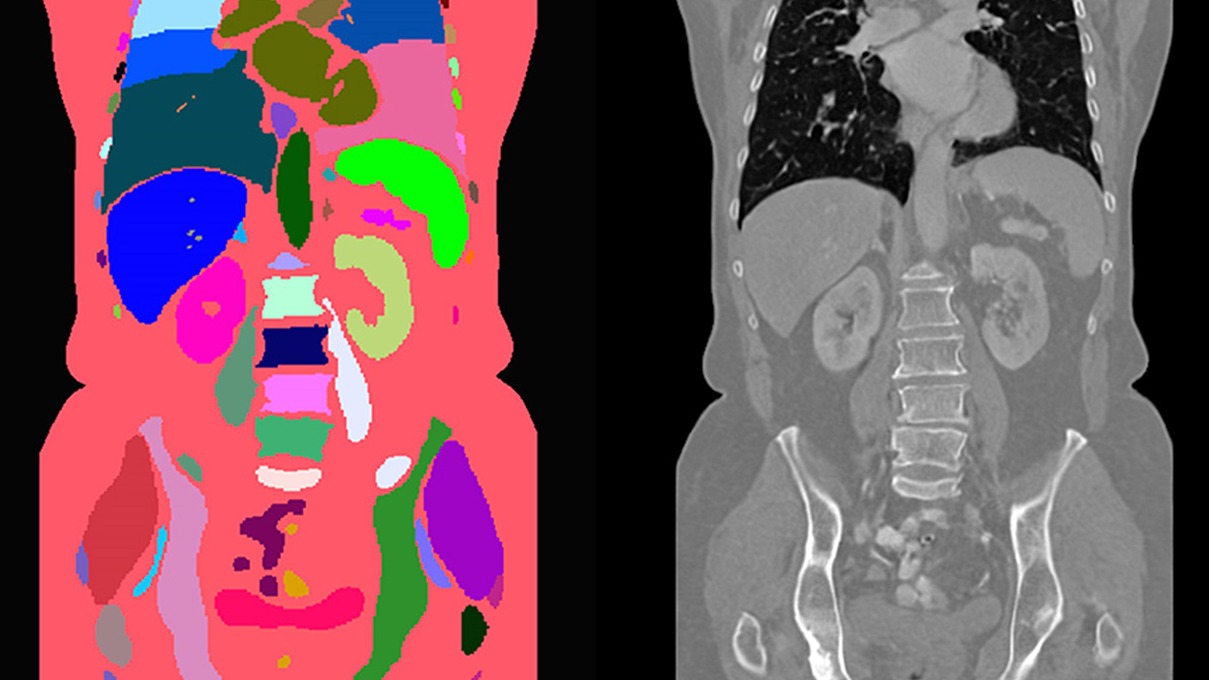
MAISI(Medical AI for Synthetic Imaging)是一种创新的基于扩散模型的方法,用于生成合成的3D计算机断层扫描(CT)图像,以应对上述挑战。MAISI利用基础体积压缩网络和潜在扩散模型,生成高分辨率CT图像,体积尺寸灵活,体素间距可调。通过引入ControlNet,MAISI能够处理包括127个解剖结构的器官分割图作为额外条件,生成带有准确标注的合成图像,适用于各种下游任务。
基础压缩网络
基础压缩网络采用变分自动编码器(VAE)训练,旨在将3D医学图像有效压缩到潜在空间中,并通过视觉编码器和解码器将生成的潜在特征映射回图像空间。为了减少内存占用,引入了张量分裂并行性(TSP),允许在统一的3D网络中对高分辨率CT体积进行编码和解码。
潜在扩散网络
潜在扩散模型在压缩的潜在空间中操作,以特定的身体区域和体素间距为条件,生成具有高度保真度的复杂解剖结构。该模型在来自不同数据集的10,277个体积上进行训练,涵盖各种身体区域和疾病条件,以增强其通用性和鲁棒性。
ControlNet支持额外条件控制
ControlNet被集成到MAISI框架中,提供了一种机制,可以在更广泛的动态控制下生成输出。它允许通过注入额外条件(如分割掩模、肿瘤掩模等)来控制生成过程,从而在不同任务或临床目标之间转换时,最小化对基础模型的广泛重新训练需求,节省时间和计算资源。

实验结果与分析
图像质量评估
通过与基线方法(如HA-GAN、DDPM、LDM等)的比较,MAISI在生成图像的保真度和解剖结构准确性方面表现出色。使用Fréchet起始距离(FID)评分,MAISI在多个数据集上均优于其他方法,生成的图像在外观上与真实图像非常相似。
| FID (Average) ↓ | MSD Task 06* | LIDC-IDRI | TCIA | |
| Real | MSD Task 06 | – | 3.987 | 1.858 |
| LIDC-IDRI | 3.987 | – | 4.744 | |
| TCIA | 1.858 | 4.744 | – | |
| Synthesis | HA-GAN | 98.208 | 116.260 | 98.064 |
| MAISI | 19.008 | 31.370 | 20.338 |
表 1. MAISI 模型和基线方法使用其发布的检查点的 Fréchet 起始距离得分,以多个公共数据集为参考,
*用于模型训练的数据集
随后,我们使用我们的数据集重新训练了几种最先进的基于扩散模型的方法。表 2 和表 3 中的结果表明,对于我们的数据集和未发现的数据集( autoPET 2023),我们的方法始终优于以前的方法。
| Method | FID (XY Plane) ↓ | FID (YZ Plane) ↓ | FID (ZX Plane) ↓ | FID (Average) ↓ |
| DDPM | 10.031 | 36.782 | 43.109 | 29.974 |
| LDM | 12.409 | 19.202 | 22.452 | 18.021 |
| HA-GAN | 10.439 | 10.108 | 10.842 | 10.463 |
| MAISI | 1.225 | 2.846 | 2.854 | 2.308 |
表 2。以我们的数据集为参考,比较我们的基础模型和重新训练的基线方法之间的 Fréchet 起始距离得分
| Method | FID (XY Plane) ↓ | FID (YZ Plane) ↓ | FID (ZX Plane) ↓ | FID (Average) ↓ |
| DDPM | 18.524 | 23.696 | 25.604 | 22.608 |
| LDM | 16.853 | 10.191 | 10.093 | 12.379 |
| HA-GAN | 17.432 | 10.266 | 13.572 | 13.757 |
| MAISI | 14.165 | 5.770 | 8.510 | 9.481 |
表 3。以 autoPET 2023 为参考,比较我们的基础模型与重新训练的基线方法之间的 Fréchet Inception Distance 得分
数据增强应用
MAISI生成的合成数据在数据增强方面表现出显著效果。通过将合成数据与真实数据结合训练分割模型,模型的性能得到了显著提升。在五种肿瘤类型上进行的实验表明,使用合成数据训练的模型在测试集上的最终性能有积极影响,平均改善约2.5%~4.5%。
| Experiment | Dataset | Tumor Type | Dice Score | Improvement |
| Real | MSD Task 06 | Lung Tumor | 0.581 | – |
| Real + Synthetic | 0.625 | 4.5% | ||
| Real | MSD Task 10 | Colon Tumor | 0.449 | – |
| Real + Synthetic | 0.490 | 4.1% | ||
| Real | In-House Bone Lesion | Bone Lesion | 0.504 | – |
| Real + Synthetic | 0.534 | 3.0% | ||
| Real | MSD Task 03 | Hepatic Tumor | 0.662 | – |
| Real + Synthetic | 0.687 | 2.5% | ||
| Real | MSD Task 07 | Pancreatic Tumor | 0.433 | – |
| Real + Synthetic | 0.473 | 4.0% |
表 4。与不同肿瘤类型的各种模型的基线性能相比,Auto3DSeg 的平均骰子得分有所提高
定性评估
图 4 显示了三例异常病例的定性评估,可以看出 MAISI 在正常器官和异常肿瘤区域都能产生良好的 CT 生成质量,如每个子图的方框所示。我们的结果表明 MAISI 能以高保真度有效地描绘异常组织边界,证明了其在医学成像中基于分割掩模条件捕捉复杂细节方面的稳健性,MAISI 有可能有效地增强生成的 CT 图像的多样性和真实性用于数据增强目的。
讨论与展望
MAISI在生成高质量CT图像方面具有巨大潜力,但仍存在一些局限性。例如,其在生成解剖学中准确表示人口统计学变化的能力尚未得到广泛验证,且生成高分辨率图像仍需大量计算资源。未来的研究可以聚焦于确保合成数据能够充分捕捉这种多样性,以避免在下游应用中出现偏差,并努力提高MAISI在资源受限环境中的可访问性。
总结
MAISI作为一种用于生成高分辨率3D CT体积的新框架,结合了基础模型和ControlNet,能够生成解剖学上准确的图像,为医学成像领域提供了灵活且通用的解决方案。实验结果表明,MAISI能够生成具有灵活体积尺寸和体素间距的真实CT图像,有望增强医学数据集并提高下游任务的性能。随着技术的不断进步和优化,MAISI有望在医学成像领域发挥更大的作用,为医疗保健带来更多的创新和突破。
关于作者

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








