FCN
1拉取代码
我的评价是直接把仓库拉取到/content
然后把对应的文件夹fcn放到mydrive里
2拉取数据集
这是代码仓库的数据集链接

我们使用下面方法下载数据集
!wget -P 路径 网址
然后我们解压tar包(时间可能会很长):
!tar -xf VOCtrainval_11-May-2012.tar
解压后可以看到

VOCdevkit
└── VOC2012
├── Annotations 所有的图像标注信息(XML文件)
├── ImageSets
│ ├── Action 人的行为动作图像信息
│ ├── Layout 人的各个部位图像信息
│ │
│ ├── Main 目标检测分类图像信息
│ │ ├── train.txt 训练集(5717)
│ │ ├── val.txt 验证集(5823)
│ │ └── trainval.txt 训练集+验证集(11540)
│ │
│ └── Segmentation 目标分割图像信息
│ ├── train.txt 训练集(1464)
│ ├── val.txt 验证集(1449)
│ └── trainval.txt 训练集+验证集(2913)
│
├── JPEGImages 所有图像文件
├── SegmentationClass 语义分割png图(基于类别)
└── SegmentationObject 实例分割png图(基于目标)
解压完成后,将train.py下数据集的路径设置为你下载的位置

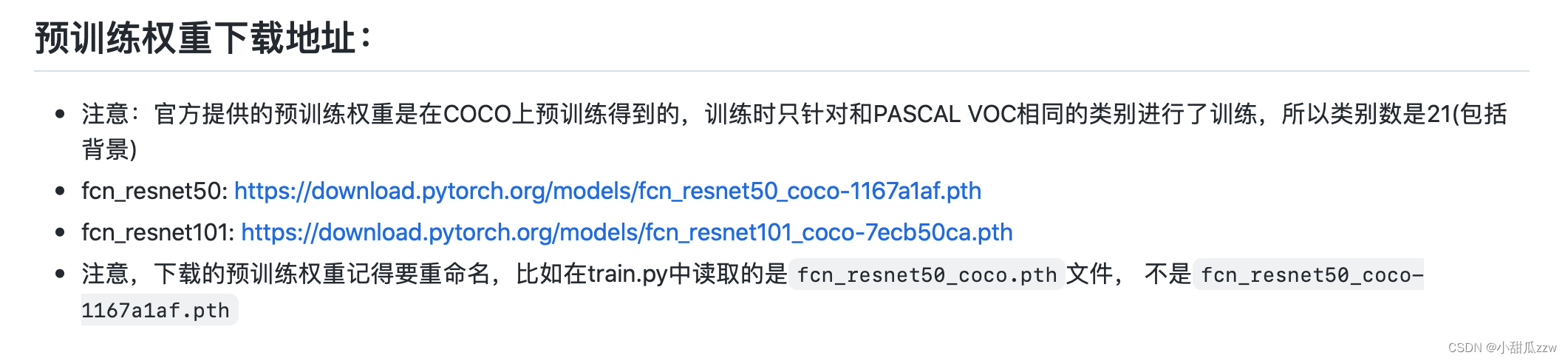
3下载预训练权重
train.py代码里的

这里也给出了下载链接
我们继续使用wget将权重下载到和train.py同级的文件夹下

记得改名噢。
4 训练
!python train.py
训练一轮结果:
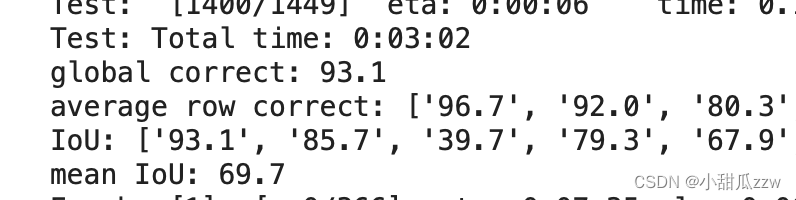
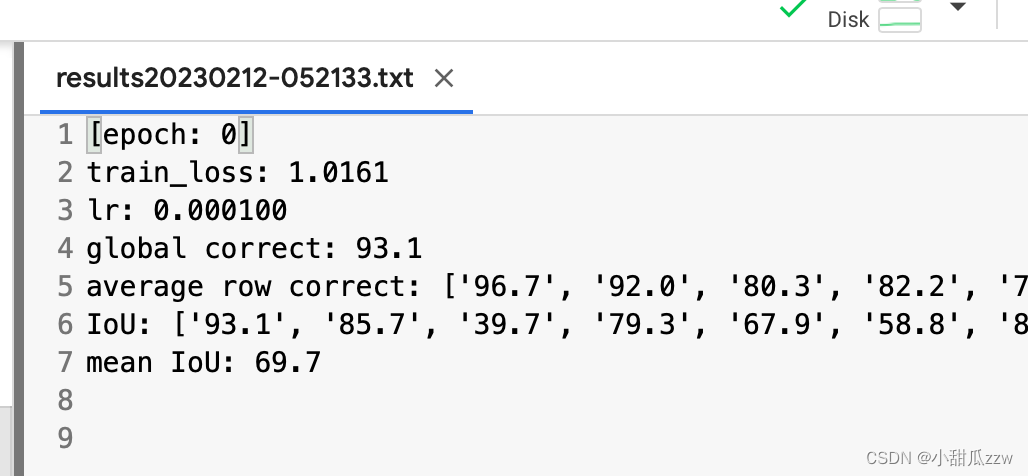
训练结果自动保存到txt
这里是训练完保存的pth(0代表epoch:0)

5预测
在predict.py里将路径改为刚才训练出来的.pth文件

放入一张图片当测试图片

看结果:

Unet(2d)
unet是对生物医学影像领域
1拉取代码(参考fcn)
2获取数据集
b导给的百度云链接下载后,再放到google drive吧。数据集不大,不一会儿就下完了。将下载好的DRIVE文件夹放到unet文件夹下
因为data-path默认路径为./

数据集(20个眼球)目录:
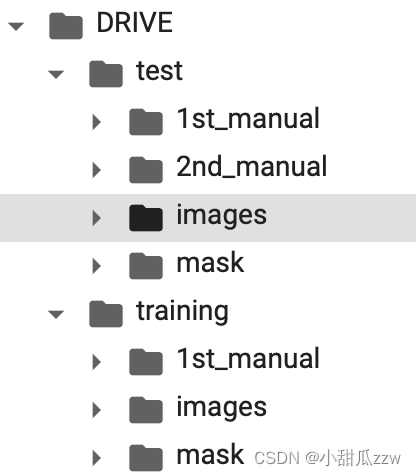
关于mask里的

指的是白色以外的区域不用管。
而manual(手动的)文件夹下是人工分割好的图片
3训练
不需要预训练权重,训练脚本和fcn的差不多。
!python train.py
训练了大概70轮, mean IoU达到80
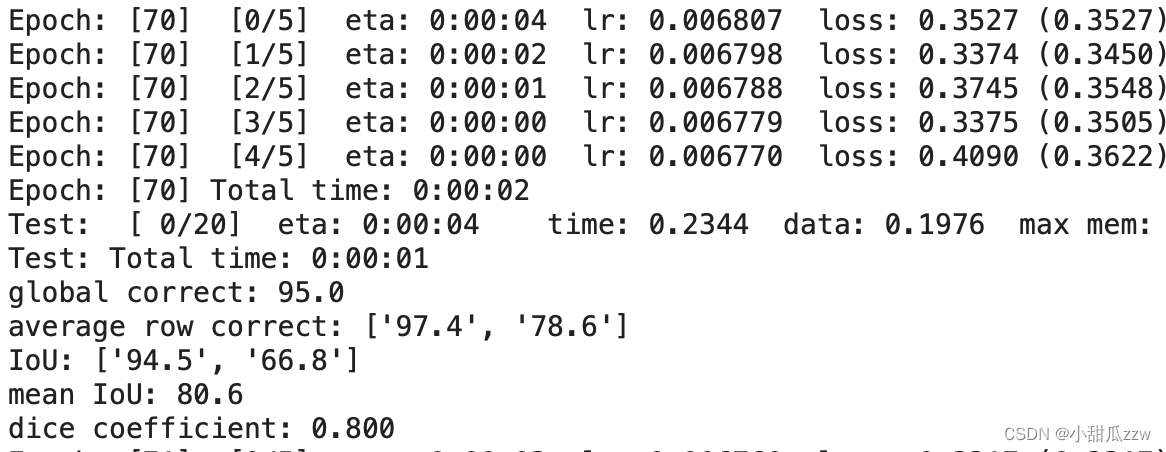
训练完保存的pth:

4预测
在predict.py下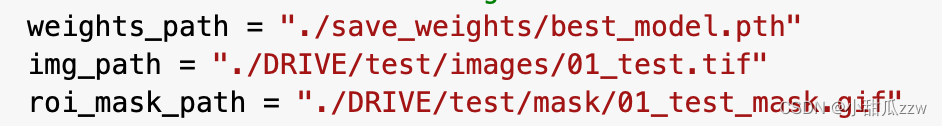
预测数据直接是test里的。
预测完会生成一张图片
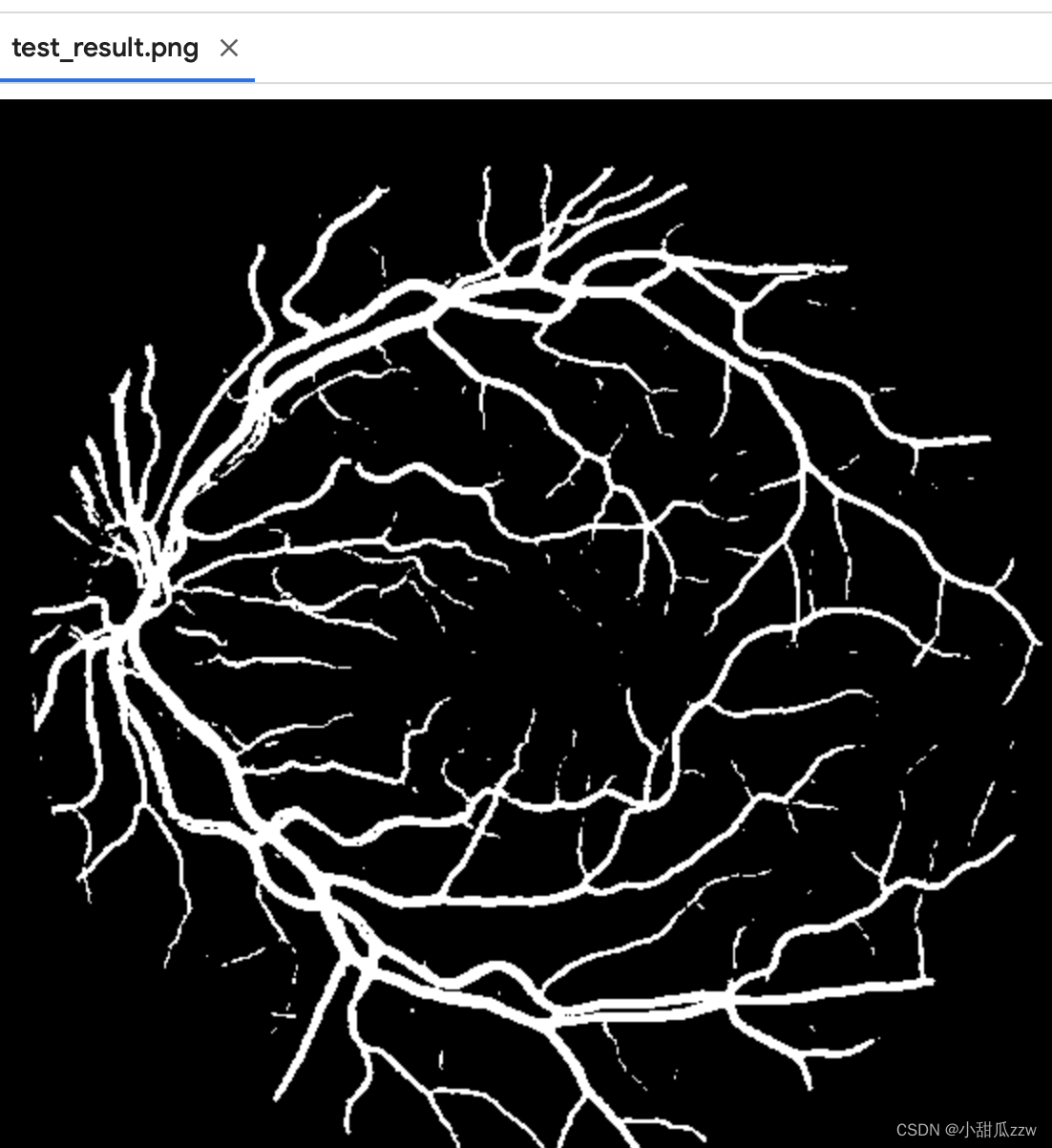
脑血管分割数据集
.mha后缀的文件打开需要3d slicer
3d slicer官网支持macOS系统。
直接将.mha文件拖到3d slicer就能看了
3d slicer
下一步思路从这个软件学习一下3d影像数据3dslicer教程
3d unet
3d-unet的实现,想试试这篇文章。
1拉取代码到google drive
2下载数据集
两种方法,但第一种好像行不通。

这篇文章使用的是这个:lits2017肝脏肿瘤数据集
数据是nii格式的
推荐的下载完的文件划分格式。
raw_dataset:
├── test # 20 samples(27~46)
│ ├── ct
│ │ ├── volume-27.nii
│ │ ├── volume-28.nii
| | |—— ...
│ └── label
│ ├── segmentation-27.nii
│ ├── segmentation-28.nii
| |—— ...
│
├── train # 111 samples(0\~26 and 47\~131)
│ ├── ct
│ │ ├── volume-0.nii
│ │ ├── volume-1.nii
| | |—— ...
│ └── label
│ ├── segmentation-0.nii
│ ├── segmentation-1.nii
| |—— ...
label文件夹里的segmentation-0.nii:
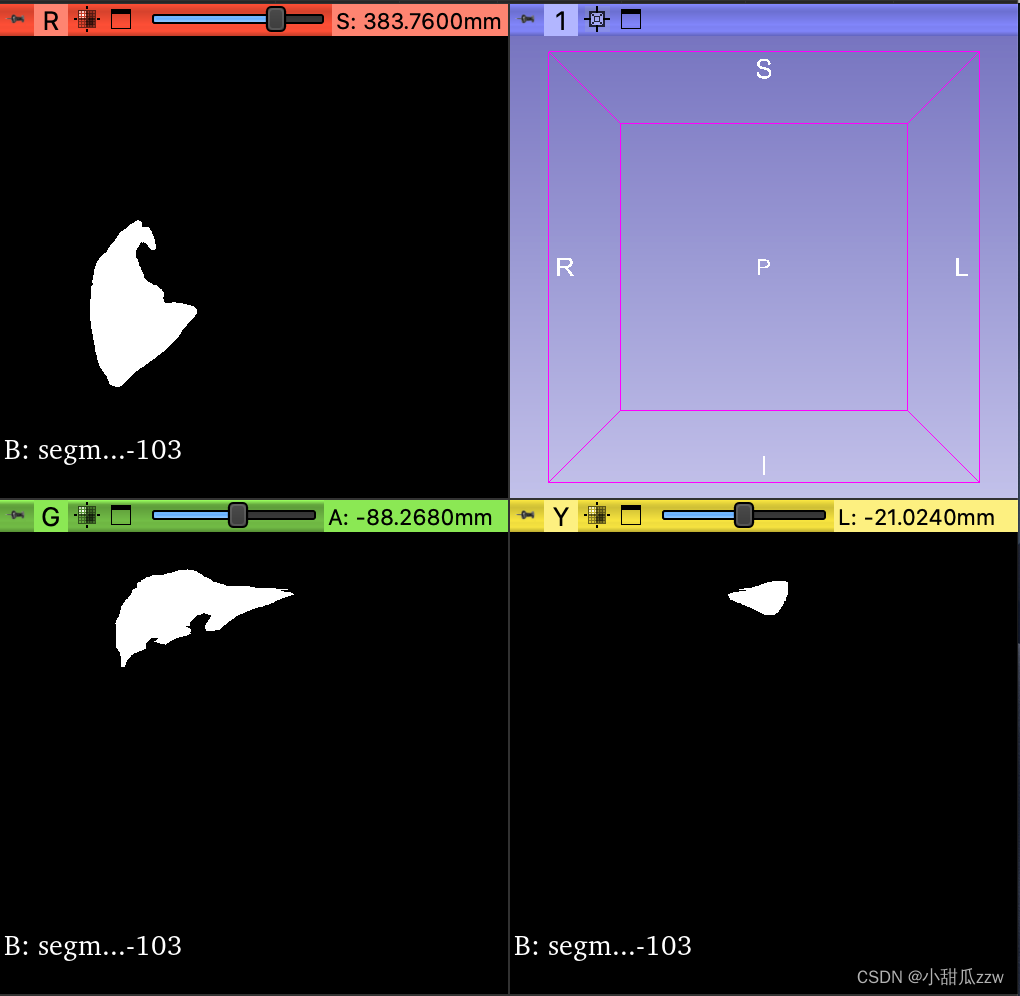
文件太大了,只用28-60.但也有5g这么多呢。
那test放28-35.train放36-60吧。
然后我们上传到google drive。
3导入必须的包
pytorch >= 1.1.0
torchvision
SimpleITK
Tensorboard
Scipy
使用
!pip install ...
4 数据预处理
然后在 preprocess_LiTS.py文件中118行更改预处理输入、输出目录:
if __name__ == '__main__':
raw_dataset_path = '/content/drive/MyDrive/raw_dataset/train' # 输入数据集路径
fixed_dataset_path = './fixed_data/' # 预处理后的数据集的输出路径
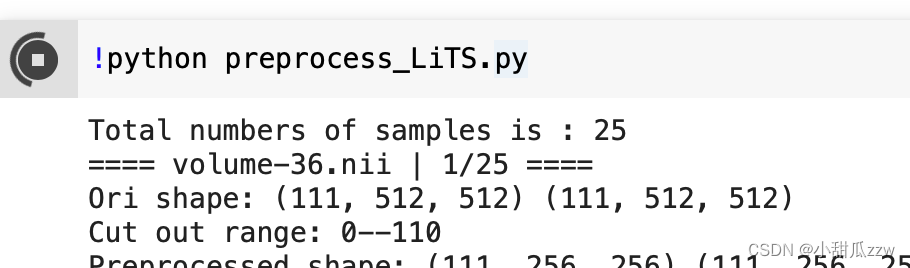
可见我们使用了25个nii文件。从36-60.
这是生成的文件夹
5模型训练
首先在 config.py中修改超参数–dataset_path为我们预处理后的数据根目录

使用这个训练
!python train.py --save model_name
发现报错1
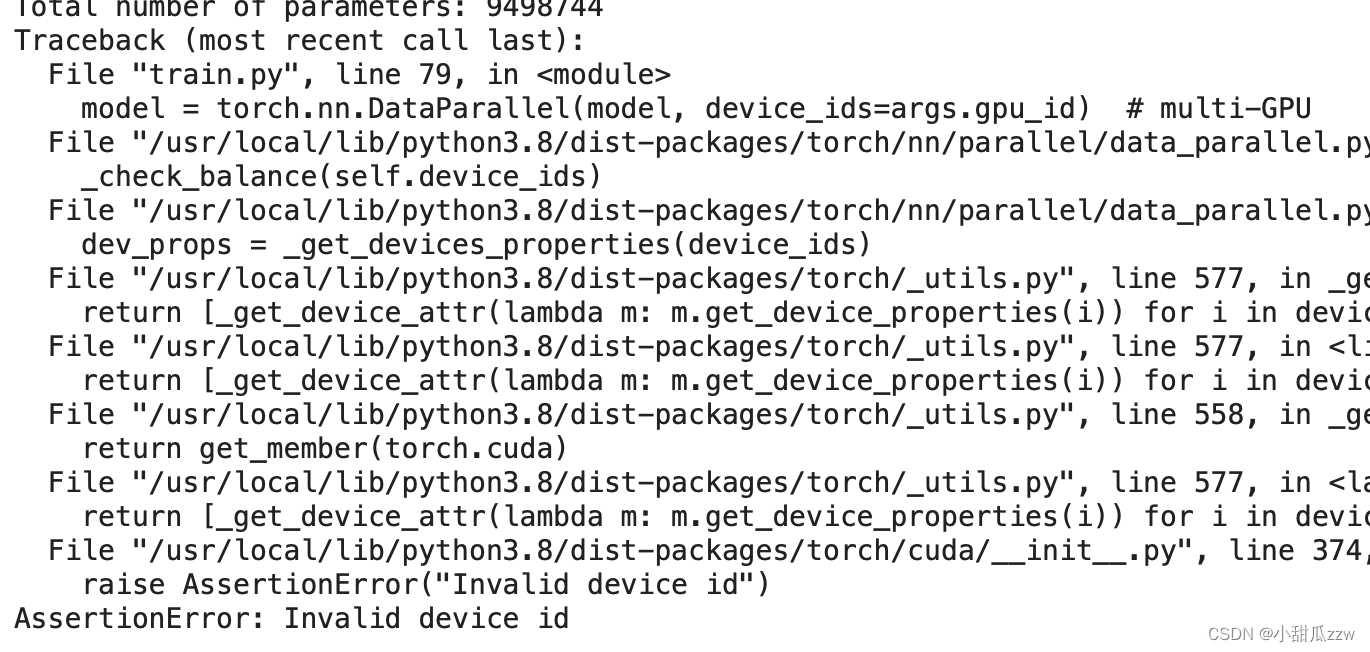
错误定位
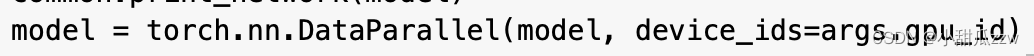

解决办法
应该是colab就提供了一个gpu,而程序默认用两个。
发现报错2

报错原因
大概意思是分类类别num_classes不对。
看config里这个,而我们用的是肝脏肿瘤数据集,所以应该改成3吧。

不对!
然后我从那个github看issue
发现和我的报错一样
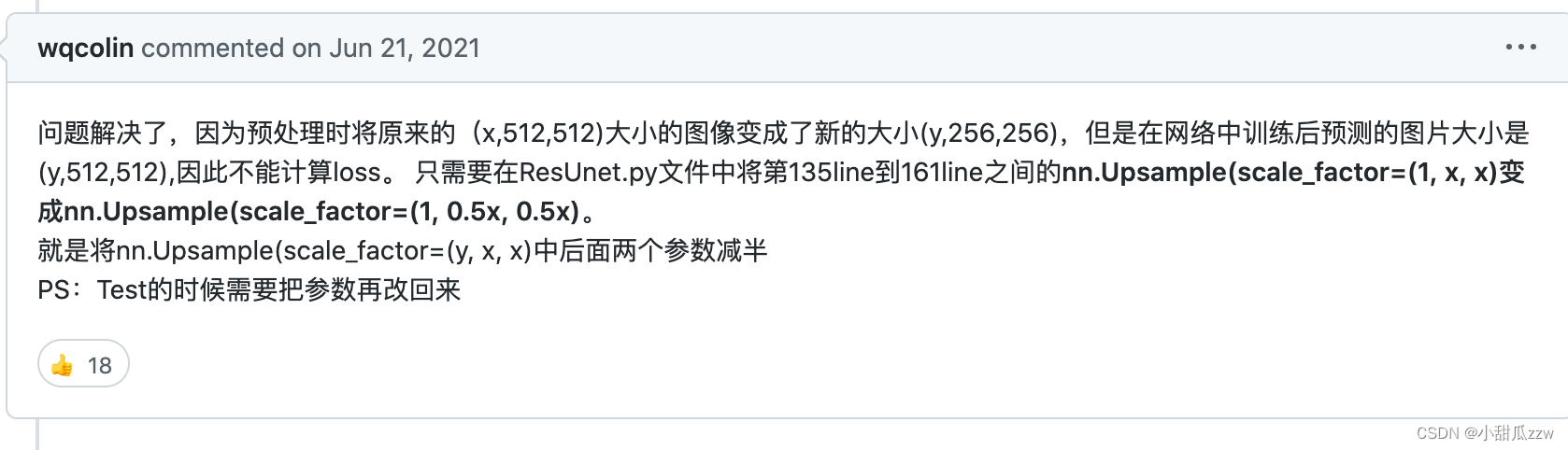
按照它的改了以后果然成功了!!!
实验结果:pth在里面

6预测
上文提到test的时候还得把参数改回来。

把test_data路径改一下

!python test.py --save model_name
报错:
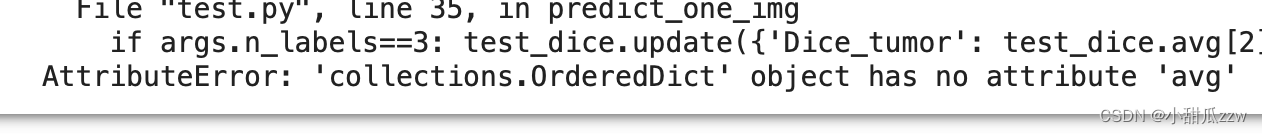






















 414
414











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








