参考资料:生物统计学
Sidak法和Bonferroni法针对LSD法犯第Ⅰ类错误风险较大的问题进行了改进,通过根据平均数个数k,减小显著水平α的值来增大t值,从而增大差数显著显著性。
Sidak法的显著水平调整公式为:
Bonferroni法的显著水平调整公式为:
Sidak法和Bonferroni法的检验尺度相差不大,Bonferroni法略偏严格。处理数不多时,对两类错误的控制较好;但处理数据较多时,检验过于严格,犯第Ⅱ类错误的风险高。
excel操作步骤如下:
1、利用数据分析工具获取方差分析结果:步骤参照
excel统计分析——单因素方差分析_为什么回归方程的假设检验用方差分析-CSDN博客
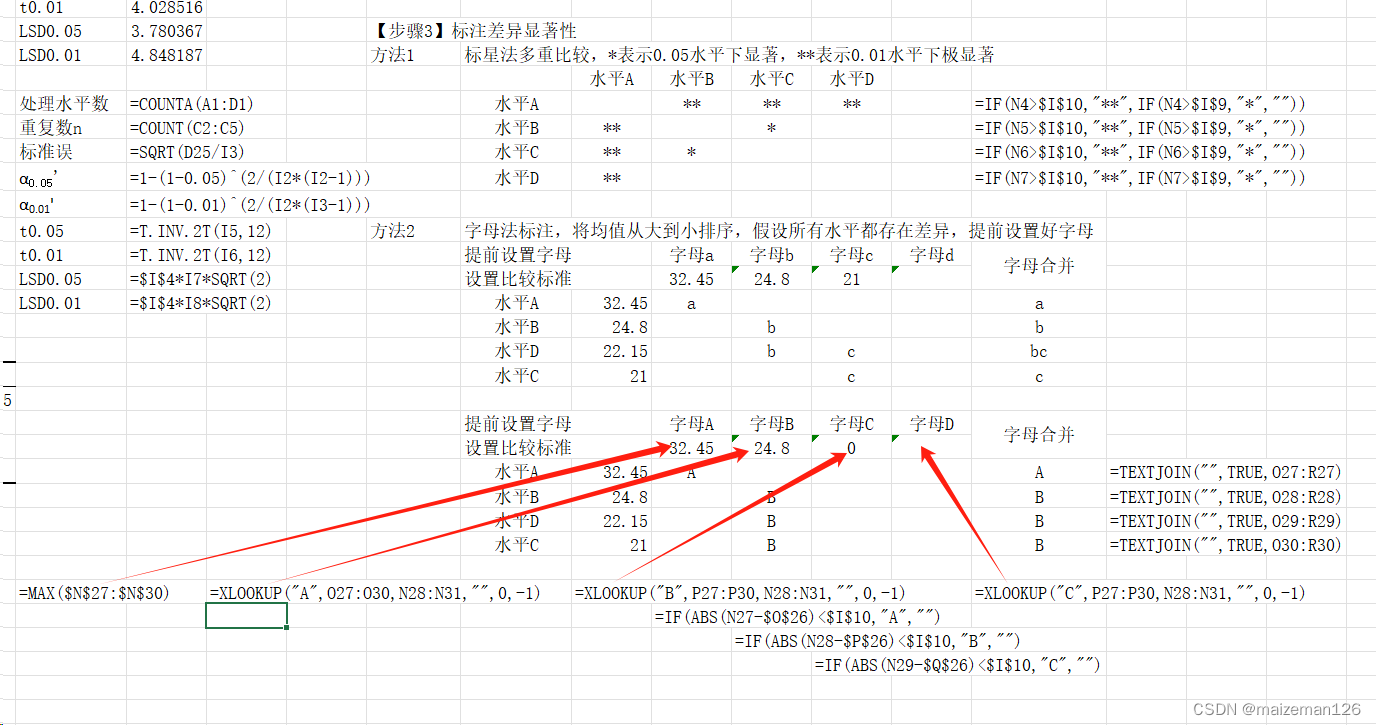
2、利用公式进行多重比较
整体操作内容可以参考excel统计分析——LSD多重比较-CSDN博客
二者的差异在于α值的调整上。Sidak法的调整公式如下:

Bonferroni法的调整公式如下:

两种方法的多重比较结果一直,结果如下:
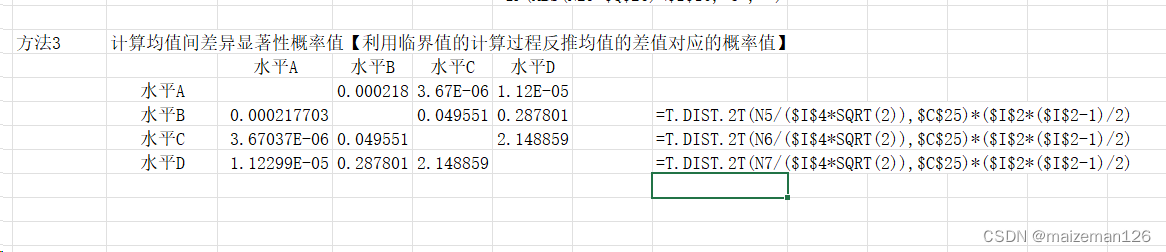
Sidak法均值间差异显著性概率如下:

Bonferroni法均值间差异显著性概率如下:其中水平C与水平D之间的均值差异概率值大于1,直接记作1即可。







 本文介绍了生物统计学中Sidak法和Bonferroni法改进LSD法以减少第一类错误的方法,讨论了两者在调整显著水平和处理大量数据时的优缺点,并详细说明了在Excel中进行方差分析和多重比较的具体步骤。
本文介绍了生物统计学中Sidak法和Bonferroni法改进LSD法以减少第一类错误的方法,讨论了两者在调整显著水平和处理大量数据时的优缺点,并详细说明了在Excel中进行方差分析和多重比较的具体步骤。














 5072
5072











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








