读《遗传算法的Python实现(通俗易懂)》佳文的思考与笔记整理.
我们拥有一个目标函数 y = 10 ⋅ s i n ( 5 x ) + 7 ⋅ c o s ( 4 x ) y=10 \cdot sin(5x)+7\cdot cos(4x) y=10⋅sin(5x)+7⋅cos(4x)
def aim(x):return 10*np.sin(5*x)+7*np.cos(4*x)
约束范围(这里是定义域):
x
∈
[
0
,
5
]
x \in [0,5]
x∈[0,5]
我们使用基因遗传算法求解
y
y
y的最大值.
文章目录
一. 基因(DNA)体现在二进制编码
首先知道十进制(整数)与二进制是一一对应的可转换的关系.我们的基因形式则为二进制数据.
bin(2**9)表示 2 9 = 512 2^9=512 29=512的二进制表达形式为'0b1000000000',取0b后面的数字,共有10位表示.bin(5)=0b101.整数5可以用二进制101代替.- 我们的定义域是一个连续的区间,最优解可能不是整数而是小数.因此我们采用更长的基因(如10个DNA)可以表达更大的整数,然后缩小到 [ 0 , 5 ] [0,5] [0,5]范围内,补充[0,5]内的小数.
- 因此数据的编码和解码公司可以参考.

- 在这里(10个DNA的情况下),设计的 decode(解码)函数为
def decode(pop):
return pop.dot(2 ** np.arange(DNA_SIZE)[::-1]) *(X_max-X_min)/ float(2**DNA_SIZE-1) +X_min
# DNA_size=10
二、个体与种群
多个基因构成一个个体.
多个个体构成一个种群.
举例假设:种群的数量为4,每个个体🈶️5个基因.则创造随机的初始种群.
pop_size=4
DNA_SIZE=5
pop = np.random.randint(2, size=(pop_size, DNA_SIZE))
pop的输出为(举例,随意random下. )
array([[1, 1, 1, 1, 0],# 物种1的基因
[0, 0, 0, 0, 0],
[1, 1, 1, 0, 0],
[1, 1, 0, 1, 0]])# 物种4的基因
三、物竞天择,适者生存
A. 计算个体的适应度
x
x
x个体(含多个DNA)–>decode解码为
x
′
x'
x′–>aim目标函数求的
y
y
y
# pred=[y1,y2,...,yn] 为pop的目标函数值
def fitnessget(pred):
return pred + 1e-3 - np.min(pred)
对于目标函数值pred同时去掉最小值np.min(pred),则pred中最小值为0.添加上 1 e − 3 = 0.01 1e-3=0.01 1e−3=0.01,防止列表中数据为0,报错.
- 适应度的计算与目标结果相关
- 求最大值时,其结果越高则适应度越高
- 求最小值时,其结果越低则适应度越高
B. 赌命运,适应度越高生存的几率越大
但这并不意味着适应度高,一定会生存下来,因此会存在一定的赌运气生存下来的情况.即是物竞天择的原理.这里的赌运气采用赌轮盘的方法.
def select(pop, fitness):
# print(abs(fitness))
# print(fitness.sum())
idx = np.random.choice(np.arange(pop_size), size=pop_size, replace=True,p=fitness/fitness.sum())
# print(idx)
return pop[idx]
C. 在没交叉没变异之前的情况,举例
依旧以 种群为4,基因为5 为例.
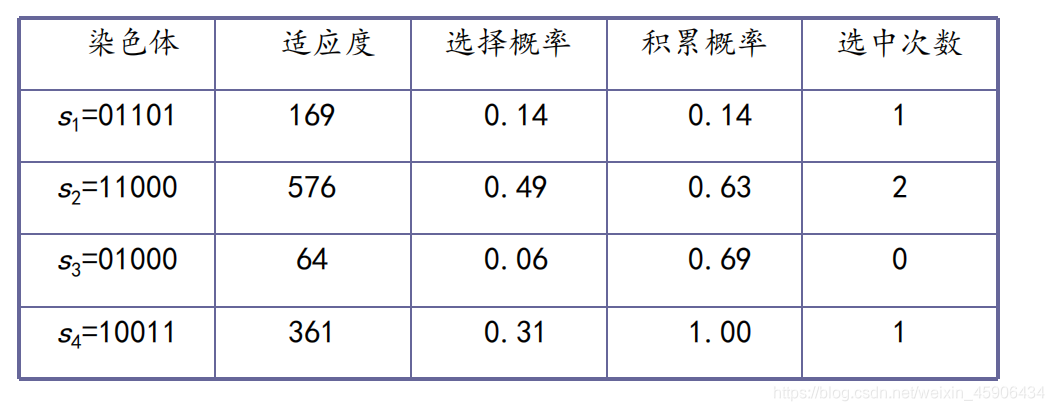

- 1号基因为1次,2号基因为2次,3号基因消失了,4号基因为1次.
- 个体总量没有发生变化,但适应度高的个体被更多的保留了下来
- 迭代下去,我们就会得到所谓的纯种个体组成的种群
- 想要适应度最高的而不是纯种个体中最优的个体
四、交叉和突变
注意:这里的交叉和变异的函数是针对基因二进制的某种方式.
A. 交叉函数

def change(parent, pop):
if np.random.rand() < PC: #交叉
i_ = np.random.randint(0, pop_size, size=1)
# print(parent)
cross_points = np.random.randint(0, 2, size=DNA_SIZE).astype(np.bool)
# print(np.where(cross_points==True))
# print(cross_points)
parent[cross_points] = pop[i_, cross_points]
# print(parent)
return parent
- 交叉概率: PC=0.6(假设)
- 交叉点:个体10个基因(DNA),随机(在每个基因上)是否生成交叉点,结果为:
cross_points- 在种群中,为parent 选择交叉的另一个pop[i_]
- 将pop[i_]上的交叉的索引cross-points赋值给parent的cross_points的索引上
B. 变异函数

def variation(child,pm): #变异
for point in range(DNA_SIZE):
if np.random.rand() < PM:
child[point] = 1 if child[point] == 0 else 0
# print(child)
return child
- 变异概率:PM=0.1(假设)
- 个体的每个基因,都有PM的变异概率
- 变异:0–>1 或1–>0
五、种群繁衍(循环迭代),适应性越来越强
- 定义初始种群(查看二):
pop = np.random.randint(2, size=(pop_size, DNA_SIZE)) - 繁衍生息(进行循环)
A: 适者生存,计算目标值和适应度
- 种群
pop—解码–>X_value- 获取目标值–>
F_values- 获得适应度–>
fitnessB:记录目标值最好的物种及其DNA
- i=0(初次循环时), 记录最好的物种为
Max其基因为max_DNA- 更新最好的物种及其基因
- 每迭代10次, 输出当前的最好的物种以及基因,查看
C: 物竞天择
- 赌命运生存,(
select函数)- 种群复制副本
pop_copy用于交叉配对的D: 交叉产生新子,新子有概率变异
每个来自于pop的物种child
- 与
pop_copy中的随机一个交叉(change函数)产生孩子child- 孩子
child可能会产生变异(variation函数)
- 迭代结束后,输出目标值最好的物种及其DNA
六、主程序代码
定义变量
# 定义全局变量
pop_size = 10 # 种群数量
PC=0.6 # 交叉概率
PM=0.01 #变异概率
X_max=5 #最大值
X_min=0 #最小值,与x_max共同构成x的定义域范围
DNA_SIZE=10 #DNA长度与保留位数有关,2**10 当前保留3位小数点
N_GENERATIONS=1000
主程序
pop = np.random.randint(2, size=(pop_size, DNA_SIZE))
# print(pop)
for i in range(N_GENERATIONS):
#解码
# print(pop)
X_value= decode(pop)
#获取目标函数值
F_values = aim(X_value)
#获取适应值
fitness = fitnessget(F_values)
# print(fitness)
if(i==0):
Max=np.max(F_values)
max_DNA = pop[np.argmax(F_values), :]
if(Max<np.max(F_values)):
Max=np.max(F_values)
max_DNA=pop[np.argmax(F_values), :]
if (i % 10 == 0):
print("Most fitted value and X: \n",
np.max(F_values), decode(pop[np.argmax(F_values), :]))
#选择
pop = select(pop,fitness)
# print(pop)
pop_copy = pop.copy()
# print(pop_copy)
for parent in pop:
# print(parent)
child = change(parent,pop_copy)
child = variation(child,PM)
# print(child)
parent[:] = child
print("目标函数最大值为:",Max)
print("其DNA值为:",max_DNA)
print("其X值为:",decode(max_DNA))
结果展示
运行一:
i: 0 Most fitted value and X:
7.384343376804747 2.7126099706744866
i: 10 Most fitted value and X:
13.372066639693031 0.21994134897360704
i: 20 Most fitted value and X:
15.560237189300736 1.4809384164222874
i: 30 Most fitted value and X:
15.560237189300736 1.4809384164222874
i: 40 Most fitted value and X:
16.975412816482176 1.5591397849462365
i: 50 Most fitted value and X:
16.975412816482176 1.5591397849462365
----------
目标函数最大值为: 16.975412816482176
其DNA值为: [0 1 0 0 1 1 1 1 1 1]
其X值为: 1.5591397849462365
运行二:
i: 0 Most fitted value and X:
11.538584764492317 1.7497556207233627
i: 10 Most fitted value and X:
13.37062486072224 2.9227761485826003
i: 20 Most fitted value and X:
13.374183016652838 2.9178885630498534
i: 30 Most fitted value and X:
13.374183016652838 2.9178885630498534
i: 40 Most fitted value and X:
13.374183016652838 2.9178885630498534
i: 50 Most fitted value and X:
13.374183016652838 2.9178885630498534
---------
i: 240 Most fitted value and X:
13.374183016652838 2.9178885630498534
i: 250 Most fitted value and X:
15.364515002303772 1.6666666666666667
i: 260 Most fitted value and X:
16.943525799534186 1.5884652981427174
i: 270 Most fitted value and X:
16.999359344557767 1.5689149560117301
--------
i: 470 Most fitted value and X:
16.999359344557767 1.5689149560117301
i: 480 Most fitted value and X:
16.943525799534186 1.5884652981427174
i: 490 Most fitted value and X:
16.970440089469758 1.5835777126099706
i: 500 Most fitted value and X:
16.970440089469758 1.5835777126099706
i: 510 Most fitted value and X:
16.991707506136905 1.5640273704789833
i: 520 Most fitted value and X:
16.999359344557767 1.5689149560117301
-------
i: 590 Most fitted value and X:
16.999359344557767 1.5689149560117301
i: 600 Most fitted value and X:
16.998364271158337 1.573802541544477
----------
i: 990 Most fitted value and X:
16.998364271158337 1.573802541544477
目标函数最大值为: 16.999359344557767
其DNA值为: [0 1 0 1 0 0 0 0 0 1]
其X值为: 1.5689149560117301
总结:
- 第一次运行到50次就接近了最优值,而第二次运行了600次接近了最优质.
- 两次最优解x都近似在1.6附近.最优值y近似在16.99上.可见该方法具有一定的稳定性和准确性.
应用思考:
- 这里只有一个变量 x x x,轻松表示其基因.如果存在多个变量 x 1 , x 2 , ⋯ , x n x_1,x_2,\cdots,x_n x1,x2,⋯,xn, 那么其基因如何表示呢?-----基因除了二进制还有多种编码方式
- 这里约束(定义域)非常简单.只有大小的范围限制. 当解决最优规划问题的时候.会存在凸性约束.那么其约束如何表示呢?
- 适应度的设计问题.这里可以看出适应度与目标值时直线关系(鞋履 k = 1 k=1 k=1).那么可否根据实际问题,重新设计 适应度的表达式呢?(比如,对数关系)
- 每一次,都需要自行编写代码嘛?(这个接下来可以解答)
七、使用库scikit-opt解决
from sko.GA import GA
import pandas as pd
import matplotlib.pyplot as plt
函数讲解
GA(func, n_dim, size_pop=50, max_iter=200, prob_mut=0.001, lb=-1, ub=1, constraint_eq=(),
constraint_ueq=(), precision=1e-07, early_stop=None)
|
| genetic algorithm
|
| Parameters
| ----------------
| func : function
| The func you want to do optimal 优化的目标函数
| n_dim : int
| number of variables of func目标函数的变量
| lb : array_like
| The lower bound of every variables of func每个变量的下限
| ub : array_like
| The upper bound of every variables of func每个变量的上限
| constraint_eq : tuple,
| equal constraint 等式约束
| constraint_ueq : tuple,
| unequal constraint不等式约束
| precision : array_like
| The precision of every variables of func 函数每个变量的精度
| size_pop : int
| Size of population种群数量
| max_iter : int
| Max of iter迭代次数
| prob_mut : float between 0 and 1
| Probability of mutation 突变概率
A. 使用GA解决上面问题
def aim(p):
x= p[0]
return -(10*np.sin(5*x)+7*np.cos(4*x))
注意到:
- 这里的
x=p[0],也就是第一个变量的目标函数.如果不加这一行.运行会有广播机制的报错问题. - 此外原目标函数为求最大值,而GA的默认时求最小值.因此,则需要加一个负号
-
ga = GA(func=aim, n_dim=1, size_pop=10, max_iter=1000,
lb=0, ub=5, precision=1e-7)
best_x, best_y = ga.run()
# `GA`是一个class类,因此要先实例化(第一行),然后用`run`函数执行.
x = np.arange(0,5,0.01)
y = (10*np.sin(5*x)+7*np.cos(4*x))
fig,ax=plt.subplots()
ax.plot(x,y)
ax.scatter(best_x,-best_y,marker='*',c="red",s=200)# 红色星号表示求的最优解
plt.show()
结果
运行一:
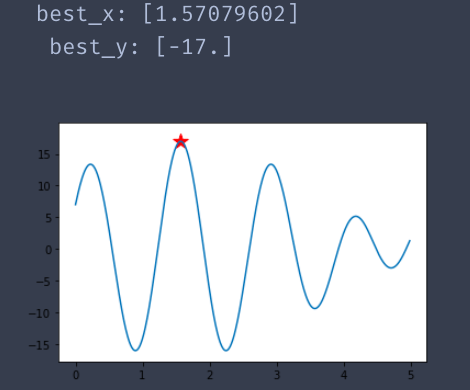
运行二:
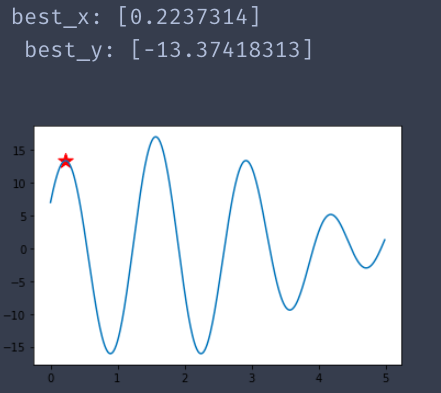
运行三:
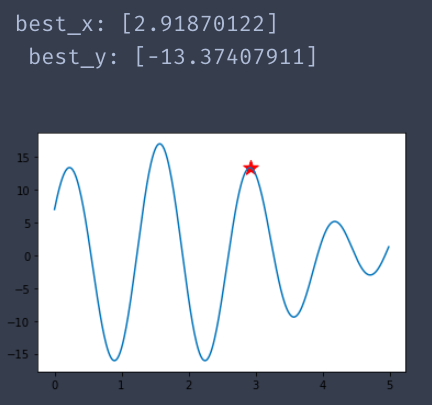
总结: 从蓝色曲线中可以看到,具有多个极值点.因此,结果有三种以上了.但是其目标函数值都是比较优秀的.切红色星星都是位于极值点处.可以说算法比较准确了.
B.使用AG解决一个单极点问题
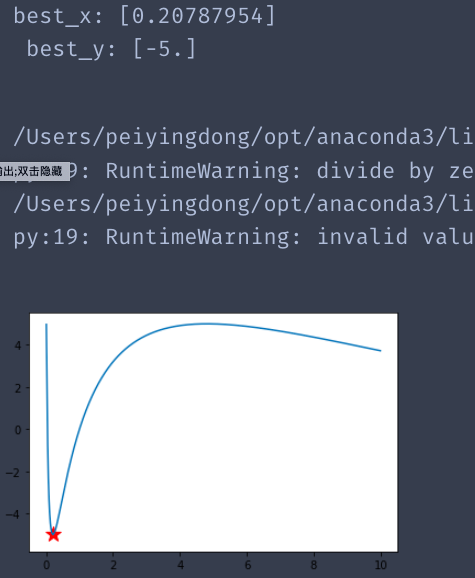
def schaffer(p):
'''
This function has plenty of local minimum, with strong shocks
global minimum at (0,0) with value 0
该函数具有大量的局部最小值,具有强烈的冲击
全局最小值为(0,0),值为0
'''
x= p[0]
return 5*np.sin(np.log(x))
from sko.GA import GA
ga = GA(func=schaffer, n_dim=1, size_pop=100, max_iter=800,
lb=0, ub=10, precision=1e-7)
best_x, best_y = ga.run()
print('best_x:', best_x, '\n', 'best_y:', best_y)
x = np.arange(0,10,0.01)
y = 5*np.sin(np.log(x))
fig,ax=plt.subplots()
ax.plot(x,y)
ax.scatter(best_x,best_y,marker='*',c="red",s=200)# 红色星号表示求的最优解
plt.show()
结果:






















 316
316











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








