点击蓝字 关注我们
Storage-D: 一个支持实用及个性化 DNA 数据存储的用户友好型平台

iMeta主页:http://www.imeta.science
方法论文
● 原文链接DOI: https://doi.org/10.1002/imt2.168
● 2024年1月21日,中国科学院深圳先进技术研究院、中国农业科学院深圳农业基因组研究所、华南理工大学等研究团队在iMeta在线联合发表了题为 “Storage-D: A user-friendly platform that enables practical and personalized DNA data storage” 的研究文章。
● 本文开发了一个名为Storage-D的DNA数据存储平台,用户可以根据自己的需求选择算法、纠错、随机访问和编解码器密码策略,实现实用和个性化的DNA数据存储,具有广泛的应用潜力。
● 第一作者:黄小罗,崔君婷,强薇,叶健文
● 通讯作者:黄小罗(huangxl@siat.ac.cn),戴俊彪(daijunbiao@caas.cn)
● 合作作者:王钰,谢鑫颖,李源真
● 主要单位:中国科学院深圳先进技术研究院、中国农业科学院深圳农业基因组研究所、华南理工大学生物科学与工程学院
亮 点
● DNA数据存储具有密度高和有效期长等显著优势,被认为是应对未来数据存储危机最有前景的介质之一;
● 本文开发了一个用户友好的平台“Storage-D”,用户可以在其中以个性化选择存储任何格式的实际数据;
● 本平台整合了一种新算法“悟空”,其中包含大量的编解码器密码,可以将数据编码到任何DNA序列中,并具有极强的隐私性;
● “悟空”算法在编码长度适合体外和体内存储的下游生化工作流程的DNA序列时,表现出比以往算法更好的整体性能;
● 该工具提供了一个开放的框架,用于集成其他编解码算法,并且可以轻松连接到商业DNA合成和测序平台,以构建一个完整的实际数据存储到DNA的流程。
摘 要
近年来,脱氧核糖核酸(DNA)被认为是一种非常有前景的数据存储介质。尽管许多研究都支持DNA数据存储,但其实际应用仍然不明确并且缺乏面向用户的平台。本研究中,我们开发了一个名为Storage-D的DNA数据存储平台,用户可以根据自己的需求选择算法、纠错、随机访问和编解码器密码策略,将他们的数据转换为任意长度的DNA序列,反之亦然。该平台采用了一种新设计的“悟空”算法,提供了超过20万亿个用于数据隐私的编解码器密码。该算法还可以控制GC含量到所选标准,并调整同源重复序列长度到规定水平,同时保持约为1.98 bis/nt的高编码潜力,使其优于以往的算法。通过“Storage-D”与商业DNA合成和测序平台连接,我们成功地将“新型冠状病毒肺炎诊疗方案”存储到了体外的200 nt寡核苷酸池,以及体内的在正常和极端细菌中均可复制的500 bp基因中。总的来说,这个平台可以实现实用和个性化的DNA数据存储,具有广泛的应用潜力。
视频解读
Bilibili:https://www.bilibili.com/video/BV1uC411r7YA/
Youtube:https://youtu.be/f50BrEBwAno
中文翻译、PPT、中/英文视频解读等扩展资料下载
请访问期刊官网:http://www.imeta.science/
全文解读
引 言
随着现代社会的发展,全球数据正在爆炸性增长,预计到2025年将达到163 ZB。传统的基于磁带或硅基存储介质的数据中心可能无法满足未来的数据存储需求,因为它们存在巨大的空间和能源消耗、高维护成本等问题。因此,迫切需要开发一种新的数据存储介质。
在不同的聚合物中,脱氧核糖核酸(DNA)作为遗传信息的天然载体,编码了生命的蓝图。近年来,DNA已经显示出其作为数据存储介质的潜力。它的密度极高,比传统的磁带或硅基存储介质高出6-7个数量级。它还具有显著的稳定性。在适当的条件下,数据可以在DNA中存档数千年。此外,通过生化复制,存储在DNA中的数据可以高保真地以相对较低的成本复制。DNA也是加密数据存储的优越候选者,并且可以在体内和体外隐藏。
尽管自20世纪60年代以来一直有人提到DNA数据存储技术,但直到Church等人和Goldman等人发表的工作之后,才开始受到广泛关注。在这些工作中,他们将基于芯片的DNA合成和下一代测序技术应用于DNA数据存储。随后,Grass等人展示了DNA封装在二氧化硅中有可能在苏黎世(9.4°C)保存数据2000年,在全球种子库(-18°C)可保存超过200万年。随后,Erlich等人证明了DNA数据存储的物理密度为每克DNA 215 PB,比以前的研究高出几个数量级。这些研究提供了关于DNA可以保留多长时间和多少信息的早期定性数据。此外,与之前的研究相比,微软的工作将超过200 MB的数据存储到制造的寡核苷酸池中,推动了DNA在大规模数据存储应用中的潜力。Koch等人还将DNA嵌入到功能材料中,可以用于制作任何形状的物体,将1.4 MB的视频存储在玻璃眼镜镜片中嵌入的DNA中,并对一小块玻璃进行了测序,将DNA数据存储与日常生活联系起来。此外,Chen等人最近的一项工作将数字文件存储在254 KB的合成染色体中,这些染色体在酵母细胞中稳定复制了100代,表明了体内DNA数据存储在大规模数据分发中的可能应用。随着越来越多的工作证明了DNA作为数据存储材料的出色性能,DNA数据存储开始接近实际应用的阶段。
主要的DNA数据存储过程包括数据编码、DNA合成和DNA测序。数据编码实现了将二进制数字信息转换为A/T/C/G序列的过程。DNA合成将A/T/C/G序列信息写入合成的DNA中。DNA测序从合成的DNA中读取序列信息。作为计算机信息和DNA序列之间的桥梁,数据编码在DNA数据存储中起着至关重要的作用。一种简单的编码方法通过将[00,01,10,11]映射到[A,T,C,G],每个核苷酸都达到了 2 bits/nt的香农极限。然而,由于从图像、文本、音频或视频中提取的二进制信息中存在大量的00、01、10或11重复,这种编码方法在编码序列中产生了许多同源重复序列和高或低GC区域,这可能会严重影响下游的DNA合成、扩增或测序,从而导致DNA数据存储失败。因此,有必要建立一种稳健的编码方法,既能满足生化实验中的约束条件,又能保持高编码潜力和数据恢复保真度。
早期由Church等人和Goldman等人提出的算法可以控制编码DNA序列中的同源重复序列,但对编码DNA序列的GC含量控制不佳,并且编码潜力相对较低。目前最先进的算法,如Erlich和Zielinski等人和Ping等人提出的算法,在控制同源重复序列和整体GC含量的同时,表现出较高的编码潜力。然而,这些“概念验证”研究并没有充分考虑实际应用环境。一个问题是它们没有考虑编码DNA序列的区域序列特性,这对于使用长DNA片段进行数据存储可能至关重要。此外,这些算法提供了固定的编码方式,并且难以适应不同的终端用户,他们可能需要定制的编码方式以考虑数据隐私。这些算法还缺乏个性化选项,并且由于缺乏有效的网络工具,其实际使用受到严重限制。这样的在线编解码平台被认为应该可以将任何数据转换为DNA序列,反之亦然,类似于“谷歌翻译”不同语言。然而,尽管这个在线编解码平台的开发已经在公开的“新闻”中报道过,但还没有进行全面的研究。例如,Song和Zeng等人基于他们的算法开发了一个非常简单的在线窗口,但这个工具的编码潜力非常有限,只能将文本转换为DNA序列。此外,这个在线窗口构建不完善,缺乏许多对DNA数据存储至关重要的专业服务的有意识的工程集成,以及提供用户友好的实际数据存储考虑的额外优化。因此,本研究的目标是开发一个对终端用户可访问且友好的编解码平台,并可用于实际和个性化的DNA数据存储应用。我们基于Web的编解码平台开发了“Storage-D”,该平台模块化了关键编解码功能,并为实际数据存储使用提供了可定制的选择。我们还开发了一种名为“悟空”的新算法,并将其集成到平台上,该算法采用了灵活的编码逻辑,并能够生成大量可用于各种DNA数据存储需求的编码规则。我们的平台或可在各种场景中大大促进DNA数据存储研究和应用。
结 果
Storage-D的界面和个性化选择
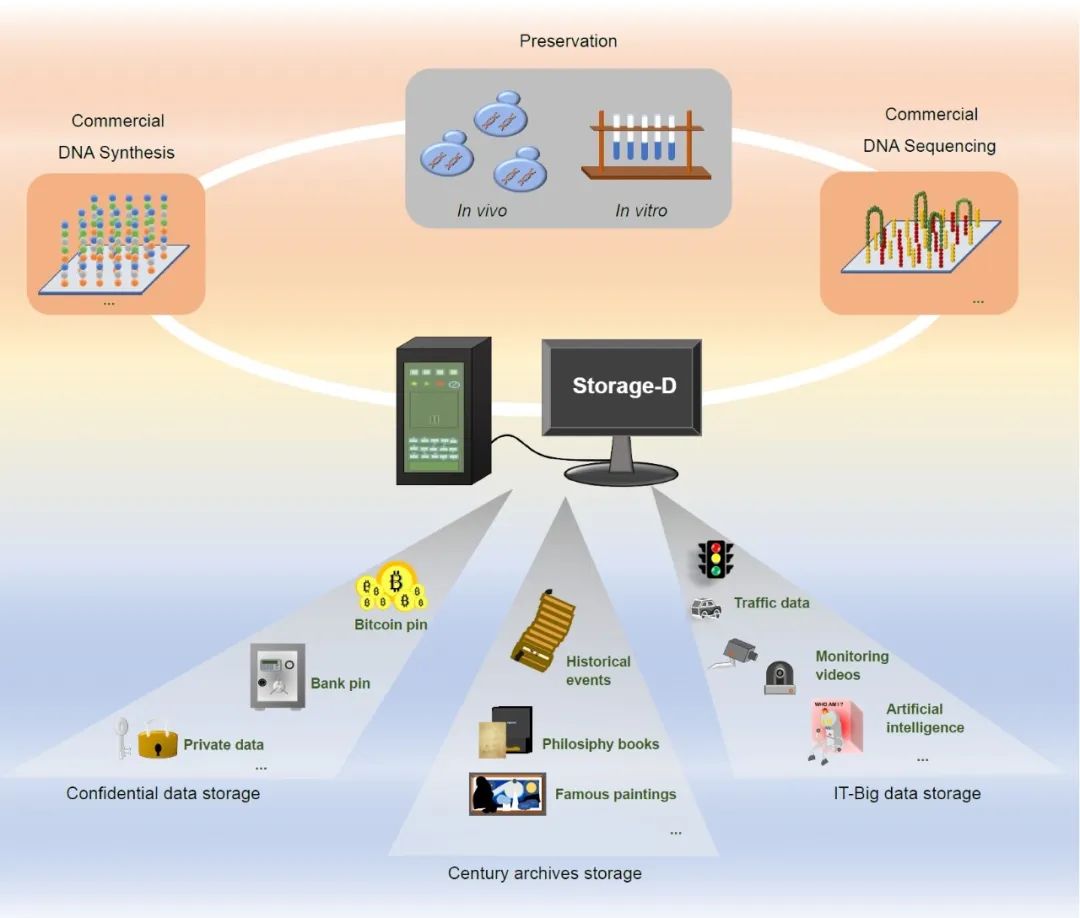
Storage-D在线工具包含数据编码和解码功能,每个功能都有其页面。页面分为输入模块和输出模块。输入模块包括“文件上传”和“预览”,以及“参数选择”,输出模块包括结果预览和与编解码过程相关的重要信息(图1)。

图1. Storage-D的工作流程和界面
(A) Storage-D的编解码器工作流程。Storage-D软件支持四种常见的文本、图像、音频和视频文件作为输入,并为用户提供“悟空”、“Church et al.”、“Goldman et al.”、“DNA Fountain”和“Yin–Yang”编码算法供选择。至于编码参数,用户可以自定义编码的DNA长度、GC比率、同源重复序列长度、纠错、冗余、前后序列和编解码器密码。(B) Storage-D的界面。这表示将“COVID-19病毒RNA聚合酶结构”的图像转换为DNA序列。
对于数据编码,可以使用不同的图像、文本、音频和视频格式作为输入,并从个人电脑和移动设备的本地文件中上传。上传的数据在“预览”窗口中可视化显示,编码的DNA序列以FASTA格式显示在输出窗口中。与进一步解码相关的内部参数记录在第一行。当提供明确的电子邮箱地址时,输出数据也可以邮件发送给用户。在解码模式下,与编码输出相同的FASTA格式的DNA序列(例如,从DNA测序获得)可以转换为所需的图像、文本、音频和视频数据,然后下载。值得注意的是,为了避免服务器拥堵,上传的数据大小目前限制为20 MB。对于较大的文件,用户可以在GitHub上下载Storage-D程序,并在本地进行数据编解码。
与“悟空”算法相关的个性化编解码器特征,包括“编码长度”、“单碱基重复序列”、“最小GC%”、“最大GC%”、“编解码器密码”、“ECC(RS)”、“侧翼序列”和“冗余”,可以被用户在Storage-D中选择。“编码长度”指的是用于编码计算数据的DNA串的长度。它与用户用于合成编码序列的DNA合成平台相关。如果用户想使用高通量寡核苷酸合成平台合成这些序列,则推荐使用默认DNA长度,200个核苷酸。“单碱基重复序列”指的是编码DNA序列中的最大单碱基重复序列长度,不包括侧翼序列。“最小GC%”和“最大GC%”指的是编码DNA序列的GC含量上下限,但不包括侧翼序列。“ECC(RS)”指的是错误纠正选择,“冗余”指的是是否在编码DNA序列中添加三分之一的数据冗余。为了提高数据保真度,用户可以在数据编码过程中选择这两个参数。为了随机访问存储的数据,用户可以使用Storage-D设计他们的随机访问侧翼序列。建议用户应将侧翼序列设计为20-25个核苷酸的长度。值得注意的是,Storage-D中实现的流程专门用于设计面向寡核苷酸池或基因池的随机访问侧翼序列,这是目前最有前景的大规模数据存储的DNA介质。此外,“悟空”还为用户提供了多个个性化编解码器密码的选项。每个编解码器密码包含来自“悟空”编解码器库的两个编解码器规则。Storage-D目前集成了30,000个编解码器密码供用户选择。这些是用于存储其DNA数据的个性化密码,将大大提高其数据的安全性。此外,该工具可以将DNA序列文件发送给用户或直接发送给商业公司进行DNA合成。
此外,用户可以选择先前建立的算法,包括Church等人、Goldman等人、Erlich和Zielinski,以及Ping等人提出的算法来编码和解码他们的数据。值得注意的是,尽管一些编解码器特性在它们的原版中并不存在,如表1所示,但个性化设计,包括“编码长度”、“单碱基重复序列”、“最小GC%”、“最大GC%”、“编解码器密码”、“ECC(RS)”、“侧翼序列”和“冗余”,如果可能与算法原理匹配,将被添加到与这些算法绑定的Storage-D界面中,为这些算法提供更灵活的编解码器选择。通过Storage-D,用户可以轻松选择任何一种算法进行数据编解码。
表1: “悟空”与其他编解码算法的比较*

–表示该功能设计在算法中不可用。
a Church等人、Goldman等人、Erlich和Zielinski,以及Ping等人提出的算法的详细特征在表中保持了它们原版的特征。使用Ping等人描述的公式计算了“悟空”算法的编码潜力。其余的编码潜力引用自Ping等人。
b Ping等人的算法在他们的原版中提供了1536个编解码器规则,这些规则可能在未来被用作编解码器密码。
在Storage-D中实现的“悟空”编解码算法
编码潜力是DNA数据存储中编解码算法的一个重要属性,因为它直接关系到特定数量的DNA可以保留的数据量以及最终的DNA数据存储成本。与其他已发表的二进制编解码算法相比,“悟空”算法目前具有最高的编码潜力,为1.98比特/核苷酸,与Erlich等人提出的“DNA喷泉”算法相同,略高于Ping等人提出的“阴阳”算法(表1)。此外,尽管具有相同的编码潜力,“悟空”在数据恢复方面表现优于“DNA喷泉码”(图S1)。同时,“悟空”在编码不同长度的DNA时也显示出比“阴阳”编解码算法更高的实际编码密度,并且以显著更快的速度编码了等效长度的DNA(图2C-D)。

图2. 使用Storage-D中实现的算法对不同长度DNA序列进行编码
通过Church等人的算法(生化约束:同聚物≤5nt;用橙红色表示),Goldman等人 (用金色表示),Erlich和Zielinski (生化约束:同聚物≤5nt,GC范围为0.4至0.6;用绿松石色表示),Ping等人 (生化约束:同聚物≤5nt,GC范围为0.4至0.6;用天蓝色表示)和“悟空”(生化约束:同聚物≤5nt,GC范围为0.4至0.6;用深蓝色表示)的算法将86,869字节的图像编码为1000 nt的DNA序列字符串,而在编码过程中未考虑纠错、冗余和侧翼序列。从编码的1000 bp DNA序列中,以140 bp的步长提取了150 bp的窗口,用于分析其GC含量和同源重复序列长度。(A)不同编解码算法编码的DNA序列的局部GC分布。计算并绘制了落入给定GC含量的150bp序列窗口的数量。特定部分内的图形区域显示该区域内的频率。面积越大,分布的频率越高。白色点表示数据的中位数。(B)不同编解码算法编码的DNA序列的区域同源重复序列分布。计算并绘制了落入给定同源重复序列长度的150bp序列窗口的数量。黑色点表示异常值。(C)通过“悟空”和其他算法将数据转换为100至800nt的DNA序列的实际编码密度。密度定义为编码数据量除以总碱基数。(D)每个算法将数据编码为不同长度的DNA序列的运行时间。
长单核苷酸同源重复序列通常会导致DNA合成和测序过程中的错误,这些错误被认为会影响存储在DNA中的数据的解码。以前的方法通常将其限制在预定的水平上。在某些情况下,这可能不合适,例如Nanopore测序,它更倾向少于4个核苷酸的同源重复序列。由于其灵活的编码方式,“悟空”能够在不同的核苷酸下调整同源重复序列的长度,例如3、4或5个核苷酸(图S2)。这可以使其能够在各种场景中使用。
局部GC含量对DNA数据存储中的随机访问和DNA测序步骤的PCR扩增有相当大的影响。局部GC含量高或低通常不适用于长DNA片段合成,且局部GC含量高或低和长同源重复序列对寡核苷酸的基因组装有特殊影响。先前的算法包括“DNA喷泉”和“阴阳”算法只考虑在整个DNA水平上控制GC浓度。这可能导致局部序列水平上的显著GC偏差(表1,图2A)。值得注意的是,“悟空”引入了一种特殊的设计来检查150 nt窗口内的GC含量和同源重复序列,使得“悟空”能够在没有区域生化限制的情况下设计长基因(图2B)。特别是,根据“悟空”算法在将二进制信息转换为DNA序列时的灵活逻辑,它能编码落入任何理想GC标准范围内的DNA序列,从相对宽泛的GC范围35-65%到相对严格的GC范围45-55%(图S2)。这使得“悟空”能够与不同的DNA合成和测序方法相匹配,尽管它们的序列限制不同。此外,“悟空”可以编码具有与生物体基因组GC含量相对应的DNA(表S1)。这能使“悟空”适用于体内DNA数据存储。
值得注意的是,“悟空”提供了灵活的数据编解码逻辑,使其能够快速生成超过20万亿个编解码规则。这为用户提供了广泛的编码选择。以往的工作在为DNA数据存储生成适当的编码方法方面存在困难,而这个编解码库提供了前所未有的大量编码方法,这会大大减轻数据编解码的工作量。虽然每个编码过程都可以利用同一对编解码规则进行数据编解码,但这对于数据加密尤为重要。例如,不同的用户可以使用不同的编解码密码来编码他们的数据。或者,不同的编解码密码也可以用作标记,以区分存储在DNA中的不同类型的数据。鉴于“悟空”算法庞大的编解码库,它或可广泛用于各种定制的DNA数据存储场景中。
综合来看,“悟空”在整体性能方面优于其他编解码算法,使其在实际数据存储中比其他算法更加稳健。
Storage-D 的计算机评估
为了展示Storage-D的实用性,我们首先评估了其在编码各种类型数据方面的性能。如今,计算机数据主要包括程序、文本、音频文件、照片和视频等类型的文件。Storage-D可以在编码过程中将这些计算机文件的任何格式转换为二进制信息,反之亦然。为了测试其运行情况,我们将不同大小的数据(从约3 KB到7.7 MB)实施到Storage-D中。如表S2所示,用户不仅可以选择“悟空”算法,还可以选择Church等人、Goldman等人、Erlich和Zielinski等人和Ping等人提出的算法来编码他们的数据,并相应地解码他们的数据。此外,Storage-D利用其开发的“悟空”算法可以在1分钟内以相对恒定的密度和相对较快的速度对模拟数据进行编码(从1 KB到104 KB)(图S3)。对于数据大小从1 KB到104 KB的数据解码也可以在不到1分钟内完成,并且具有很高的准确性(图S3)。此外,编码的DNA片段具有相当相似的GC表现,其中大多数收敛于约50%的GC含量(图S3)。总体而言,Storage-D为DNA数据存储提供了一个高效的数据编解码平台。
鉴于当前商业DNA合成提供了不同长度的DNA片段(例如,基于柱状寡核苷酸合成的约120 nt,基于芯片寡核苷酸合成的约300 nt,以及组装基因从约200 bp到10 KB不等),作为第二个例子,我们测试了“Storage-D”在编码不同长度的DNA方面的能力。如图2D所示,通过Storage-D,用户可以根据其下游DNA合成需求改变DNA的编码长度。
此外,作为Storage-D的独特功能,我们通过各种编解码器密码测试了其数据编码。Storage-D目前集成了30,000个编解码器密码,这些密码与“悟空”编解码器规则库配对,存在显著差异。用户可以通过选择不同的编解码器密码将不同的数据编码为不同的DNA序列,或者将相同的数据编码为不同的DNA序列(图3A)。尽管只计算给定位置上每个核苷酸的总计数,但当数据转换为DNA序列时,不同的编解码器密码显示出明显的编码差异(图S4)。这与各种编解码器密码编码的序列之间的汉明距离显著不同的发现一致(图S5)。当不同的编解码器密码编码不同的DNA序列,使用错误的编解码器规则解码序列是不可能的(图3B)。这为用户提供了一种加密数据的可用方法。
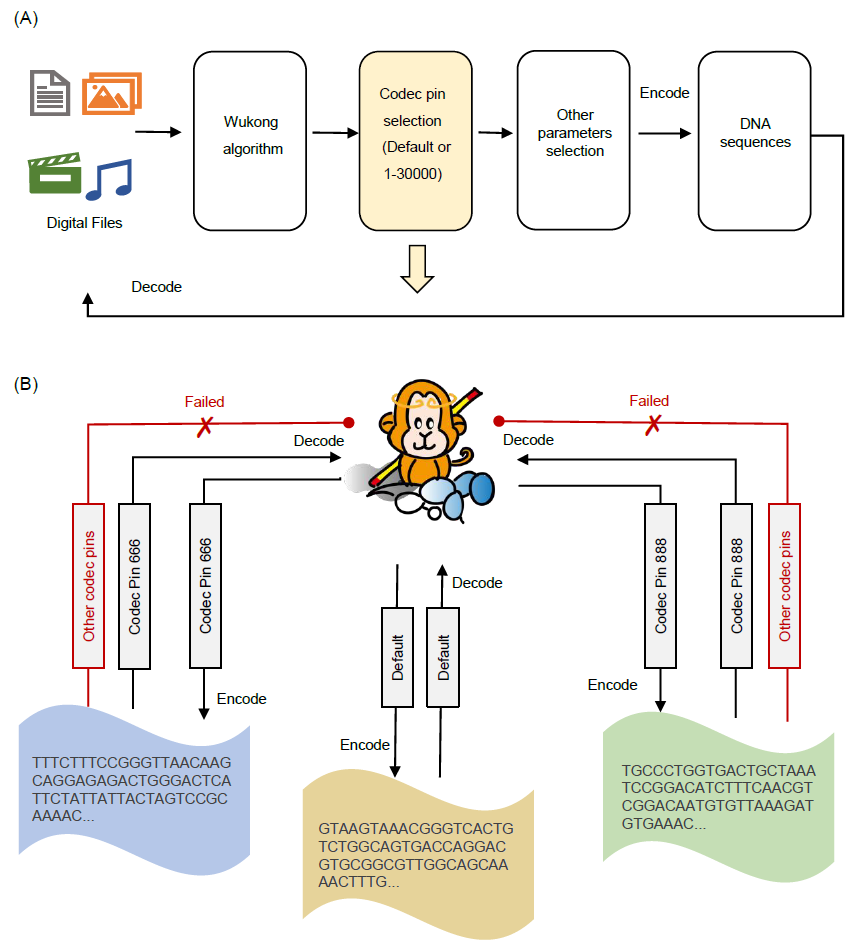
图3. 使用不同的编解码器密码对数据进行编码
(A) 将不同编解码器密码用于将数据编码为DNA的工作流程图。(B) 基于专用密码的数据编解码器的示例。一个卡通图像显示,中国传统神话中的主要角色“孙悟空”通过默认选择或编解码器密码666和编解码器密码888被编码为DNA序列。对于相同的输入文件,如果用户选择不同的编解码器密码,将获得不同的编码序列。在解码时,如果输入错误的编解码器密码,将无法获得正确的结果。
通过实际数据存储验证“Storage-D”
鉴于DNA有潜力存储数据数千年,我们打算将过去三年中抗击COVID-19病毒的宝贵经验存储在DNA中,以便将这些经验传承给我们的后代。因此选择了“悟空”算法用来将中国国家卫生健康委员会的《新型冠状病毒肺炎诊疗方案(试行第7、8、9版)》(分别包含333,360位、462,480位和416,280位)以及一本经典的中医书籍《伤寒杂病论》(575,040位)编码为1909、2669、2402和3319个DNA链。每个链包含200个碱基对,经过生化约束条件“GC = 40−60%”和“同源聚合物长度≤4个核苷酸”筛选。每个二进制字符串都添加了两个字节的RS纠错码,并且通过OR(XOR)转换生成的数据冗余的三分之一被添加到总的二进制数据中,以提高数据恢复的准确性。从记录数据的二进制位中每隔两个二进制字符串被编码成一个单独的DNA字符串,然后进行筛选。为了随机访问数据,一个精心设计的有20个核苷酸的侧翼序列被添加到每个字符串的两端(图S6A)。最终编码的DNA序列被合成为一个寡核苷酸池(表S3)。
通过随机访问引物(表S4)对每个数据文件进行PCR扩增,在琼脂糖凝胶中产生了明显的条带,表明每个数据文件可以成功访问(图S7A)。测序结果显示,每个文件的测序深度非常相似,表明我们的文库构建具有高度的同质性。所有数据文件的平均测序深度为387x,如表S5所示。不幸的是,三个寡核苷酸完全缺失于测序数据中(图S7B),这可能归因于DNA合成、DNA测序或PCR扩增的问题。然而,借助RS编码和每个DNA字符串中添加的冗余信息,当测序深度超过30x时均能够实现100%的数据恢复率。此外,当测序深度降低到约10x时,实现了超过99%的数据恢复率(图4B)。这个结果证明了“悟空”编解码算法在低测序深度下的数据恢复鲁棒性。
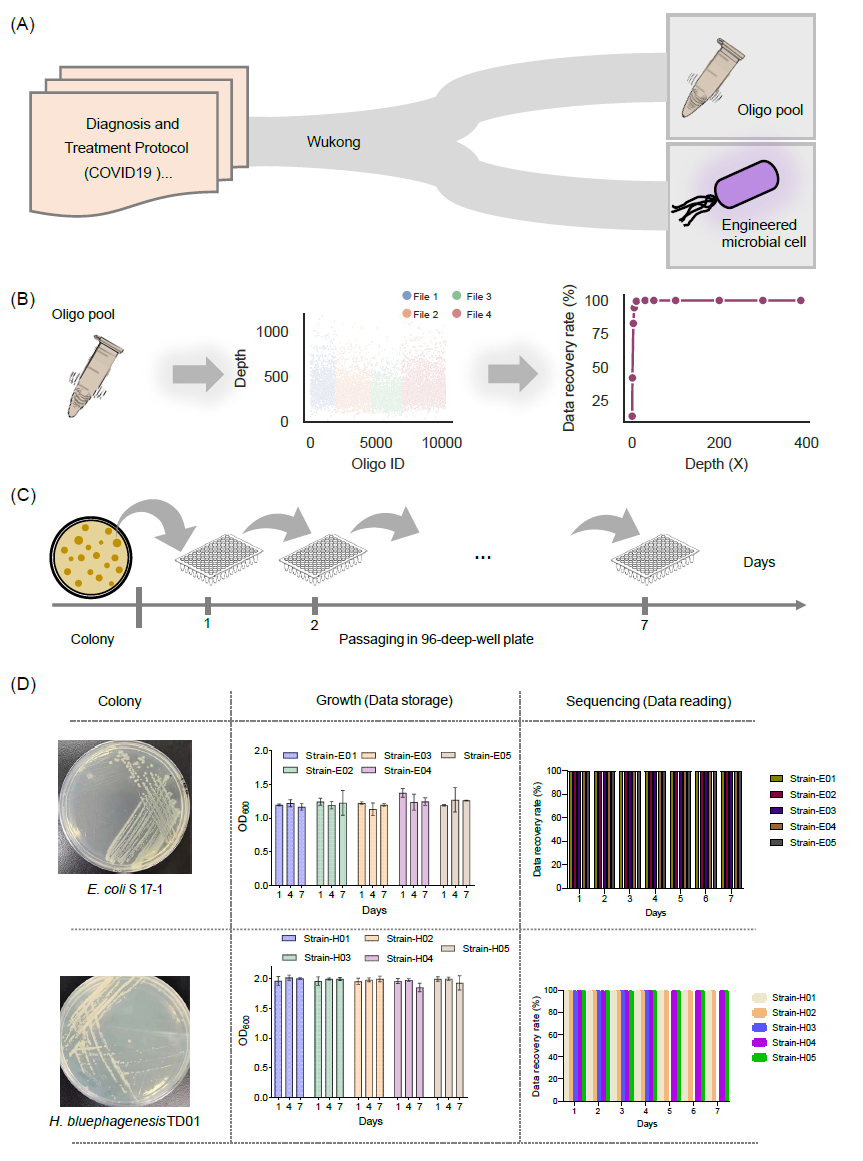
图4 “悟空”算法在体外和体内的验证
(A) “悟空”算法在体外和体内存储数据的示意图。我们通过寡核苷酸池合成了从《新型冠状病毒肺炎诊疗方案》第七、第八和第九版以及古代中医著作《伤寒杂病论》中编码的序列,用于体外存储。此外,我们还将一些用于治疗COVID-19的传统中药方剂编码并合成到细胞中的基因中;(B) 通过测序读取寡核苷酸池。所有寡核苷酸的测序深度显示在中间,数据恢复率显示在右侧。(C) 体内数据传递的示意图。(D) 数据存储在大肠杆菌S17-1和噬盐单胞菌TD01菌株中,并稳定复制了7天。通过测序获得的生长状态和数据恢复率如图所示。
由于前面的测试是基于一个在体外保存的200 bp合成寡核苷酸池,我们还尝试使用“Storage-D”将数据存储到组装基因中,并在体内保存。作为体内DNA数据存储的两个示例宿主,我们分别选择了一种典型的细菌大肠杆菌和一种极端细菌噬盐单胞菌作为宿主。上述体外存储的一些数据通过“悟空”算法(图S6B)编码成500 bp基因序列(表S6),遵循生化约束条件,即“单碱基重复序列长度≤4 nt”和“GC含量在物种编码DNA序列(CDSs)的中位数GC值的±10%范围内”(表S1)。这些基因序列被合成并进一步转化到每个菌株中。如图4D所示,含有存储信息的细菌菌株显示出稳定的生长,并且在生长7天内以100%的准确率解密了信息。通过“悟空”算法在大肠杆菌和噬盐单胞菌中的数据存储证明了数据可以在正常和极端细菌中存储。
总的来说,将《新型冠状病毒肺炎诊疗方案试行版》和《伤寒杂病论》存储到合成DNA中,并以高精度解码,这表明“Storage-D”在实际数据存储方面具有实用性。
讨 论
Storage-D的开发主要是为用户提供一个用于研究DNA数据存储的快速、简化和可行的编解码平台。实现二进制信息与DNA序列之间的高效转换,可以帮助研究人员优化与DNA数据存储应用相关的生化工作流程,包括随机访问PCR扩增、体外或体内保存DNA存储信息以及更高效的DNA合成和DNA测序流程。目前,Storage-D中除了专门设计的“悟空”算法以外,还有Church等人、Goldman等人、Erlich和Zielinski、以及Ping等人提出的算法。此外,Storage-D还设计了可变参数选择,包括“编码DNA长度”、“GC含量”或“单碱基重复序列长度”、“纠错”、“侧翼序列”、“冗余”和“编解码密码”。这些功能使用户能够根据个人兴趣对数据进行编码。虽然整合其他优秀算法也是值得的,但Storage-D定义了一种通用的输入和输出风格,并提供了一个易于使用的框架,以便将来整合更强大的算法。一些算法,尽管它们可能不是通用算法或者与当前算法过于相似,但可以包含在我们的平台中,为使用者提供额外的选择。此外,如果完成更多测试,跳过二进制转换步骤的创新算法(目前处于概念验证阶段)也可以实现到平台上。还可以将针对各种数据文件格式或特定类型数据的更个性化的考虑纳入平台。此外,未来的平台还可以测试和实施无损压缩和有损压缩等数据压缩算法。总体而言,Storage-D是DNA数据存储中编解码的强大工具。
Storage-D采用了一种特别设计的算法,“悟空”。“悟空”的概念源于我们寻求在DNA数据存储的背景下,利用不同的加密密钥(或称为“密码”)对单个数据元素进行加密的简单直观方法。尽管多年来已经开发了用于数据加密的计算程序,但没有一个程序被设计用于在二进制信息和A/T/C/G序列之间的转换过程中创建防火墙。这导致了关于DNA数据存储的数据安全性方面的差距。在本研究中,我们通过将4个二进制位映射到2个核苷酸以及排列组合,创建了一个包含超过20万亿个DNA数据存储编解码规则的池。随后,我们开发了一种能够在编码过程中选择编解码“密码”的算法。这种方法可以有效地填补关于DNA数据存储的安全漏洞。事实上,“悟空”可以通过将6个二进制位映射到3个核苷酸、8个二进制位映射到4个核苷酸等进一步扩展。如果未来研究需要更大的编解码规则库进行数据加密,这将创建一个更大的编解码规则库。此外,可以根据“悟空”算法的流程将3、4、5个或更多的编解码规则组合和集成到编解码过程中。这种方法通过编解码规则的组合增强了数据加密的可行性,从而显著降低了敏感信息意外泄露的风险。此外,“悟空”在不牺牲编码潜力的情况下,使用户能够以理想的GC比率和短同源重复序列长度对序列进行编码。具体而言,“悟空”可以在150 bp窗口内以均匀的GC比率和短同源重复序列对DNA进行编码,这可以使其适用于DNA数据存储中使用的更多生化技术,包括DNA合成、PCR扩增和DNA测序。
作为无缝集成最先进算法的编解码平台,其与商业DNA合成和DNA测序平台的集成有助于简化建立全面的DNA数据存储流程。因此,Storage-D可以被研究人员有效地用于将信息存储到体内或体外的DNA中。基于Storage-D的优势,我们推测它在数据存储应用(如图5所示)方面是可行的,例如世纪档案存储。在理想情况下,DNA已被提出作为一种能够保存信息的介质,最长可达200万年,这个时间超过了迄今为止任何其他存储介质。人类文明的集体面貌,包括文化、历史事件和技术进步,需要跨越代际传递,可以通过使用Storage-D平台在DNA中进行准确的存档(表S7)。此外,该平台同样适用于安全存储过去几个世纪的数据,包括个人兴趣,是高度机密数据存储的优秀解决方案。通过使用Storage-D,数据可以以合成寡核苷酸的形式体外存储,也可以以细菌、植物或动物细胞的形式体内存储。此外,编解码密码还在二进制信息和A/T/C/G序列之间建立了一个机密防火墙。因此,比特币密码、银行卡密码等机密数据可以安全存储。此外,Storage-D还有潜力在未来存储由互联网、人工智能或摄像头创建的大数据(特别是冷数据),因为DNA的密度比目前最常用的介质(如硬盘和磁带)大6个数量级以上。大数据可以分割成小规模的数据,例如20 MB,并使用Storage-D平台进行进一步的DNA存储处理。未来的发展方向还可以建立一个PB级别的数据存储框架,可能需要进行彻底的索引设计。综上所述,Storage-D在未来的DNA数据存储中将发挥重要作用。

图5. Storage-D引导的数据存储:应用展望
将商业DNA合成、体内和体外保存以及商业DNA测序与Storage-D集成,可以开发出一套全面的DNA信息存储应用系统。这个先进的系统可以用于安全数据存储、长期存档目的以及IT大数据的高效管理。
然而,由于高昂的DNA合成成本,当前DNA数据存储的成本相对较高,这阻碍了其当前的应用。根据现有商业DNA合成速率,存储1 MB数据的合成寡核苷酸池的成本超过1500美元。这种成本结构在一定程度上限制了Storage-D的广泛应用,使其主要适用于对价格敏感性不太重要的应用。然而,由于超高通量DNA合成技术和基于酶的DNA合成技术的发展,DNA合成价格一直在下降。为了降低DNA数据存储的成本,现有的基于芯片的DNA合成商业供应商,如Twist Bioscience和CustomArray,正在努力提高给定芯片大小下的DNA合成通量。预计如果一个DNA合成芯片的通量从目前的百万寡核苷酸水平增加到约1014个寡核苷酸水平(一个手掌大小的芯片(约10 cm x 10 cm),具有约10 nm缩放的合成点,类似于当前的电子芯片),并通过持续优化合成反应,DNA合成价格将下降6-8个数量级以上,DNA数据存储成本可能开始接近当前硬盘成本。同样,如果基于酶的DNA合成的价格与一个细菌细胞中的基因组复制价格相一致(在1 L LB培养基中过夜培养可以以约1美元的成本产生约109个大肠杆菌细胞,每个细胞具有约4 MB的基因组),DNA数据存储成本甚至可能低于标准硬盘。因此,我们推测未来应该在更广泛的数据存储领域使用Storage-D。
结 论
总之,我们开发了一个强大且用户友好的DNA数据存储编解码平台——Storage-D。通过Storage-D,用户可以使用多种经过验证的算法和个性化参数,对不同格式的数据进行编码和解码。特别是它采用了一种新开发的编解码算法,名为“悟空”,其中包含了大量的编解码规则,使用户能够通过唯一的编解码密码保护他们存储的数据。根据我们的研究结果,我们相信Storage-D将大大有益于DNA数据存储的使用。
方 法
Storage-D的整体框架
Storage-D的开发旨在提供一个用户友好的平台,用于计算数据和A/T/C/G序列之间的“翻译”。它将DNA数据存储的基本功能模块化,包括数据编解码、纠错和随机访问,并具有多个可选设置。为了将数据转换为DNA序列,开发了一种名为“悟空”的新型编解码算法。它能够以可控的单碱基重复序列长度和区域均匀的GC含量编码不同长度的DNA,同时保持高编码潜力,并提供独特的编解码密码库。在此基础上,通过集成“XOR”冗余和纠错码实现了纠错功能。最后,使用特别设计的“侧翼序列”生成过程实现了随机访问功能。此外,还建立了一个用于解码存储的DNA数据的测序分析流程,并将其附加到该平台上。
除了这些,先前建立的算法,包括Church等人、Goldman等人、Erlich和Zielinski,以及Ping等人提出的算法,也被整合到该平台中。这些算法的基本逻辑已经发布,并且统一了输入和输出风格以适应平台的整体框架。如果可能与算法匹配,个性化设计会被添加到与这些算法绑定的Storage-D在线界面中。使用Storage-D,用户可以轻松选择任何数据编解码器的算法。
“悟空”编解码算法
首先构建了“悟空”编解码系统的编解码规则库。通过四个二进制位和两个核苷酸之间的编解码映射关系的排列组合(A_16^16),生成了一个包含20,922,789,888,000个编解码规则库 (图S8)。然后,从该库中随机选择两个编码规则来编码数据信息。无论这些二进制位来自单个二进制数据字符串还是多个不同的二进制数据字符串,都被视为一个编码单元。在第(2n-1)个编码单元中使用一个编码规则,在第2n个编码单元中使用另一个编码规则。值得注意的是,通过这种方式,可以将两个二进制片段、三个二进制片段和四个二进制片段编码成一个DNA序列字符串,从而实现了二进制数据和DNA序列之间的相对自由转换(图S9)。
为了通过“悟空”对数据进行编码,首先将计算机文件转换为二进制位,然后将其切割成所需长度的片段。随机选择二进制片段,并使用从“悟空”编解码器库中选择的两个规则进行编码。如果编码后的DNA序列满足预设的GC和同源多聚物运行,则从原始二进制片段池中删除所选的二进制片段。未能生成理想序列的所选二进制片段被放回原始池中(图6)。偶尔,在30次重复以下时所选的二进制片段无法生成理想序列,因此添加一个虚拟片段以产生理想序列。最终,设计出具有理想序列特性的DNA序列。

图6. “悟空”编解码算法原理
“悟空”的编码过程。计算数据首先被转换为二进制信息,然后被分割成所需的长度(Lstring=N,“N”表示任意数字)。两个或更多的二进制片段经过随机配对处理并合并成一个二进制片段,然后通过两个选定的编解码规则进一步转码为DNA序列。随后,选择具有所需特性(GC,单碱基重复序列等)的DNA序列进行进一步的DNA合成。不符合期望标准的序列被解码回原始二进制片段池中,进行新一轮的编码过程。
纠错设计
DNA合成、聚合酶链反应(PCR)扩增和DNA测序在DNA数据存储过程中会产生错误。尽管PCR扩增的错误率很低(通常低于1/100,000),但商业化学寡核苷酸合成的错误率高达1/2000至1/200,而DNA测序错误率可达到超过10%。尽管深度测序可以生成冗余读取的折叠以进行错误纠正,但在组装的DNA序列中仍然存在一些错误,需要进行数据解码。因此,如果需要100%的数据恢复,则必须进行错误纠正。
处理存储数据中的错误的一种典型方法是添加纠错码。虽然不同的纠错码,如汉明码、Reed-Solomon(RS)码和低密度奇偶校验(LDPC)码已经被整合到DNA数据存储中,但目前最广泛使用的是RS码。本研究中,我们在Storage-D中集成了RS码进行纠错设计。我们允许用户在使用“悟空”算法时添加n个字节的RS码(n≥1)。如果用户想要添加RS码,提供的RS码字节将在从原始数据编码的每个二进制字符串的索引和冗余(如果用户要求)之后插入。我们引入了一个Python模块“reedsolo”,经过Erlich和Zielinski,以及Ping等人的验证,用于计算和创建RS码,以及进行解码的纠错和验证。这些RS码可以检测多达n个字节的错误并纠正多达n/2个字节的错误。值得注意的是,这里使用的纠错符号是“1个字节”,如果以DNA编码,相当于“4个核苷酸”。这意味着一个n个字节的RS码可以检测多达4 n个碱基错误并修复其中多达2n个。对于其他集成算法,除了Goldman等人提出的算法外,无论RS码是否被整合到他们的原版中,这个RS码也被附加到他们的编码过程中。因此,用户可以在使用Storage-D时添加RS码的字节。
有些时候在DNA合成、PCR扩增或DNA测序过程中可能会发生序列丢失或部分序列丢失。因此,尽管DNA测序生成的纠错码和冗余读取可以修正生化实验中发生的几个错误,但完全的数据恢复仍然具有挑战性。引入信息冗余可以解决这个问题,并实现高保真度的DNA数据存储。我们在“悟空”算法中引入了冗余设计。两个连续片段进行XOR转换以创建一个新片段,从而在总数据中生成三分之一的冗余(图S10)。这些可以帮助用户在生化实验中发生相当大的损失时以高保真度解码他们的数据。我们还对Church等人和Ping等人描述的算法进行了相同的操作。然而,鉴于Goldman等人通过编码重叠的DNA序列引入了四倍的冗余,并且Erlich和Zielinski使用“DNA喷泉”代码在编码序列中生成一定程度的冗余,我们在Storage-D中没有为这些在整个编码过程中有自己创建冗余的逻辑的方法添加额外的冗余。
随机存取设计
在编解码过程中,还应考虑数据的随机访问。尽管几种随机访问方法已经被提出,但最简单的方法是在编码的DNA序列的配对末端添加侧翼序列,然后通过PCR进一步扩增。2018年,Organick等人在合成的寡核苷酸池中存储了35个不同文件,其中包含超过200 MB的数据,并成功地使用基于PCR的随机访问方法恢复了一个单独的文件。为了随机访问数据,设计了可以通过PCR扩增的侧翼序列,具体步骤如下。首先,使用功能强大的寡核苷酸设计工具Primer3程序创建了一个以生成的随机序列为模板的侧翼序列池。我们将设计的侧翼序列限制为GC含量为40%-60%,TM值为58°C-70°C。为了避免随机访问引物对编码DNA序列可能的不特异性扩增,对侧翼序列池进行了筛选。删除了那些在3'端具有与编码DNA序列或反向互补编码DNA序列相同的6个核苷酸的侧翼序列。为了进一步避免随机访问引物与不同侧翼序列之间的交叉配对,剩余的侧翼序列通过BLAST进行同源分析,从中选择得分最低的一对侧翼序列,并将其添加到编码序列的配对末端(图S11)。该过程的程序可以与Storage-D中实现的任何算法结合使用。值得注意的是,由于Primer3程序设计引物池的速度相对较慢,因此在Storage-D中实现了一个预设计的包含约20,000个序列的池。这些序列也通过同源分析和汉明距离计算相互交叉检查,确保它们适用于下游工作流程。
编码DNA长度设计
目前的DNA合成技术通常产生10 KB以下的DNA片段。对于商业DNA合成,不同长度的DNA通常有不同的价格。因此,为了给用户提供设计不同长度DNA片段的选择,Storage-D采用了专门设计的流程来实现这一目标。DNA长度定义为Church等人、Ping等人提出的算法和“悟空”算法的数据有效载荷区、索引区、纠错区和随机访问适配器的长度之和。Erlich和Zielinski提出的算法将DNA长度定义为数据有效载荷区、种子区、纠错码区和随机访问的侧翼序列的长度之和。对于Goldman等人提出的算法,DNA长度是根据算法的内部逻辑来设计的(详见补充文本)。虽然索引和纠错码的长度不同,但最终编码的DNA长度等于或接近用户输入Storage-D的长度。
通过实际数据存储验证Storage-D平台
Storage-D平台在体外和体内都进行了验证。对于体外存储,使用“悟空”算法将四个包含1,787,160位的数据文件编码为10,299个寡核苷酸序列。编码的DNA序列在高通量寡核苷酸合成平台(Twist Bioscience,美国)上合成。一瓶冻干的寡核苷酸池(124 ng)以常温从美国旧金山邮寄到中国深圳。为了扩增合成的寡核苷酸池,使用基于柱的寡核苷酸合成平台在清华(中国)合成了与每个DNA字符串的侧翼序列相对应的随机访问引物(表S4),并使用寡核苷酸纯化柱(OPC)进行纯化。
为了模拟真实的DNA数据存储使用,首先将合成的寡核苷酸池稀释至108个分子/μl。为了获取信息,每个文件通过与每个文件的随机访问侧翼序列相对应的指定引物从寡核苷酸池中扩增。PCR混合物和程序被设置进行,并且在琼脂糖凝胶电泳中看到清晰的条带。使用Q5高保真DNA聚合酶(NEB)在总反应体积为50 μl的条件下,以以下条件扩增约2 μl稀释的寡核苷酸池:98℃,25秒;65℃,5秒,共25个循环。然后,根据其寡核苷酸数量的比例混合每个文件的PCR产物,并在Illumina MiSeq平台上进行测序(详见补充文本)。
对于扩增PCR产物的测序数据分析,首先使用PEAR v0.9.11进行配对末端读取的组装。未组装和低质量的读取被丢弃。然后,根据随机访问的侧翼序列选择和过滤组装序列。接下来,记录最后一步输出测序读取的数量和碱基质量。将具有低于Q30质量值的80%以上碱基的读取消除。然后检查所有读取的长度,并保留具有相同长度且计数最高的读取。随后,首先按质量排序,然后按数量排序。每个序列根据其顺序号获得一个分数。使用BLAST分析对所有序列进行聚类,并选择每个聚类中顺序得分最低的序列作为输出(图S12)。然后删除序列池的侧翼序列区域,并根据用于编码数据的特定“悟空”编解码规则解码DNA序列。然后应用RS编解码来纠正替代错误。通过索引区域过滤和重新排序二进制片段后,剩余的错误通过编码过程引入的冗余的三分之一进行纠正。接下来,将二进制信息转换为原始数据文件。
为了进行体内储存,部分数据文件(来自体外数据存储)的4560位被编码成每个基因有500个碱基对(bp),并在北京六合BGI合成。然后,合成的基因在克隆到广宿主pSEVA框架载体后(详见补充文本)被转化并复制到大肠杆菌S17-1和噬盐单胞菌 TD01菌株中。为了检查稳定性,存储的数据菌株连续转移到96孔板中。噬盐单胞菌菌株在LB补充培养基中培养,添加5 g/L无水醋酸钠、50 g/L氯化钠和25 mg/L氯霉素,而E. coli菌株在含有10 g/L葡萄糖和25 mg/L氯霉素的LB培养基中生长。每天从生长的细胞中扩增基因,PCR产物用于Sanger测序以解码基因序列。
Storage-D在线工具的实现
Storage-D平台包含离线程序和一个集成的在线工具。Storage-D在线工具是用Python开发的,运行在Ubuntu 18.04.4 LTS上,并适应个人电脑和移动设备。它使用基于模型-视图-控制器的Web框架(https://www.djangoproject.com/) Django编写。Storage-D的前端采用Bootstrap框架(http://getbootstrap.com/)设计整体风格。它包括管理模块和显示模块。管理模块包含文件上传、编码、解码和结果下载参数。显示模块包含上传文件显示和下载文件显示。Storage-D的后端包含编解码模块和文件管理模块。此外,Storage-D还保留了未来集成其他策略的开放功能。
作者贡献
黄小罗和戴俊彪设计了“悟空”编解码方法。崔君婷、强薇和王钰编写并优化了该工具的代码。强薇进行了研究的体外实验。叶健文和谢鑫颖进行了体内验证实验。李源真进行了PCR实验和测序文库构建。黄小罗、强薇、崔君婷和叶健文为论文准备了图表。黄小罗、强薇和戴俊彪撰写了论文。戴俊彪和黄小罗指导了研究。所有作者都进行了数据分析、阅读,并批准了最终的论文。
致 谢
本研究得到了国家重点研发计划(2022YFF1201800, 2021YFF1201700)、国家自然科学基金(32201207, 32322003)、华南理工大学中央高校基本科研业务费(2023ZYGXZR095)、中国农业科学院创新计划、深圳市科技计划(KQTD20180413181837372, RCYX20221008092950122, JCYJ20220818101407017)和深圳市优秀人才培养基金的支持。我们感谢深圳合成生物学基础设施的雷朝碧在测序实验中的协助。
利益冲突声明
黄小罗和戴俊彪已申请了一项名为“悟空”算法的专利,申请号为CN202011343923.9。
数据可用性声明
Storage-D可在http://storage.dailab.xyz:16666/上获得。在Storage-D中实现的算法的代码包以及用于分析寡核苷酸池测序数据的代码可在GitHub上找到,地址为:https://github.com/DNAstorage-iSynBio/Storage-D/。补充材料(方法、图表、脚本、图形摘要、幻灯片、视频、中文翻译版本和更新材料)可在在线DOI或iMeta Science http://www.imeta.science/上找到。
道德声明
本研究与动物或人类实验无关,不存在伦理问题。
引文格式:
Xiaoluo Huang, Junting Cui, Wei Qiang, Jianwen Ye, Yu Wang, Xinying Xie, Yuanzhen Li, and Junbiao Dai. 2024. Storage‐D: A User‐Friendly PlatformThat Enables Practical and Personalized DNAData Storage. iMeta e168. https://doi.org/10.1002/imt2.168
作者简介

黄小罗(第一/通讯作者)
● 中国科学院深圳先进技术研究院高级工程师,博士生导师。
● 主持/参与承担国家重点研发计划,国家自然科学基金,国家档案局科技项目计划等项目多项。已在Nature Compuational Science,Science China Life Sciences等杂志发表论文多篇,申请/获批相关技术专利~30项。

崔君婷(第一作者)
● 西安电子科技大学通信工程专业学士,2021年入职中国科学院深圳先进技术研究院任工程师。
● 主要方向为DNA数据存储相关研究和开发。

强薇(第一作者)
● 中国科学院深圳先进技术研究院工程师,中山大学生物信息学硕士。
● 目前研究方向为DNA信息存储算法研发及数据挖掘,相关学术成果已发表于iMeta等期刊,申请专利及软件著作权8项。

叶健文(第一作者)
● 华南理工大学教授,博士生导师。
● 主要从事微生物细胞工厂设计与构建、代谢静/动态调控、转录翻译调控等领域的研究;在Nature Communications 等高水平期刊发表文章20余篇,申请发明专利18项。现担任Military Medical Research、Synthetic and Systems Biotechnology、Frontiers in Chemical Engineering期刊编辑、合成生物学创新孵化平台SynBio Hive创新大赛评委。

戴俊彪(通讯作者)
● 中国农科院深圳基因组所研究员,中国科学院深圳先进技术研究院客座研究员。
● 主要从事合成基因组学及合成生物学相关使能技术研究。戴俊彪研究员是人工合成酵母基因组国际计划(Sc2.0)中方主要参与者,基因组编写计划(GP-write)科学委员会共同主席,牵头发起了“国际基因组编写计划·中国(GP-write China)”和“Sc3.0”国际合作项目;以通讯/共同通讯作者身份在Science、Nature Plants, Nature Communications、Cell Research、Developmental Cell 等国际著名杂志发表论文50余篇;其中“酵母长染色体的精准合成”工作入选2017年度中国科学十大进展。
更多推荐
(▼ 点击跳转)
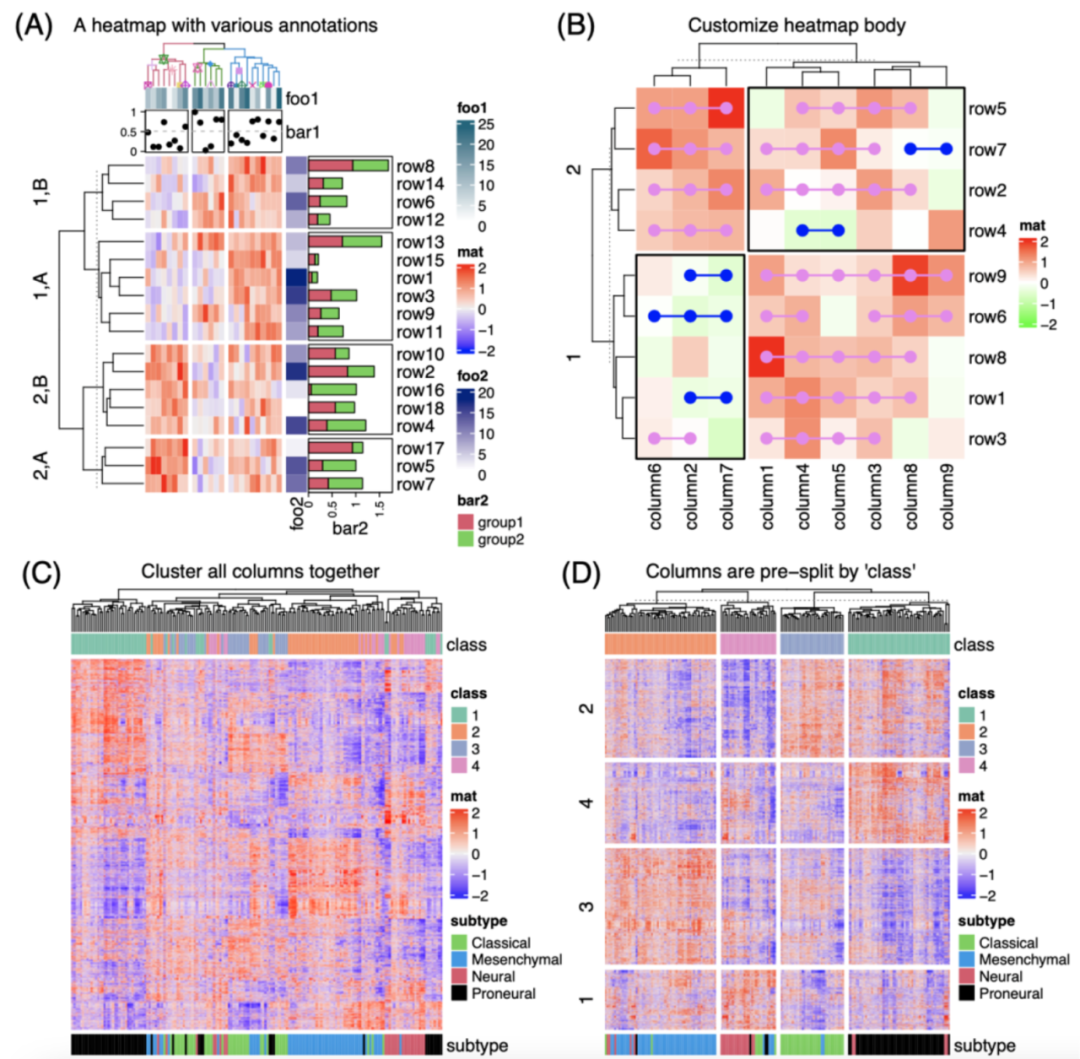
iMeta | 引用7000+,海普洛斯陈实富发布新版fastp,更快更好地处理FASTQ数据
iMeta | 德国国家肿瘤中心顾祖光发表复杂热图(ComplexHeatmap)可视化方法
1卷1期
1卷2期
1卷3期
1卷4期
2卷1期
2卷2期
2卷3期
2卷4期
期刊简介
“iMeta” 是由威立、肠菌分会和本领域数百位华人科学家合作出版的开放获取期刊,主编由中科院微生物所刘双江研究员和荷兰格罗宁根大学傅静远教授担任。目的是发表原创研究、方法和综述以促进宏基因组学、微生物组和生物信息学发展。目标是发表前10%(IF > 15)的高影响力论文。期刊特色包括视频投稿、可重复分析、图片打磨、青年编委、50万用户的社交媒体宣传等。2022年2月正式创刊发行!目前期刊已经被ESCI、Scopus等数据库收录。
联系我们
iMeta主页:http://www.imeta.science
出版社:https://onlinelibrary.wiley.com/journal/2770596x
投稿:https://mc.manuscriptcentral.com/imeta
邮箱:office@imeta.science
高颜值免费 SCI 在线绘图(点击图片直达)
往期精品(点击图片直达文字对应教程)

































































 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








