北京时间 2023 年 12 月 11 日,中国科学院深圳先进技术研究院合成所罗小舟团队在 Nature Communications 发表题为 UniKP: A unified framework for the prediction of enzyme kinetic parameters 的文章。
针对合成生物学和代谢工程领域湿实验测量酶动力学参数成本高、干实验算法难以准确预测等问题,研究团队提出了一种基于预训练大语言模型的统一框架 UniKP,可从蛋白质序列和底物结构直接高精度预测酶的动力学参数,包括酶周转数(kcat)、米氏常数(Km)和催化效率(kcat / Km),并在考虑包括 pH 和温度等环境因素、高值区间预测等特定任务上实现了很好的适应。该模型结合了目前先进的深度学习算法(IT)和生物技术(BT),实现了酶动力学参数准确高效的预测和特定酶的高效挖掘和进化,大大加速了酶改造和设计进程,为化学生物学、代谢工程等领域的研究和相关的工业应用,提供了新的解决方案。
这是罗小舟团队继 2022 年利用预训练语言模型和集成模型实现高效准确的多肽和蛋白质功能预测后(doi: 10.1093/bib/bbac476),融合前瞻性的技术在 IT 和 BT 交叉领域的又一重要进展。

文章上线截图
探秘酶的「速度秘密」:为什么这么重要?
研究特定底物的酶催化效率是一个基础而重要的生物学问题,对推动酶工程、代谢工程和合成生物学领域发展,具有深远影响。而酶在特定反应中的催化效率通常通过酶动力学参数来衡量,包括酶周转数(kcat)、米氏常数(Km)和催化效率(kcat / Km)等。然而,目前酶动力学参数的测量主要依赖湿实验,这一过程耗时、昂贵且劳动密集,导致实验测得的酶动力学参数数据库规模相对较小。以包含序列信息的 UniProt 数据库为例,它包含了超过 2.3 亿条蛋白质序列,而酶学数据库 BRENDA 和 SABIO-RK 等仅包含数万条实验测得的动力学参数值,在某些物种中可能只有数十条序列,这种数据的稀缺性极大的限制了下游系统生物学和代谢工程领域的发展。
研究人员们尝试利用计算方法加速酶动力学参数的预测过程,但当前的模型主要集中在单独的 kcat 或者单独的 Km 的预测上,通过这些工具计算得到的 kcat / Km 的数值往往与实验测量的真实值明显偏离,而这恰恰是反映催化效率最直接、最关键的参数。此外,当前模型未考虑真实生物实验场景下的适应性,如会显著影响测得的酶动力学参数数值的环境因素,生物学家关心的特定区间的预测性能等。这些局限性使得当前工具停留在理论模型的开发和数据分析上,并未对酶挖掘和定向进化等真实生物实验中面临的挑战产生实质性的影响,而解决这些挑战对下游应用具有重要意义。
黑科技登场!IT 技术助力预测
为了解决上述问题,研究团队提出了基于预训练大语言模型和机器学习模型的酶动力学参数预测框架(UniKP),该框架仅通过给定酶的氨基酸序列和底物的结构信息,就可以实现多种不同的酶动力学参数(kcat、Km、kcat / Km)的预测。此外,为了实现对生物实验中不同任务场景的适应,我们对模型和数据进行了微调以实现更好的酶动力学参数的预测,包括针对不同环境因素、高值区间的预测等(图 1)。

图 1 UniKP 框架结构
首先,研究团队以代表性的 kcat数据集为例,系统的探索了机器学习模块 16 种不同的机器学习模型和 2 种代表性的深度学习模型(卷积神经网络和循环神经网络)在该数据集上五折交叉验证的性能,结果显示集成模型表现最好,尤其是随机森林和极端随机树显著优于其他模型,其中极端随机树表现最佳(R2 = 0.65)(图 2)。
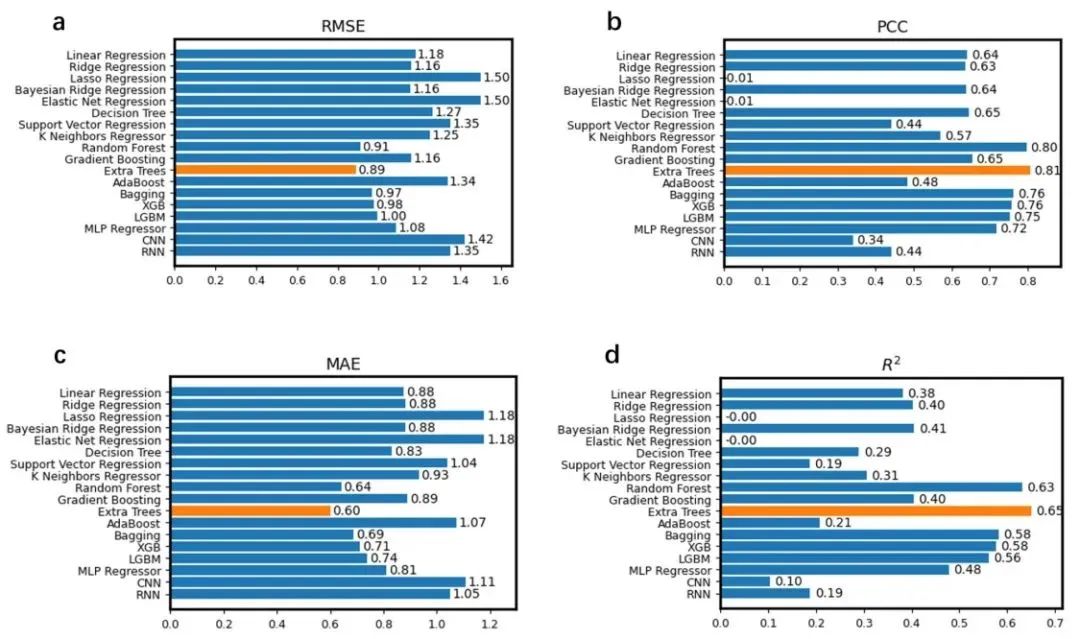
图 2 不同机器学习模型的性能比较(机器学习模块)
接着,研究团队证实了 UniKP 框架在多个评估指标上显著超过了目前最先进模型的性能。UniKP 框架在独立测试集上,决定系数 R2 相较于最先进的模型有高达 20 个百分点的优势;此外,在更严格的数据划分规则和不同数值区间下,UniKP 也展现出了更好的稳健性。并通过 SHAP 特征分析发现,UniKP 显示出对酶信息有一定的偏好性,这证实了在催化反应中酶信息的关键性作用。此外,UniKP 也可以显著地对野生型酶及其突变体进行区分(图 3-4)。
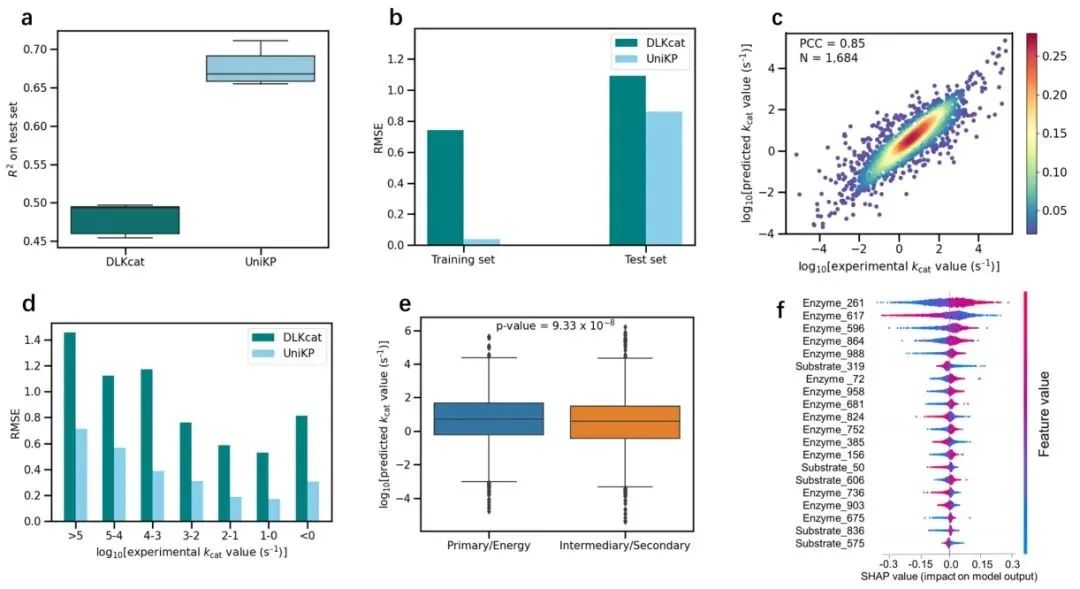
图 3 UniKP 以高度的准确性预测 kcat 数值
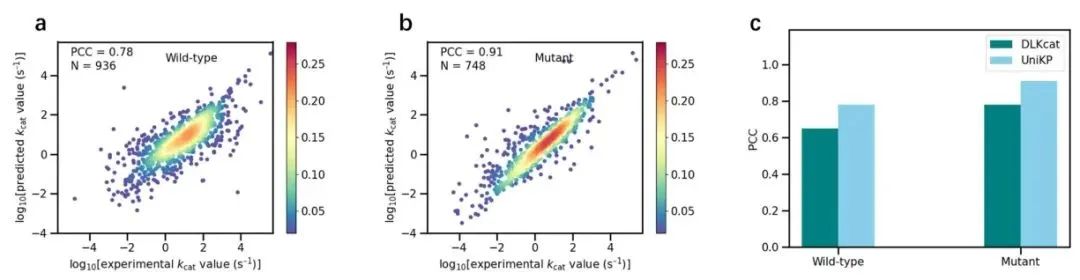
图 4 UniKP 显著区分野生型酶及其变体
更真实的预测:考虑环境因素的两层框架
为了更好的模拟生物实验环境,研究团队进一步通过将环境因素纳入考量,提出了基于 UniKP 的两层框架: EF-UniKP,实现了更准确地酶动力学参数的预测。为了验证这个策略,我们创建了涵盖 pH 和温度信息的两个数据集来对 EF-UniKP 进行评估。在测试集上,EF-UniKP 相较于 UniKP 和 Revised UniKP 表现更佳,R2 分别高出 20% 和 8%(pH 数据集),以及 26% 和 2%(温度数据集)。在酶和底物至少有一个不在训练集的测试中,EF-UniKP 在 pH 数据集上的 R2 值相较于 UniKP 和 Revised UniKP 分别高出 13% 和 10%,在温度数据集上分别高出 16% 和 4%。此外,RMSE 和 PCC 的优势也证实了 EF-UniKP 在考虑环境因素下对模型的显著改善(图 5)。
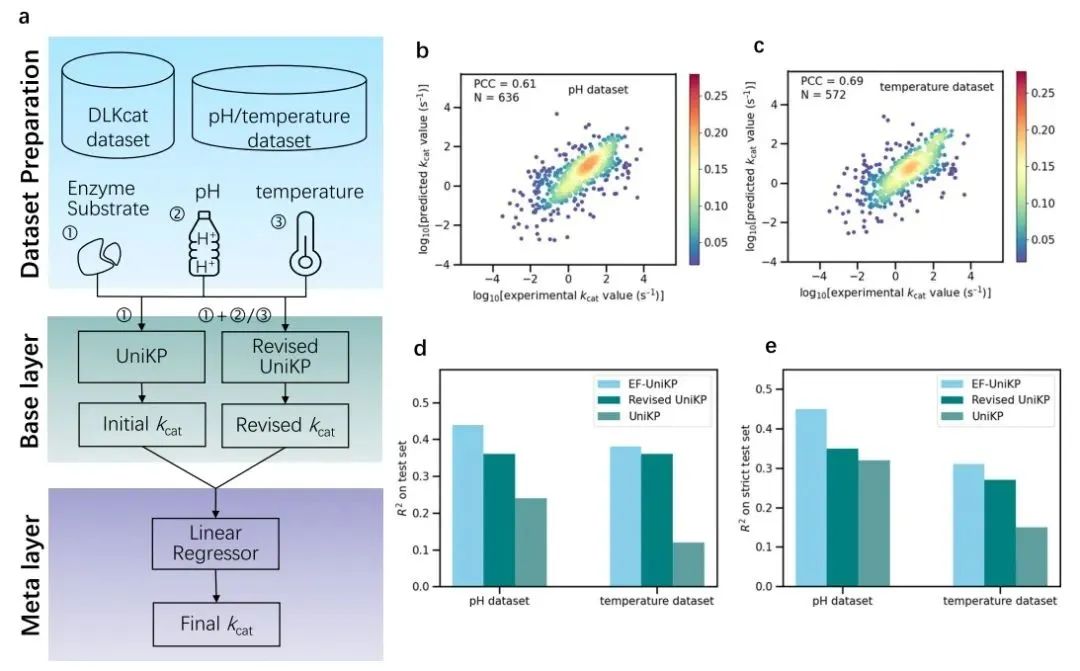
图 5 EF-UniKP 框架实现环境条件下更准确的预测
重塑实验数据,预测更具针对性
此外,在对已有的 kcat 数据集分析发现,其分布高度不均衡,大部分样本集中在中间,两端只有少数样本,呈现正态分布的特征。这种严重的数据失衡导致了高 kcat 值区间的预测有较高的误差。为了解决该问题,研究团队运用了代表性的重新加权方法,包括直接修改样本权重(DMW)、成本敏感的重新加权方法(CSW)、类平衡的重新加权方法(CBW)以及标签分布平滑(LDS)等,以 kcat数据集为例进行了优化。这些方法都显著降低了高值区间的误差,其中 CBW 效果最佳,高 kcat 值的样本在 CBW 的优化下均方根误差比初始模型降低了 6.5%(图 6a-c)。
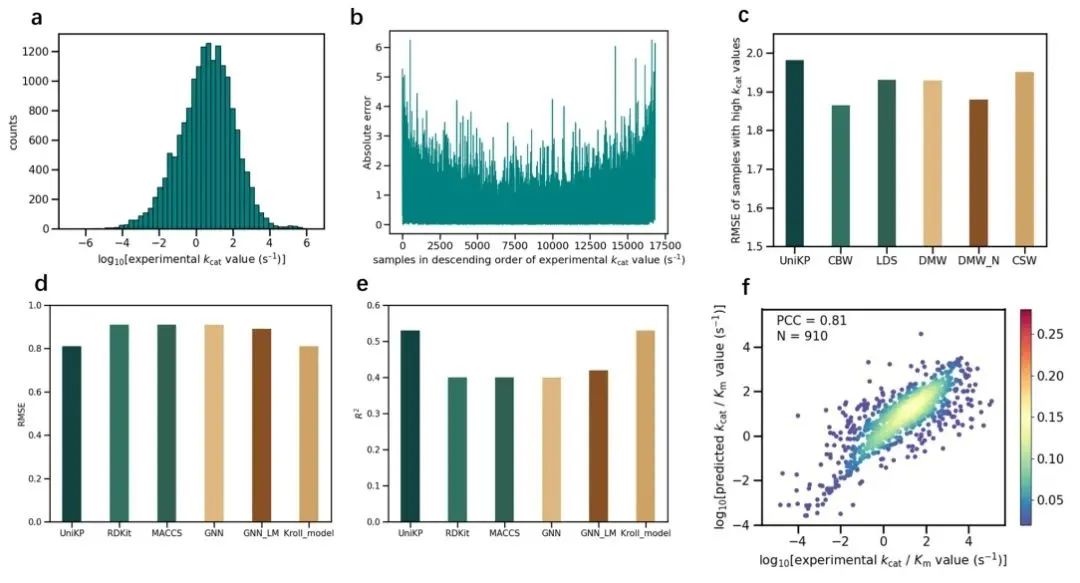
图 6 重新加权方法改善高值区间预测 & UniKP 框架泛化性的验证
进一步地,为了评估 UniKP 框架在 Km 和 kcat / Km任务上的预测性能,研究团队选取了目前公开可获得的 Km数据集和新构建的 kcat / Km 数据集,获得了目前最先进的预测性能,这也证实了通过该框架可以实现小分子-蛋白质相互作用任务的统一化预测(图 6d-f)。
UniKP 助力酶挖掘和定向进化
最后,为了探索 UniKP 及其衍生框架在酶工程领域的实际应用,研究团队以类黄酮合成中的关键限速酶,酪氨酸脱氨酶(Tyrosine ammonia lyase, TAL)为例来进行挖掘和进化。实验结果表明,UniKP 有效地识别了在数据库中 TAL 的同工酶里以及已知 TAL 的突变体里的高活性 TAL 酶,无论是挖掘新酶,还是进化已有酶,都得到了显著提高的催化效率,其中 RgTAL-489T 的 kcat / Km值比野生型酶高出 3.5 倍。此外,衍生框架 EF-UniKP 也可以在考虑环境因素的情况下,精准识别高活性的 TAL 酶,验证的 5 条序列 kcat 和 kcat / Km 值均优于野生型 TAL,最高的的 kcat / Km 值比野生型酶在给定 pH 情况下高出了 2.6 倍。该结果证实了 UniKP 可以有效的加速酶挖掘和进化的过程,有望成为推动生物催化、药物发现、代谢工程等领域的强大工具(表 1)。

表 1 UniKP 和 EF-UniKP 辅助酶的挖掘和进化
总结与展望
该项工作融合了先进的人工智能模型(IT 技术)和生物实验(BT 技术),建立 UniKP 框架实现了酶动力学参数的准确预测,同时对环境因素、高值区间等特定的实验任务进行了很好的适应。实验结果表明,利用 UniKP 框架可以有效助力酶挖掘和定向进化的过程,快速筛选到对特定底物更高活性的变体,显著降低了酶筛选的时间和成本。目前,研究团队正在与合成生物学领域公司森瑞斯生物科技展开进一步的合作,推动该技术的落地和转化。
该项工作利用 IT 领域先进的人工智能技术在 BT 领域进行尝试,即合成生物学基本的功能元件酶的催化效率的预测,表明了人工智能在合成生物学领域应用的巨大潜力,这对酶工程、系统生物学、代谢工程等领域具有重要的参考价值和应用意义,也为合成生物学方法的设计和研究开拓了新的思路。
中国科学院深圳先进院合成生物学研究所研究员罗小舟为本文的通讯作者,团队成员硕士研究生余函和助理研究员邓华祥为文章共同第一作者,研究助理何佳慧在生物实验等方面做出了重要贡献,Jay D. Keasling 教授在实验设计及文章修改方面提出了宝贵意见。该研究获得国家重点研发计划、国家自然科学基金委、广东省基础与应用基础研究基金委、深圳市科技计划等多个项目的支持,以及深圳市微生物药物智能制造重点实验室、深圳合成生物学创新研究院和定量合成生物学重点实验室等平台的支持。同时,感谢科研助理魏珍琴在项目实施过程中协助组织会议讨论等支撑工作。
课题组招聘
罗小舟,深圳先进院合成生物学研究所研究员,博士生导师,合成生物化学研究中心执行主任、PI,深圳市合成生物研究重大科技基础设施副总工艺师,森瑞斯生物科技(深圳)有限公司创始人。2010 年于新加坡南洋理工大学获得学士学位,2016 年于美国圣地亚哥斯克里普斯研究所获得化学专业博士学位(导师 Peter G. Schultz 院士),2016-2019 于加州大学伯克利分校进行博士后研究(合作导师 Jay D. Keasling 院士)。2019 年加入中国科学院深圳先进技术研究院。先后入选国家重大人才工程(青年)专家、广东省杰青、深圳市优青、深圳市国家级高层次人才、2023 南山十大杰出青年等。在 Nature, Nature Chemical Biology, Cell Chemical Biology, Nature Synthesis, Nature Communications, PNAS, Angewandte Chemie 等知名学术期刊上发表论文 40 余篇。课题组聚焦于合成生物学领域中生命体内生物化学过程相关研究,主要结合遗传密码扩充技术,酶的定向进化,基因挖掘和代谢工程等多种化学生物学方法,基于大数据机器学习及高通量自动化,深入研究多种不同类别的天然产物及其衍生物的生物全合成的方法,并利用合成生物学方法,将研究成果转化至制药、个性化治疗、新材料等领域。
课题组现长期诚聘生物、化学、生物信息学、生物医学工程等交叉学科背景,或有酶定向进化、机器学习、高通量筛选、天然及非天然化合物的生物全合成等研究背景的博士后,欢迎感兴趣的同志投递简历至邮箱 xz.luo@siat.ac.cn。
高颜值免费 SCI 在线绘图(点击图片直达)
最全植物基因组数据库IMP (点击图片直达)
往期精品(点击图片直达文字对应教程)
























































 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








