本文代码已经上传至https://github.com/iMetaScience/iMetaPlot如果你使用本代码,请引用:Changchao Li. 2023. Destabilized microbial networks with distinct performances of abundant and rare biospheres in maintaining networks under increasing salinity stress. iMeta 1: e79. https://onlinelibrary.wiley.com/doi/10.1002/imt2.79
代码翻译及注释:农心生信工作室
写在前面
冲击图(alluvial diagram)是流程图(flow diagram)的一种,最初开发用于代表网络结构的时间变化。在微生物群落分析中可以用来展示不同分类学水平微生物的丰度及其从属关系,利用R语言做冲击图实现菌群相对丰度的变化是生信分析中较常见的。
本期我们挑选2023年1月9日刊登在iMeta上的Destabilized microbial networks with distinct performances of abundant and rare biospheres in maintaining networks under increasing salinity stress,以文章中Figure 1E为例,讲解和探讨如何用冲击图展示菌群随盐度的变化,先上原图:

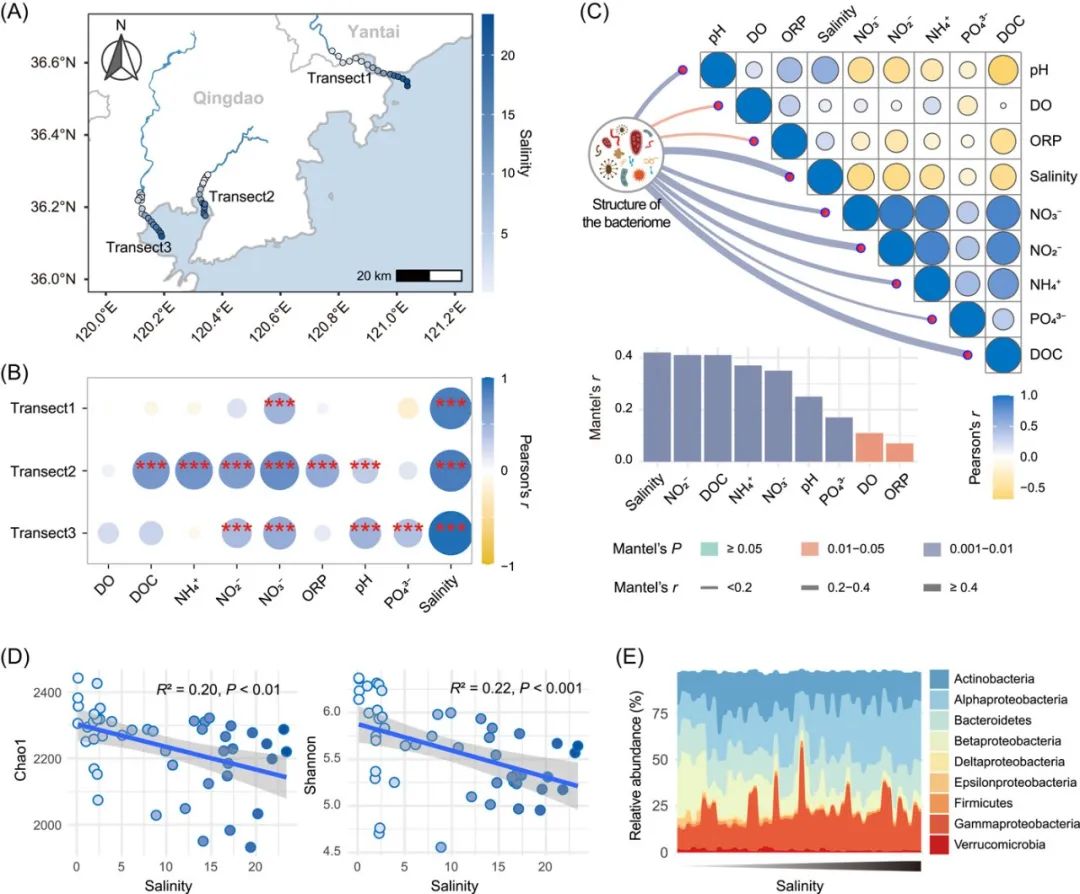
代码、数据和结果下载,请访问https://github.com/iMetaScience/iMetaPlot/tree/main/230407ggalluvial
接下来,我们将通过详尽的代码逐步拆解原图,最终实现对原图的复现。
R包检测和安装
01
安装核心R包ggalluvial以及一些功能辅助性R包,并载入所有R包。
library(reshape2) # 重塑数据框的包,可以实现长数据和宽数据之间的转化# 加载ggalluvial包(绘图本图所用关键R包),基于ggplot2的ggalluvial包可用来绘制冲击图if (!require("ggalluvial"))install.packages("ggalluvial")library(ggalluvial)library(ggsci) # ggsci包含期刊或软件的经典配色,可用于ggplot2()配色
读取数据
02
读取绘制所需数据并进行必要转换。
# 读取绘制数据,行为菌的名称,列为不同盐度,表中数据为菌的相对丰度d1e0 <- read.csv("ProClass.csv") # 示例数据可在GitHub上获取# 将宽数据拉长,转化后"自变量"列为variable,代表梯度不同的盐度,"因变量"列为Value,代表菌的相对丰度d1e = as.data.frame(melt(d1e0, id.vars = c("phylumpro"))) # id.vars=c()用于指定要保留的字段head(d1e) # 列出前几行看看数据转化后长什么样
绘制冲击图
03
基础图形绘制。
# 预设渐变色taxcol <- c("#64A4CC", "#9CCEE3", "#C7E5DB", "#ECF6C8", "#FEEDAA","#FDC980", "#F89D59", "#E75B3A", "#D7191C")p1e <- ggplot(data=d1e,aes(x = variable, y = value, alluvium = phylumpro))+ # alluvium设置冲积层,在这里将菌(phylumpro)设置冲积层geom_alluvium(aes(fill = phylumpro), alpha = 1)+ # 画冲击图,按菌(phylumpro)进行颜色填充scale_fill_manual(values = taxcol) # 对冲积层填充颜色p1e
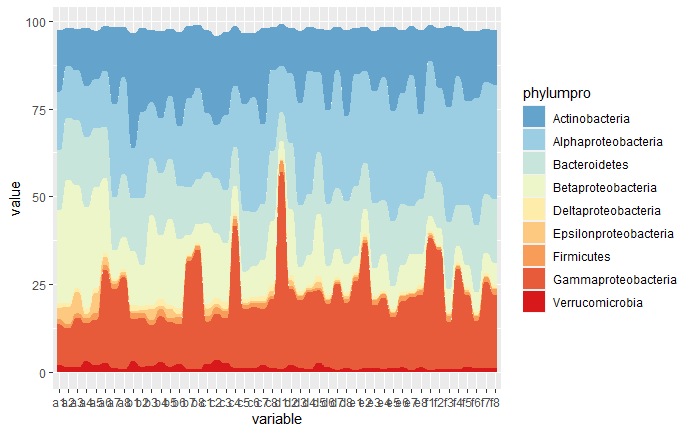
04
对图形进行细节美化
p1e1 <- p1e +theme_minimal() + #预设主题theme(axis.text.x = element_blank(), # 因预期x轴另外加一条渐变的图形表示,在这里取消x轴panel.grid.minor = element_blank(), # 移除图中的网格panel.grid.major = element_blank(), # 移除图中的网格panel.spacing.x = element_blank()) +xlab("Salinity")+ # 设置x轴标题ylab("Relative abundance (%)")# 设置Y轴标题p1e1
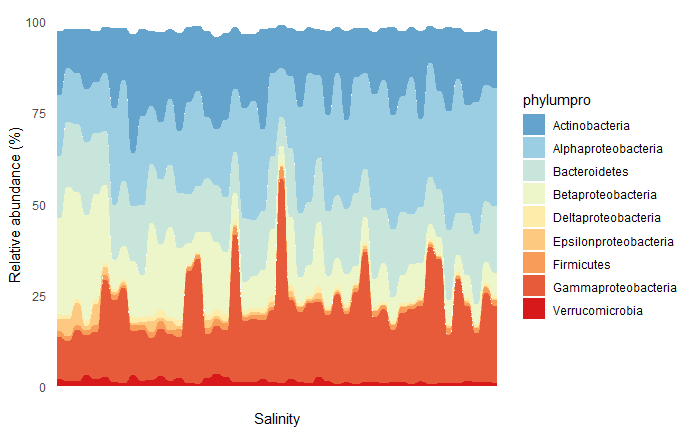
完整代码
library(reshape2) # 重塑数据框的包,可以实现长数据和宽数据之间的转化# 加载ggalluvial包(绘图本图所用关键R包),基于ggplot2的ggalluvial包可用来绘制冲击图if (!require("ggalluvial"))install.packages("ggalluvial")library(ggalluvial)library(ggsci) # ggsci包含期刊或软件的经典配色,可用于ggplot2()配色# 读取绘制数据,行为菌的名称,列为不同盐度,表中数据为菌的相对丰度d1e0 <- read.csv("ProClass.csv") # 示例数据可在GitHub上获取# 将宽数据拉长,转化后"自变量"列为variable,代表梯度不同的盐度,"因变量"列为Value,代表菌的相对丰度d1e = as.data.frame(melt(d1e0, id.vars = c("phylumpro"))) # id.vars=c()用于指定要保留的字段# 预设渐变色并绘制冲击图taxcol <- c("#64A4CC", "#9CCEE3", "#C7E5DB", "#ECF6C8", "#FEEDAA","#FDC980", "#F89D59", "#E75B3A", "#D7191C")p1e <- ggplot(data=d1e,aes(x = variable, y = value, alluvium = phylumpro))+ # alluvium设置冲积层,在这里将菌(phylumpro)设置冲积层geom_alluvium(aes(fill = phylumpro), alpha = 1)+ # 画冲击图,按菌(phylumpro)进行颜色填充scale_fill_manual(values = taxcol)+ # 对冲积层填充颜色theme_minimal() + #预设主题theme(axis.text.x = element_blank(), # 因预期x轴另外加一条渐变的图形表示,在这里取消x轴panel.grid.minor = element_blank(), # 移除图中的网格panel.grid.major = element_blank(), # 移除图中的网格panel.spacing.x = element_blank()) +xlab("Salinity")+ # 设置x轴标题ylab("Relative abundance (%)")# 设置Y轴标题p1eggsave("冲击图展示菌群随盐度的变化.pdf", p1e, width = 8, height = 5)
以上数据和代码仅供大家参考,如有不完善之处,欢迎大家指正!

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










