核糖核酸(RNA)是一种重要的核酸,在调节多种生物活性中起着至关重要的作用。
最近,一项研究利用机器学习算法自动分类 耻垢分枝杆菌孔a纳米孔陷阱产生的RNA结构。
虽然它可以获得理想的分类结果,但与深度学习(deep learning, DL)方法相比,这种经典的机器学习方法需要领域知识手动提取特征,复杂、劳动密集型和耗时。同时,生成的原始RNA结构事件长度不是严格相等的,这与DL模型的输入要求不兼容。
为了解决这一问题,我们提出了一个序列到序列(sequence-to-sequence, S2S)模块,该模块将不相等长度的序列(UELS)转换为相等长度的序列。
此外,为了从RNA结构事件中自动提取特征,我们提出了一种基于DL的序列-序列神经网络。此外,我们增加了一个注意机制来捕捉重要的信息分类,如停留时间和堵塞幅度。通过定量和定性分析,实验结果与之前的方法相比,性能提高了约2%(准确度)。该方法也可以应用于其他纳米孔平台,如著名的牛津纳米孔。
值得注意的是,所提出的方法不仅旨在追求最先进的性能,而且还提供了一个整体思路,以处理纳米孔数据与UELS。
1.提取的序列到序列的模块 S2S 把不等长的序列转为等长的序列
2.net 添加注意力机制来捕捉信息间的分类.
3.关于处理纳米孔数据 和uels提供了思路
介绍
核糖核酸(ribonucleacids, RNAs)在调节基因表达和生理方面发挥着重要作用[1,2]。RNA在生物体中普遍存在,在维持生物活性方面发挥着积极作用。rna具有多种功能,包括蛋白质翻译、基因沉默、表观遗传调控、遗传信息存储和生物催化等[3,4]。RNA的功能多样性来自于其高度通用的三级结构[5,6]。然而,现有的关于RNA结构和功能的研究大多需要复杂的预处理过程,这使得直接检测RNA结构非常困难。因此,有必要研究rna的功能和结构之间的关系。原则上,高通量测序技术的发展开启了基因组学的新纪元。脱氧核糖核酸(DNA)和RNA序列的爆炸式增长使世界各地的研究人员受益匪浅。然而,大多数来自(或组成)这些序列的rna需要进一步的结构表征。一般来说,RNA结构表征(即功能性RNA结构的识别)需要结合热力学、系统发育和实验分析。随着RNA种类的增多,精细的RNA鉴定需要昂贵的设备,进一步的实验分析需要快速、廉价的鉴定方法。
== 特别是纳米孔已经成为一种新兴的大分子识别技术==,实现了DNA(或RNA)的测序、检测和数据存储[ 7,8]。实验上,该纳米孔装置由两个充液储层组成,储层由单个纳米孔[9]连接。被驱动通过纳米孔的分子可以产生特征封锁的电流,反映了它们的物理化学性质和结构信息。具有代表性的纳米孔类型是生物纳米孔,它已经发展成为一系列用于单分子传感和测序的通道蛋白[10-12]。
然而,对具有复杂结构的生物分子(如RNA)的分析是有限的,因为这些分子太大,无法通过大多数生物纳米孔。耻垢分枝杆菌孔A (MspA)是一个由刚性β-桶结构组成的锥形生物纳米孔。在最近的一篇论文[13]中,作者利用MspA纳米孔的纳米空腔在单分子水平上进行RNA三级结构分析。
MspA纳米孔可以直接分辨出小干扰RNA (overhung small-interfering RNA, siRNA)、钝性siRNA (blunt siRNA)、转移RNA (transfer RNA, tRNA)和5S核糖体RNA (5S rRNA, 5S rRNA)等低分子量RNA结构。
众所周知,由于纳米孔信号在传感和测序方面的多样性,目前的纳米孔数据分析依赖于其具体的应用。现有的纳米孔数据处理方法主要通过以下两种策略:
(1)统计方法,通过统计分析具体识别统计差异;
(2)基于机器学习的方法,这是一种新兴而强大的工具。虽然这些方法提供了有价值的策略,但它们受到昂贵计算和缺乏专业特征选择的限制。
具体来说,在前一篇论文[13]中,作者应用了随机森林算法[14]对不同RNA分子的信号进行自动分类。虽然随机森林算法可以取得显著的性能,但它需要专业领域知识来进行特征选择,这限制了算法的有效性。
上述经典方法往往需要复杂的特征提取,耗时较长。相比之下,
深度学习(deep learning, DL)是另一种分析纳米孔数据的方法[15-22]。由于该方法具有端到端特性,因此被认为是一种很有前途的特征提取工具。近年来,DL在机器学习的各个应用领域取得了巨大的成功,包括计算机视觉、自然语言处理(NLP)和广泛的数据分析。特别是,由于不同的分子在纳米孔中保持时间差异,因此纳米孔信号是不等长序列。
相关研究将DL用于UELS[22],其中数据集是由Oxford Nanopore Technologies (ONT)设备生成的。任务是对八类条形码进行分类。由于序列之间的长度差较小,他们主要通过插值技术对UELS进行变换。
然而,对于RNA数据集来说,序列长度可能相差数万个点,这是插值技术难以处理的。
因此,为了克服这些限制,我们开发了一种替代策略,将unequal length sequence (UELS)与等长序列(ELS)对齐,称为序列-序列(S2S)模块。随后,我们利用这些创新构建了一个基于dlc的序列到序列神经网络(S2Snet)来自动提取特征和分类。本文的主要贡献如下:
本文主要贡献
(i)我们首先使用MspA纳米孔来检测不同的RNA物种。每个RNA事件都有很好的可区分的特征
(ii)我们提出了一个S2S模块,该模块对纳米孔数据进行转换策略。S2S为UELS提供了定长向量表示
(iii)我们提出了一个基于S2S模块的S2Snet。在纳米孔RNA数据集上的实验验证了S2Snet达到了最先进的性能。
(iv)进一步评价所提方法在特征提取方面的有效性,通过对特征映射结果的分析验证了DL在特征提取方面优于传统机器学习。
Preliminaries (前言):
在描述所提出的S2Snet结构之前,本节我们将简要解释理解所提出方法的三个基本概念:(1)重点介绍RNA类型预测问题,(2)简要描述DL在纳米孔领域的应用,(3)简要描述注意机制
Problem statement(问题陈述)
在这里,我们首先澄清了与RNA类型预测相关的几个概念。显然,纳米孔数据被认为是一个连续的时间序列,这意味着处理时间序列的几个概念可以映射到纳米孔数据分析。因此,在本文中,RNA类型预测的主要对象与纳米孔的时间序列有关。
一般来说,假设给定一个纳米孔信号,RNA类型预测的任务是预测纳米孔序列类型。
Input :纳米孔信号
Output: 纳米孔序列的类型
通常我们用S来表示原来的长序列,用T来表示RNA类型。然后,分割方法将长原始序列分割成短UELS (unequal length sequence)
(Supplementary Section B),将长序列S截断为n个子序列,S = [s1, s2,…]。, sn], T型RNA被截断为n个子靶点T = [t1, t2,…]。, tn]。从根本上说,RNA类型预测可以定义为一个分类任务,学习n个子序列和子目标的函数映射。假设新的输入序列为x,模型可以输出y型RNA,如下所示:
f(x) : (s, t) → y. (1)
为了克服人工特征提取的局限性,本文采用深度学习的方法进行特征提取。如图1C所示,

纳米孔序列分析方法,上半部分为传统手工特征工程,下半部分为DL方法
必须解决一个问题:DL模型输入序列的长度必须相等。因此,我们使用S2S模块(参见序列到序列段)将n个子序列s转换为n个相等长度的子序列a = [a1, a2,…,一个]。同时,变换后的长度明显小于原始的纳米孔序列信号长度。因此,这个问题可以重新表述为:
g(x) : (a, t) → y. (2)
Deep learning在纳米孔领域的应用
DL是一种最先进的机器学习技术,被用于解决语音识别、纳米孔数据分析等复杂问题。DL模型可以描述为一个函数f,

其中θ是将输入数据转换为输出数据的参数
DL通常是一个分层非线性模型,由多个堆叠的处理层[23]组成。除输入层和输出层外,输入层和输出层之间还有多个隐藏层。对于l-层DL模型,第l层的映射函数,输入来自(l−1)第l层,输出xl-1,定义如下:
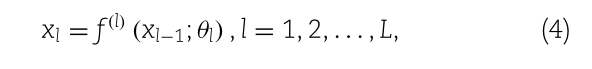
式中θ l为LTH层参数,f (l)(xl−1;θl)为LTH层的映射函数。
注意机制
基于注意的神经网络模型已成功应用于图像处理[24-27]、NLP[28-30]、语音识别[31]和序列分析[32]。注意机制最早应用于自然语言处理领域,它可以定义为一个神经网络翻译机器来寻找一个注意向量,并用一个固定大小的向量有效地表示整








 最低0.47元/天 解锁文章
最低0.47元/天 解锁文章















 556
556











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








