DOI: 10.1186/s13059-023-02921-0
期刊 :Genome Biology
中科院分区:Q1
影响因子:12.3
作者 Longxian Chen; Liang Ou; Xinyun Jing; Yawei Kong; Bingran Xie; et al
出版日期 2023-04-17
网址: https://genomebiology.biomedcentral.com/counter/pdf/10.1186/s13059-023-02921-0
摘要
A-I的RNA修饰的单分子检测阶段仍然是一个不好解决的问题。long-read和纳米PCR-free技术为最原始的RNA测序和检测提供了方便。在本文中,作者设计出了一个神经网络模型DeepEdit,该模型可以解决两个问题:第一个就是能够识别RNA序列纳米孔中A-I的编译过程;第二个就是RNA编译的转录过程。通过两组数据(粟酒裂殖酵母和智人转录组数据)来验证DeepEdit模型的鲁棒性。
创新点:该模型能够从新的角度为RNA编辑提供有力工具
背景
RNA修饰在不同生命的转录过程十分关键。腺苷脱氨基是多细胞动物中最丰富的RNA修饰,用来将腺苷转化为肌苷( A-to-I ) 在翻译过程中 ,随着肌苷被翻译成鸟嘌呤(G).A-I这种RNA修饰很容易改变蛋白质序列,并且影像着蛋白质产品的诸多功能。
之前对于鉴定RNA修饰位点的方法:通过分析作图数和基因组参考之间单个核苷酸发生变异,短读数全转录组测序是一种高通量测序技术。
然而该方法也面临着一些问题,该方法面临着无法确定编辑碱基的相对位置信息、可能会误识别假阳性编辑位点以及计算复杂性难以解析RNA编辑事件与其他转录后事件之间的关系等几个挑战。
对于牛津纳米孔技术的出现不需要反转录和PCR扩增技术也能够解决RNA测序问题 。通过记录电信号的变化,有很多用于RNA修饰的相关测序。再进一步的研究方面,机器学习也被用到纳米孔测序,但是对于RNA修饰的纳米孔测序读数问题还是有所欠缺,对于解决更进一层的RNA修饰的问题更是不可能。
在本研究中,作者为了解决不同纳米孔读数之间的RNA修饰问题,开发了一个神经网络模型,命名为DeepEdit,该模型可以识别单个纳米孔读数的A到I的修饰问题,而且还能够通过RNA纳米孔确定转录物上的位置信息。
方法和数据集
数据集
我们可能和机械相关的我目前想到的就是试剂盒的应用,在本文中也有所体现,对于外观的设计可能有所相关,本文中所用到的是质粒迷你试剂盒来提取质粒,随后再用凝胶试剂盒转化成菌株,随后在培养基中培养菌株,随后在中期提取细胞中的DNA和RNA,在此只对该问题进行了简单描述,因为这些过程过于偏向生物,不过多赘述,如果像深入了解可以看原文熟悉一下。
S.pombe RNA序列数据集
还需要使用Illumina生成测序文库,产生长度为150bp的序列,对于得到RNA修饰位点的步骤还需要经过使用Samtools的mpileup工具包,再通过以下标准步骤来得到高可信度的A-I RNA修饰位点。包括:(1)仅保留在FY-ADAR2-1和FY-ADAR2-2样本中出现的A-to-G单核苷酸变异(SNVs);(2)删除在FY-HFF1-1或FY-HFF1-2中出现的A-to-G SNVs;(3)删除与基因组SNP重叠的位点;(4)所有FY-ADAR2样本中的覆盖深度≥50且编辑比例≥0.1。综上所述,该实验使用了一系列的步骤和方法,包括数据修剪、去重、质量过滤、比对、变异调用和筛选,以鉴定A-to-I RNA编辑位点。
H. sapiens RNA序列数据集
通过NCBI数据库进行下载,通过上述相同的方法来进行筛选合适的修饰位点。
纳米孔的RNA序列数据集
通过牛津纳米孔数据获得的,采用试剂盒进行测序
H. sapiens的纳米孔数据集
从Github上下载GM12878的纳米孔直接RNA测序数据,使用ONT R9.4方法获得1300的纳米孔数据。
通过KS-test获得2318个修饰A位点的数据,非位点一样。
特征
在这里的特征提取与我们之前所理解的有所不同, 把以下四种定义为特征:分别是“平均值”(norm_mean)、“标准差”(norm_std)、“长度”(length)以及“碱基一致性”(base identity)。这些特征是根据编辑位点周围六个碱基的信息计算得出的。以上的数据选择用80%的数据作为训练集,20%的数据集作为测试集。
方法
DeepEdit
本文最主要的用到的一个深度学习的算法CNN,DeepEdit的神经网络模型具有一个相对较大的输入层,两个较小的隐藏层,和一个输出层,用于执行二元分类任务,即预测一个样本是否为正例。dropout正则化技术有助于提高模型的泛化性能,防止过拟合。这种模型结构通常用于处理具有复杂特征的数据集,如文本分类或图像识别等任务。 流程图解释
流程图解释
DeepEdit对于人类数据集的应用
为了评估模型的性能,选择已编辑和未编辑的位点,如果编辑比例大于0.9则被认为完全编辑,如果编辑比例为0则认为未编辑,正负样本的设计也是同理,接下来的步骤特征提取等与上述方法一致,进一步来证明模型的稳定性。
结果和讨论
讨论
这项研究描述了构建酵母菌株来研究A-to-I RNA编辑的实验过程,简言之就是自己构建了数据集的过程。以及如何通过引入人类ADAR2基因并进行验证,成功地实现了A-to-I RNA编辑的检测。这一研究为进一步研究RNA编辑事件提供了重要的实验基础。
为了研究A-I RNA修饰对于纳米孔测序中对电信号的变化做了一些实验,首先采用不同的RNA样本,FY-ADAR2和FY-HFF1从这两个样本中获得了大量的数据,分别是2,328,631个读数来自FY-HFF1,以及4,224,232个读数来自FY-ADAR2。研究结果显示,在编辑位点及其附近的碱基位置(-3,-2,-1和+1)上,编辑后的“A”位点的电信号与FY-HFF1中的相应位点存在显著差异。这表明A-to-I RNA编辑事件会导致纳米孔原生RNA测序中的电信号发生变化。特别地,编辑后的碱基电信号受到最大影响,而-1,-2,-3和+1位置的电信号受到较小程度的影响。其他更远的位点几乎没有受到影响。
此外,在研究过程中也发现了一个问题,对于碱基的识别存在一个特殊错误(ASEs)ASEs在编辑位点和附近碱基位置经常出现,主要集中在-1到+1位置(见图1e和附表2)。相比之下,在随机“A”位点周围只观察到基线水平的、随机分布的错误(附表1),这表明ASEs与RNA编辑事件之间存在相关性。这些错误可以帮助区分编辑过的RNA分子和未编辑的RNA分子。这对于纳米孔测序中的RNA编辑研究非常重要。
结果
特征选择结果
为了获得最佳性能的神经网络模型,他们选择了并比较了五种不同的原始特征,包括电信号均值、相邻碱基间的均值偏差、标准偏差、原始信号值的数量和碱基类型。独立的交叉验证显示,除了相邻碱基均值偏差外,其他原始特征的组合显著提高了性能。电信号均值、碱基类型、标准偏差和信号值数量的组合表现最佳(AUC分数:0.9653)

DeepEdit应用于总共79,426个纳米孔读数后,取得了AUC(曲线下面积)得分为0.9076,表明DeepEdit在区分编辑的RNA分子和未编辑的RNA分子方面表现良好。此外,应用DeepEdit来预测基因组位点上的编辑状态时,编辑位点的编辑比例显著高于未编辑位点,这也进一步表明DeepEdit在人类中具有良好的性能。
与其他研究的比较:研究人员还将他们的方法检测到的RNA编辑位点与以前已发表的研究中报告的位点进行了比较。结果显示,与第一项研究的位点有56.32%的重叠率,与第二项研究的位点有54.98%的重叠率。这表明,与以前的研究相比,我们的方法具有可比较的重叠率。
综合来看,研究结果表明,使用S. pombe数据训练的DeepEdit模型在不同物种上都具有广泛的应用潜力,可以有效地检测RNA编辑事件。DeepEdit与其他方法相比,如Dinopore,具有明显的优势。它能够在单个纳米孔读数中检测RNA编辑事件,而其他方法只能提供编辑位点的预测。此外,DeepEdit还可以确定编辑事件的相位信息,这是其他方法无法实现的。
长读取方法优势:DeepEdit采用长读取方法,相比短读取方法具有多个优势。首先,它可以在重复元素和高度编辑的区域中检测编辑位点,而短读取方法通常会错过这些区域。其次,DeepEdit具有单分子分辨率,可以准确报告RNA分子上的编辑位置。
编辑事件相位信息:DeepEdit能够提供A-to-I RNA编辑事件的相位信息,即哪些RNA分子在哪些位置进行了编辑。这对于了解编辑事件的生物学含义非常重要。
应用领域:RNA编辑在多种人类疾病中扮演关键角色。DeepEdit的能力可以用于研究蛋白质编码和RNA次级结构变化等分子调控过程。
局限性:尽管DeepEdit具有多个优势,但它也有一些局限性。为了训练该模型,需要从头构建RNA编辑机制,这在一些特定区域仍然具有挑战性。研究人员计划通过创建来自多个物种的额外训练数据集来克服这一问题,以扩展DeepEdit的应用范围。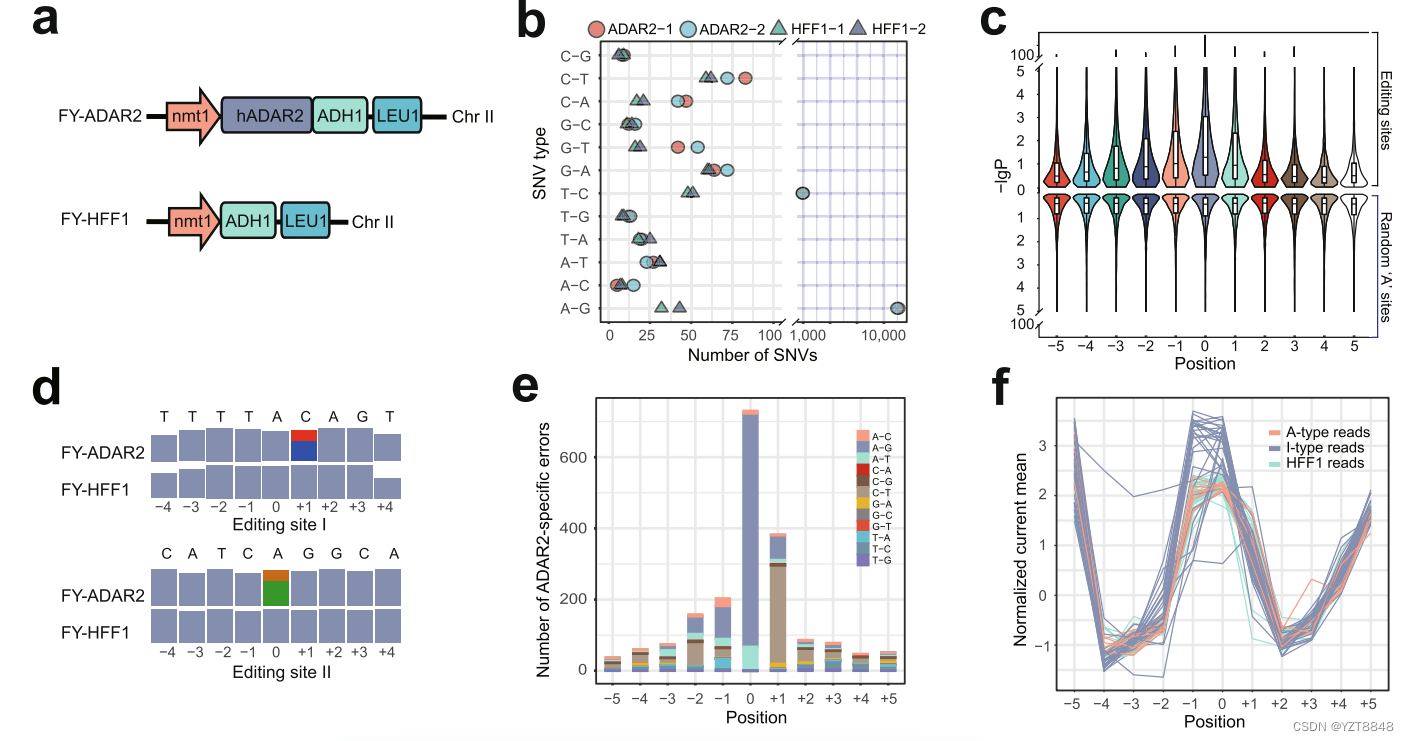
结论
这项研究展示了一个名为DeepEdit的工具,它可以在不同物种的RNA测序数据中检测和分析RNA编辑事件,这有助于我们更深入地了解RNA编辑与人类疾病相关的机制。该工具将对RNA编辑研究产生重要影响。DeepEdit可以稳健地检测来自不同物种(包括酿酒酵母和人类)的纳米孔RNA测序读取中的A-to-I编辑事件,使我们能够分析编辑事件的相位,并获得有关RNA的转录后调控的新见解。

























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








