Title:MuLan-Methyl - Multiple Transformer-based Language Models for Accurate DNA Methylation Prediction
期刊: bioRxiv (Cold Spring Harbor Laboratory)
文章链接:http://ab.cs.uni-tuebingen.de/software/mulan-methyl/
dataset:http://lin-group.cn/server/iDNA-MS/download.html
code:https://github.com/husonlab/mulan-methyl
摘要
基于transformer的语言模型成功地用于处理大量与文本相关的任务。DNA甲基化是一种重要的表观遗传机制,其分析为基因调控和生物标志物鉴定提供了有价值的见解。已经提出了几种基于深度学习的方法来识别DNA甲基化,每种方法都试图在计算工作量和准确性之间取得平衡。在这里,我们介绍了MuLan-Methyl,这是一种用于预测DNA甲基化位点的深度学习框架,它基于多个(五个)流行的基于transformer的语言模型。该框架确定了三种不同类型的DNA甲基化的甲基化位点,即n6 -腺嘌呤(6mA), n4 -胞嘧啶(4mC)和5-羟甲基胞嘧啶(5hmC)。使用的五种语言模型中的每一种都适用于使用“预训练和微调”范式的任务。预训练是在使用自监督学习的自定义语料库上进行的,语料库由DNA片段和分类谱系组成。然后微调的目的是预测每种类型的dna甲基化状态。这五个模型被用来共同预测DNA甲基化状态。我们报告了MuLan-Methyl在基准数据集上的出色性能。此外,我们表明,该模型捕捉了与甲基化相关的不同物种之间的特征差异。这项工作证明了语言模型可以成功地适应这一应用领域,并且联合使用不同的语言模型可以提高模型的性能。
数据集
该数据集包含三种主要类型的DNA甲基化位点——6mA、4mC和5hmC——涉及12个基因组(一种细菌和11种真核生物),共有250,599个阳性样本。此外,该数据集提供了 阴性样本相同数量的非甲基化序列。对应的数据集整理如下:
6mA | 4mC | 5hmC | |
T. thermophile | 53800 | ||
A. thaliana | 15937 | ||
H. sapiens | 9168 | ||
Xoc. BLS256 | 8608 | ||
D. melanogaster | 5596 | ||
C. elegans | 3981 | ||
C. equisetifolia | 3033 | ||
S. cerevisiae | 1893 | ||
Tolypocladium | 1690 | ||
F . vesca | 1551 | ||
R. chinensis | 300 | ||
C. equisetifolia | 183 | ||
F . vesca | 7899 | ||
S. cerevisiae | 990 | ||
Tolypocladium | 7664 | ||
M. musculus | 1840 | ||
H. sapiens | 1172 |
方法
数据的长度都是41,采取滑动窗口为6的句子中。
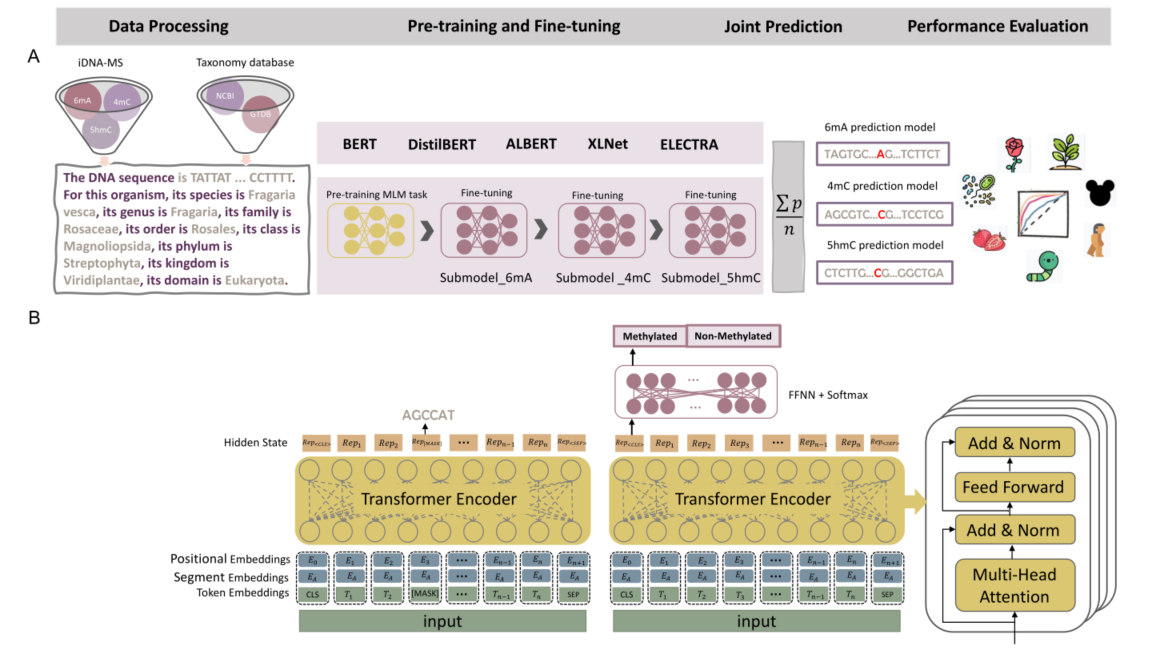
我们的方法使用了五种基于transformer的语言模型,我们将在下面介绍它们。
(1) BERT:能够使用去噪和基于自动编码的预训练对双向上下文建模。BERTbase的变压器结构采用12层编码器堆栈,768个前馈网络隐藏单元和12个注意头;共110M参数。
(2)DistilBERT:通过减少层数,可以得到BERT的。它的大小是BERT的40%,速度快60%,而精度只低3%。
(3) ALBERT:对12个变压器编码器块采用了跨层参数共享技术,并在词汇表和隐藏层之间引入嵌入分解,以减小BERT的参数大小。
(4) XLNet:采用了创新的预训练步骤;它的广义自回归预训练方法能够通过最大化所有因子分解顺序排列的期望似然来学习双向上下文,克服了BERT忽略隐藏位置之间的依赖关系所引起的问题。
(5)ELECTRA:与其他架构相比,ELECTRA训练两种变压器模型;生成器替换序列中的令牌,判别器尝试识别哪些令牌被生成器替换,而不是在MLM任务上进行训练。
结果
与现有方法比较iDNA-ABF和 iDNA-ABT

位点预测性能
6mA

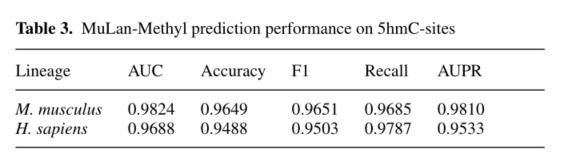
4mC
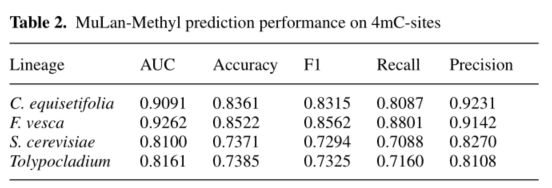
5hmc
结论
MuLan-Methyl中的每个子模型都在训练数据集上进行预训练和微调,然后它们在独立的测试数据集上集体预测甲基化位点。用多种指标评价了木兰甲基的性能,并与现有的两种方法进行了比较,结果表明该方法表现出非常好的性能。
我们的研究还表明,在预训练步骤中具有增强算法的模型,如XLNET,以及具有更少参数和更少内存消耗的模型,如ALBERT,在存储和计算资源有限的情况下比BERT更合适。
与其他生物域适应语言模型相比,我们训练木兰-甲基的自定义语料库包含多模态数据,包括来自DNA- ms的DNA序列和来自NCBI和GTDB分类法的文本格式的分类学谱系。据我们所知,MuLan-Methyl是第一个考虑分类法信息的语言模型框架。
这提高了模型的准确性和特征贡献分析。MuLanMethyl发现的DNA甲基化基序很大程度上得益于变压器结构的自注意机制。此外,DNA序列对分类学谱系的重视权重有助于分析核苷酸序列与分类学谱系的关系。
以前的方法为每个分类谱系和每个甲基化位点类型构建单独的分类器,为这里使用的数据产生17个不同的分类器。相比之下,MuLan-Methyl将分类谱系视为一个特征,因此只产生三个分类器,每个甲基化位点类型一个。
总之,我们提出了一个框架,该框架集成了五种流行的NLP方法来解决一个重要的生物学问题。MuLan-Methyl能够可靠地检测来自已知分类谱系的DNA序列的DNA甲基化位点,性能略好于目前最先进的方法。
这项研究表明,当人们想要将基于变压器的语言模型适应特定领域时,BERT并不是唯一的选择,还应该考虑它的变体。
结果表明,多语言模型的集成可以在一定程度上弥补个体模型的不足,从而获得更好的集成预测性能。






















 914
914











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








