点击下方“ReadingPapers”卡片,每天获取顶刊论文解读
论文信息
题目:LKM-UNet: Large Kernel Vision Mamba UNet for Medical Image Segmentation
LKM-UNet: 大型内核视觉 Mamba UNet 用于医学图像分割
作者:Jinhong Wang, Jintai Chen, Danny Chen, Jian Wu
源码链接:https://github.com/wjh892521292/LKM-UNet
本文创新点
提出了大型内核 Mamba U 形网络 (LKM-UNet):作者提出了一种新的基于 Mamba 的 UNet 模型,用于 2D 和 3D 医学图像分割。这种模型利用 Mamba 的强大序列建模能力和线性复杂性,通过为 SSM 模块分配大内核来实现大感受野。
设计了新颖的层次化和双向大型内核Mamba块(LM块):这种设计增强了 SSM 在视觉输入中的全局和邻域空间建模能力。特别是,LM 块包含**像素级 SSM (PiM)和块级 SSM (PaM)**,这两者共同工作以增强局部邻域像素级和长距离全局块级建模。
引入了双向 Mamba (BiM):与原始的单向 Mamba 相比,作者提出的双向 Mamba 结构通过同时进行前向和后向扫描并叠加输出结果,改进了位置感知序列建模。这种双向设计使模型能够更好地关注图像中心区域的信息块,而不是仅仅关注角落区域,并且能够更好地建模每个块的绝对位置信息和与其他块的相对位置信息。
摘要
在临床实践中,医学图像分割提供了有关目标器官或组织的轮廓和尺寸的有用信息,有助于改善诊断、分析和治疗。在过去几年中,卷积神经网络(CNN)和 Transformers 在这一领域占据主导地位,但它们仍然受到有限的感受野或昂贵的长距离建模的限制。Mamba,一种状态空间序列模型(SSM),最近作为长距离依赖建模的有前途的范式出现,具有线性复杂性。在本文中,我们介绍了一种大型内核视觉 Mamba U 形网络,或称为 LKM-UNet,用于医学图像分割。我们 LKM-UNet 的一个区别特征是其使用大型 Mamba 内核,与基于小内核的 CNN 和 Transformers 相比,在局部空间建模方面表现出色,同时保持了与二次复杂性的自注意力相比的优越效率。此外,我们设计了一种新颖的层次化和双向 Mamba 块,以进一步增强 Mamba 对视觉输入的全局和邻域空间建模能力。综合实验表明,使用大尺寸 Mamba 内核实现大感受野是可行且有效的。
关键词
医学图像分割 · UNet · Mamba
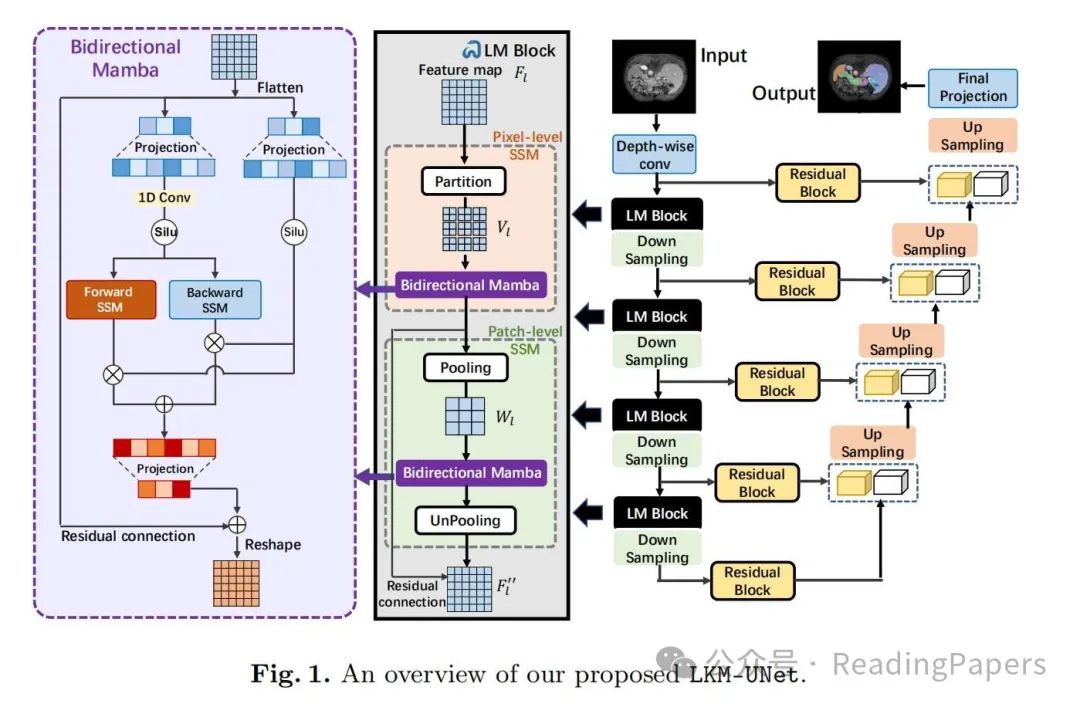
方法
在本节中,我们首先介绍 LKM-UNet 的整体架构。随后,我们详细阐述了核心组件,LM 块。
3.1 LKM-UNet
LKM-UNet 的概述如图 1 所示。具体来说,除了常见的 UNet 组成,包括深度可分离卷积、编码器下采样层、解码器上采样层和跳跃连接外,LKM-UNet 通过在编码器中插入提出的大内核 Mamba(LM)块来改进 UNet 的结构。给定一个分辨率为 C × D × H × W 的 3D 输入图像,深度可分离卷积首先将输入编码成特征图 F0 ∈ R48× D/2 × H/2 × W/2。然后特征图 F0 被送入每个 LM 块和相应的下采样层,并获得多尺度特征图。一个LM 块包含两个 Mamba 模块:像素级 SSM(PiM)和块级 SSM(PaM)。对于第 l 层,过程可以公式化为:
其中 PiM 和 PaM 分别表示像素级 SSM 和块级 SSM。Down-sampling 表示下采样层。在每个阶段之后,生成的特征图 被编码为 ,其中 表示特征图 的通道和分辨率。至于解码器部分,我们采用了 UNet 解码器和带有跳跃连接的残差块进行上采样和预测最终的分割掩码。

3.2 LM 块
LM 块是用于进一步空间建模每个阶段不同尺度的特征图的核心组件。与之前使用 CNN 进行局部像素级建模和 Transformer 进行长距离块级依赖性建模的方法不同,LM 块可以同时完成像素级和块级建模,得益于 Mamba 的线性复杂性。更重要的是,较低的复杂性允许设置更大的内核(窗口)以获得更大的感受野,这将提高局部建模的效率,如图 2(a) 所示。具体来说,LM 块是一个层次化设计,由像素级 SSM(PiM)和块级 SSM(PaM)组成;前者用于局部邻域像素建模,后者用于全局长距离依赖性建模。此外,LM 块中的每个 Mamba 层都是双向的,这是为了位置感知序列建模而提出的。像素级 SSM(PiM)。由于 Mamba 是一个连续模型,输入像素的离散性质可能会削弱局部相邻像素之间的相关性建模。因此,我们提出了一个像素级 SSM,将特征图分割成多个大子内核(子窗口)并对子内核进行 SSM 操作。我们首先将整个特征图等分为 2D 的非重叠子内核或 3D 的子立方体。以 2D 为例,给定一个 H × W 分辨率的输入,我们将特征图分割成大小为 m×n 的子内核(m 和 n 可以高达 40)。不失一般性,我们假设 H/m 和 W/n 都是整数。然后我们有 HW/mn 个子内核,如图 1 中的像素级 SSM 所示。在这种方案下,当这些子内核被送入 Mamba 层时,局部相邻像素将连续输入到 SSM;因此,局部邻域像素之间的关系可以更好地建模。此外,在大内核分割策略下,感受野被扩大,模型可以获得更多局部像素的细节。然而,图像被分割成非重叠的子内核。因此,我们需要一个机制来实现不同子内核之间的通信,以进行长距离依赖性建模。块级 SSM(PaM)。我们引入了一个块级 SSM 层,以在不同的子内核之间传递信息。如图 1 中的块级 SSM 所示,一个分辨率为 H × W 的特征图 首先通过一个大小为 m × n 的池化层,允许每个 HW/mn 个子内核的重要信息被总结成一个代表。因此,我们获得了具有 HW/mn 个代表的聚合图 ,然后这些聚合图被用来通过 Mamba 在子内核之间进行全局范围的依赖性建模。在 Mamba 中的交互之后,我们将聚合图反池化回与初始特征图 相同的大小,并应用残差连接。 在方程(5)中的过程可以执行为:
其中 Pooling 和 Unpooling 分别表示池化层和反池化层。Bi-Mamba 表示提出的双向 Mamba 层。双向 Mamba(BiM)。与基于前向扫描方向 SSM 层的原始 Mamba 块不同,LM 块中的每个 SSM 层(包括 PiM 和 PaM)都是双向的。图 2(b) 显示了差异。在原始 Mamba 中,作为一个连续模型,一些信息遗忘发生在早期进入的元素上,而最后进入 Mamba 的元素将保留更多的信息。因此,具有单一扫描方向的原始 Mamba 将更多地关注后部块,而不是通常包含更多器官和病变的特征图的中心区域。为此,我们提出了一个双向 Mamba 结构,通过同时进行前向和后向扫描并叠加输出结果。图 1 的左部分显示了详细结构。BiM 有两个优点。首先,模型可以更多地关注图像中心区域的信息块,而不是角落区域。其次,对于每个块,绝对位置信息和与其他块的相对位置信息可以被网络很好地建模。
实验

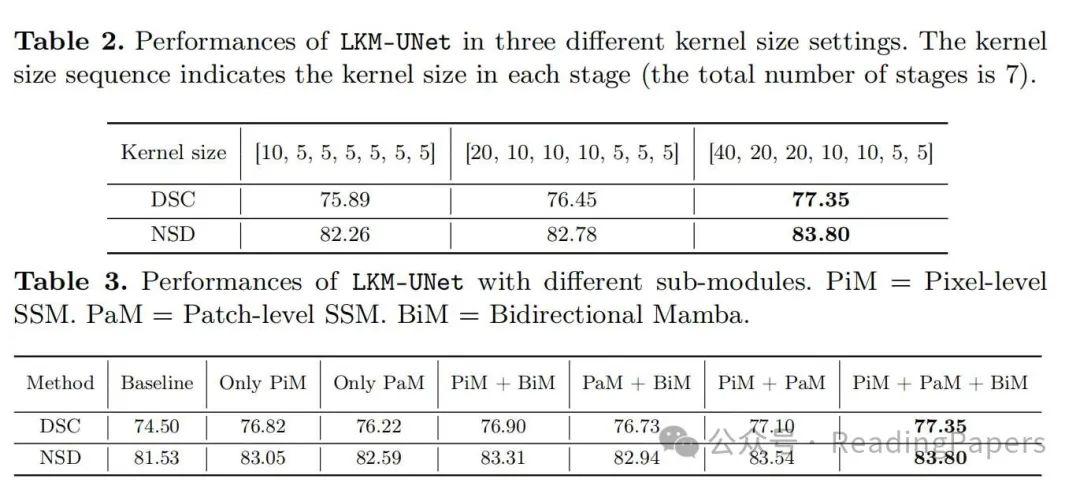
声明
本文内容为论文学习收获分享,受限于知识能力,本文对原文的理解可能存在偏差,最终内容以原论文为准。本文信息旨在传播和学术交流,其内容由作者负责,不代表本号观点。文中作品文字、图片等如涉及内容、版权和其他问题,请及时与我们联系,我们将在第一时间回复并处理。
























 2674
2674

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








