目录
1. 声明
本文是关于医学图像处理的,针对的是nii文件,主要是如何将nii文件转换成我们常见的数据格式,目前当然是numpy数据。本人并不是医学相关人员,只是需要用到nii文件,写这篇文章也是相当于个人笔记了,以防遗忘,文中的很多观点仅是个人理解,请不要上纲上线,若有错误,请专业人员批评指正!
2. nii文件简介
nii文件是常见的医学图像格式,常规的照片查看器无法打开,本人常用的软件是ITK-SNAP,这个除了可以查看文件的信息,还能画标签,比较方便。在python中有一个相关的库SimpleITK,提供了医学图像相关的很多功能,可以方便的处理和查看各种医学图像格式。本节主要介绍一些nii文件相关的属性以及其含义。
2.1 灰度值范围
在ITK-SNAP中打开nii文件,如图所示,在对比度选项中可以查看图像的灰度值分布。不同的机器,不同的器官拍出来的图像灰度值范围都是不一样的,有的甚至可以是 -几十万 至 +几十万,我们常见的灰度值范围是0~255,而且为了突出我们想要观察的器官区域,使其与背景的对比度增加,一般会进行灰度值变换,这在后面会单独一节介绍。
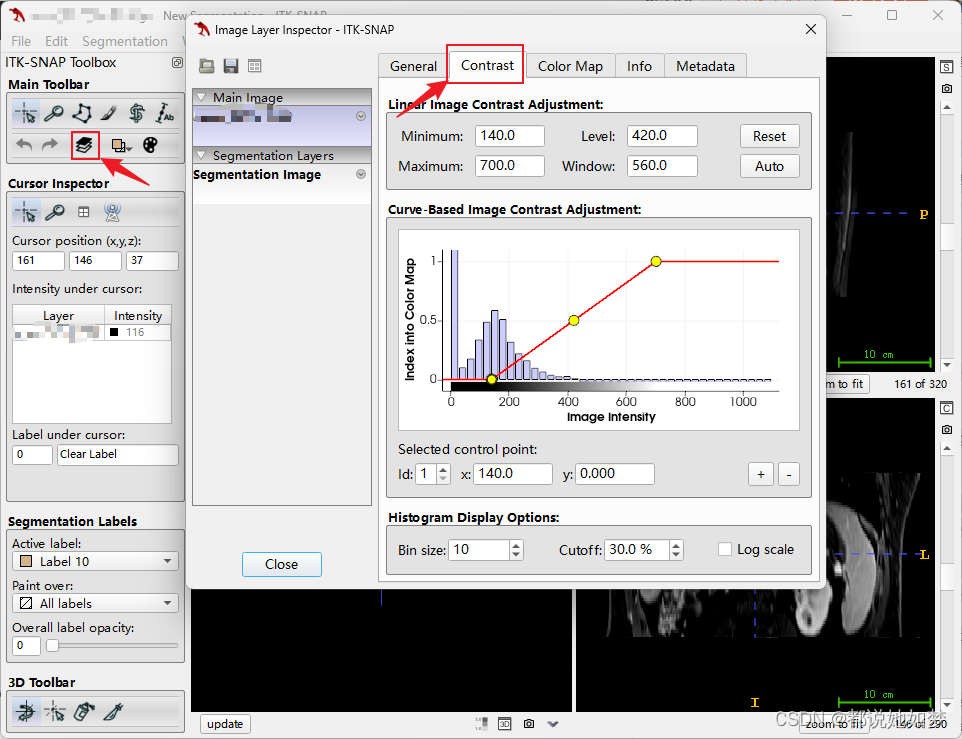
2.2 Dimensions
dimensions参数表示的是图像的维度大小,如图所示,在info选项中可以查看详细信息。这里需要注意的是,nii文件中的坐标轴与numpy中的顺序是相反的,这里的x,y,z分别表示的横轴,纵轴与深度轴,而在numpy中表示的是C×H×W,也就是说顺序是z,y,x。如果是使用的SimpleITK中的GetArrayFromImage函数直接转换的,那么得到的numpy数组会自动转换为z,y,x顺序。
2.3 Orientation
Orientation参数的字面意思是朝向,这是与真实的三维空间对应的,医学图像一般是扫描的3D数据,分别规定了病人在三个垂直轴上的方向,也就是说以病人为中心,有以下6个方向:
- superior-inferior -> 头到脚
- anterior-posterior -> 正脸到后脑勺
- right-left -> 右半边到左半边
所以说S/I, A/P, R/L这三对方向每一对选取一个就可以组成一个坐标系,选定之后这三个方向就分别与+x, +y, +z三个轴对应。例如常见的组合有RPI, LAS等,RPI就表示+x指向病人的右边,+y指向病人的后背,+z指向病人的脚,LAS类推。
更详细的关于nii文件方向系统的解释可以参考这篇文章:
nii文件中的方向理解
2.4 Direction
Direction参数具体含义不太好理解,中文网络上目前也没有找到合适的介绍,所以仅仅从使用的角度来做解释。Direction参数是一组向量,常见的向量值是:[1,0,0,0,1,0,0,0,1],每三个值看做一个方向向量,分别代表x,y,z轴在我们常见的图像坐标系的方向坐标。在numpy和我们的常识中,图像的第一个像素从左上角开始,然后做左往右从上往下依次排列 ,往每一列的方向看是1轴,往每一行的方向看是2轴,往每一个通道(切片)的方向看是0轴。而direction向量的第1-3,4-6,7-9个元素组成的三个坐标分别对应2,1,0三个轴(因为numpy与nii中的轴顺序相反,所以对应也是相反的)。
所以Direction怎么用,答案是判断读取的numpy数组是否需要翻转,标准的Direction值是[1,0,0,0,1,0,0,0,1],也就是说图像的实际方向与常见的坐标轴是一致的,不用翻转,如果第1,5,9个元素出现了负值,那么说明是负值的元素所代表的轴方向与实际相反,在numpy中就需要翻转该轴。例如:现在有一个Direction值为[-0.9,0,0,0.01,-1,0.1,0,0,1],那么在numpy数组中需要将1,2轴翻转,而0轴保持不变,使用flip函数完成即可。
代码会在后面给出。
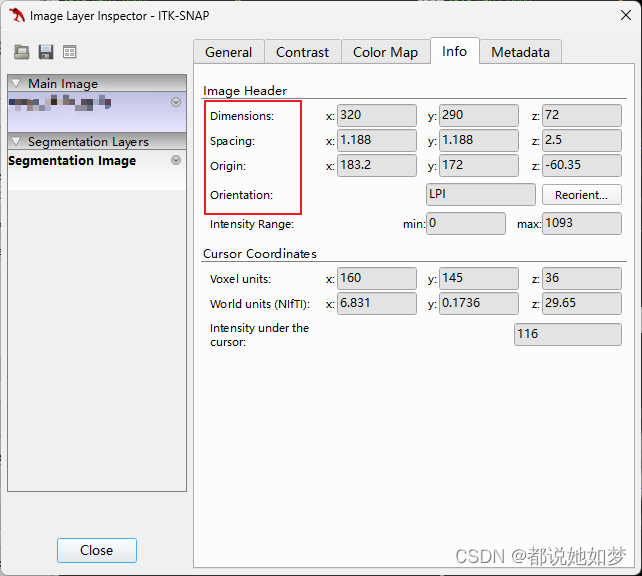
2.5 Origin
该参数在实际使用中用处不多,了解即可,表示的是图像的起点的坐标,我么处理数据的时候一般正确获取数据值就行,可以不用关注这个参数。
2.6 Spacing
这是一个非常重要的参数,决定着图像是否需要重采样,它也是由三个值组成,对应的是x,y,z三个轴,表示各个轴上一个像素所代表的的实际物理距离,单位一般是mm。在ITK-SNAP中如果放大图像会发现,有的图像的最小单元——一个像素的形状不是正方向,而是长方形的,这就是因为不同轴之间的Spacing值不同造成的,我画了一个图来解释:
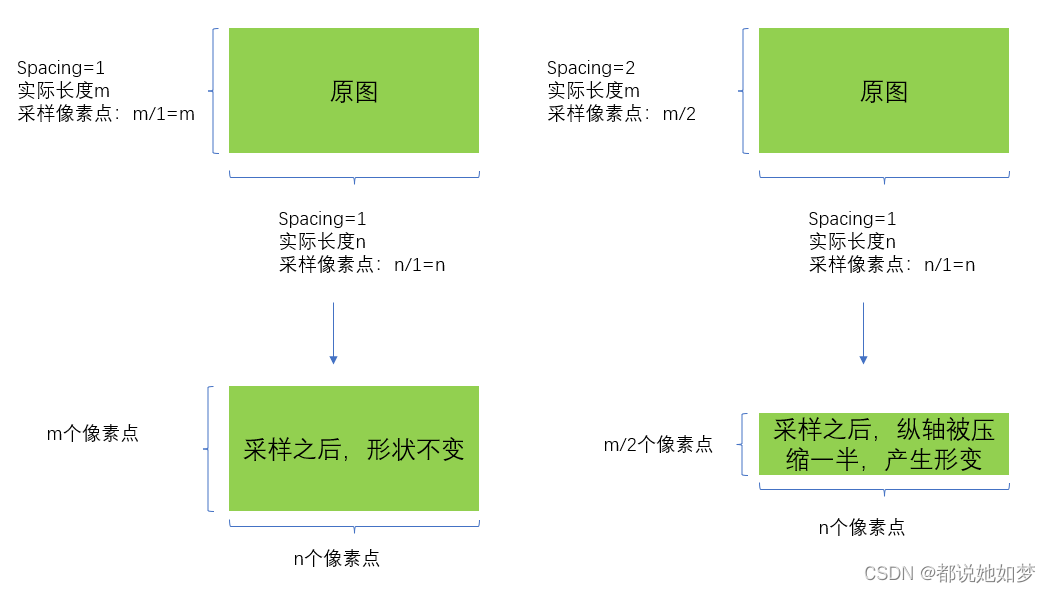
这里仅仅画了两个轴,三个轴的原理一样,当各个轴的Spacing值一样时,采出来的像素是一个正方形,也就是说采样图像与原图比例是一致的,不会变形。如果Spacing值不同,为了获取图像数据就不能直接读出numpy数组了,需要进行重采样,否则会产生形变。重采样一般是用线性插值的方式,在SimpleITK中有ResampleImageFilter类专门来处理,具体代码在后面章节给出。
需要注意的是,重采样有时候是必要的,但是毕竟插入的值是被计算出来的,总带有误差,所以个人认为能不重采样就不要做(可能是个人迷信)。例如,现在有一个图像大小是512×256,Spacing分别是1,2,那么我会在第二个轴上插值即可,最后得到512×512的图像。
3. 方向处理
这里给出上面提到的处理Direction参数的代码,一般在获取到numpy数组之后就立马处理。
import SimpleITK as sitk
import numpy as np
image=sitk.ReadImage('xxx.nii')
direction=image.GetDirection()
array=sitk.GetArrayFromImage(image)
if direction[0] < 0:
np.flip(array, axis=2)
if direction[4] < 0:
np.flip(array, axis=1)
if direction[8] < 0:
np.flip(array, axis=0)
4. 灰度值变换
前面提到了,nii图像的灰度值变化范围一般范围很大,我们只需要关注某一个特定的区域,所以需要调节对比度使该区域更加明显,在SimpleITK中提供了处理函数:
sitk.IntensityWindowing(img, window_min, window_max)
这里涉及了两个概念,窗宽和窗位,窗宽就是在实际灰度值范围中你选择映射的最大值和最小值之间的宽度,窗位就是最大值和最小值中点的灰度值。
如果使用SimpleITK中的函数,需要注意的是传入的img是Image对象,而不是numpy数组,然后直接传入最大和最小值即可,返回的也是Image对象,值是映射到0~255的灰度值。
此外,如果不用这个函数,也可以很容易自己实现一个映射函数,传入numpy数组,返回的也是0~255的numpy数组:
def gray_affine(img, max_value, min_value):
img = (img - min_value) / (max_value - min_value)
img[img < 0] = 0
img[img > 1] = 1
return (img * 255).astype(np.uint8)
这两种方法本人都试验过,绝大部分的像素值都是相等的,只有少数像素值相差1,应该是最后四舍五入的方式不一样。
5. 重采样
对于重采样,SimpleITK中也有专门的类来处理,但是这个类也是针对Image对象的,而且会对所有的切片都做处理,我们一般都只需要处理一个nii文件中的部分切片,是针对numpy的,有时候只需要用到一百多张切片中的2~3张,使用这个类还是会处理所有切片,所以个人感觉不那么完美(有点强迫症)。最后自己也简单写了一个切片插值方式的程序,不知道是不是与SimpleITK中的处理方式一样,但是目前输进网络中训练没有发现明显在的问题。
ResampleImageFilter()
这个类别可以自由设置重采样之后Spacing,像素尺寸的一系列参数,网上有很多使用案例,这里给出一个个人认为比较好的:
def resize_sitk_3D(image_array, outputSize=None, interpolator=sitk.sitkLinear):
"""
Resample 3D images Image:
For Labels use nearest neighbour
For image use
sitkNearestNeighbor = 1,
sitkLinear = 2,
sitkBSpline = 3,
sitkGaussian = 4,
sitkLabelGaussian = 5,
"""
image = image_array
inputSize = image.GetSize()
inputSpacing = image.GetSpacing()
outputSpacing = [1.0, 1.0, 1.188]
if outputSize:
outputSpacing[0] = inputSpacing[0] * (inputSize[0] / outputSize[0])
outputSpacing[1] = inputSpacing[1] * (inputSize[1] / outputSize[1])
outputSpacing[2] = inputSpacing[2] * (inputSize[2] / outputSize[2])
else:
# If No outputSize is specified then resample to 1mm spacing
outputSize = [0.0, 0.0, 0.0]
outputSize[0] = int(inputSize[0] * inputSpacing[0] / outputSpacing[0] + .5)
outputSize[1] = int(inputSize[1] * inputSpacing[1] / outputSpacing[1] + .5)
outputSize[2] = int(inputSize[2] * inputSpacing[2] / outputSpacing[2] + .5)
resampler = sitk.ResampleImageFilter()
resampler.SetSize(outputSize)
resampler.SetOutputSpacing(outputSpacing)
resampler.SetOutputOrigin(image.GetOrigin())
resampler.SetOutputDirection(image.GetDirection())
resampler.SetInterpolator(interpolator)
resampler.SetDefaultPixelValue(0)
image = resampler.Execute(image)
return image
需要格外注意的是,如果是对图像重采样,要用sitk.sitkLinear,也就是线性插值,如果是对标签重采样,一定要用sitk.sitkNearestNeighbor,也就是最近领插值,也就是要保证标签中不能出现新的标签值。
直接给切片插值
def resample(slice_id, img, label, spacing):
imgs, labels = [], []
for s in slice_id:
img_slice = img[s]
label_slice = label[s]
assert img_slice.shape == label_slice.shape
if spacing[0] != spacing[1]:
resize_x, resize_y = img_slice.shape[1], int(img_slice.shape[0] * (spacing[1] / spacing[0]))
img_slice = cv2.resize(img_slice, (resize_x, resize_y), interpolation=cv2.INTER_LINEAR)
label_slice = cv2.resize(label_slice, (resize_x, resize_y), interpolation=cv2.INTER_NEAREST)
s0, s1 = img_slice.shape[0], img_slice.shape[1]
# 填充成正方形后再插值,防止形变
if s0 != s1:
max_shape = max(s0, s1)
temp1 = np.zeros((max_shape, max_shape))
temp2 = np.zeros((max_shape, max_shape))
if s0 > s1:
single_width = (s0 - s1) // 2
temp1[:, single_width:single_width + s1] = img_slice
temp2[:, single_width:single_width + s1] = label_slice
elif s0 < s1:
single_width = (s1 - s0) // 2
temp1[single_width:single_width + s0, :] = img_slice
temp2[single_width:single_width + s0, :] = label_slice
img_slice, label_slice = temp1, temp2
if img_slice.shape != (256, 256):
img_slice = cv2.resize(img_slice, (256, 256), interpolation=cv2.INTER_LINEAR)
label_slice = cv2.resize(label_slice, (256, 256), interpolation=cv2.INTER_NEAREST)
imgs.append(img_slice)
labels.append(label_slice)
return imgs, labels
传入的slice_id是需要处理的切片索引,img,lable都是numpy数组,spacing是nii文件的Spacing参数。这里除了按照spacing比例插值之外,还将图像尺寸resize到了想要的大小,因为深度学习输入的图像大小一般是正方形,所以需要上下采样到指定大小(这里是256),而有些时候原始图像大小是长方形,直接采样会变形,所以这里采用了填充的方式,用0将较短的边补齐,然后再采样。
6. 说在最后
这是第一次在csdn上正式写文章,初衷还是记录一下学习的知识点,希望以后可以更多地记录一些学习的过程吧!




















 1794
1794











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








