X-MOL: large-scale pre-training for molecular understanding and diverse molecular analysis
目录
1.1 Molecular property prediction (classification)
1.2 Molecular property prediction (regression)
1.3 Chemical reaction productivity prediction
1.4 Drug-drug interaction prediction
3. Visualization and Interpretation
总结
作者提出了X-MOL,在11亿个分子的SMILES序列进行预训练。在微调阶段,作者将X-MOL进行了微调,以适应分子性质预测、化学反应分析、药物-药物相互作用预测、从头分子生成和分子优化五种不同的任务。此外,作者还进行了三种知识嵌入(链接嵌入、环嵌入、类型嵌入)的探索。但是结果表明,所有的嵌入策略都不能提高模型在相应任务中的性能。相反,这些额外的信息可能会使模型的学习更加困难,都会在一定程度上降低模型性能。
一、Introduction
作者探讨了基于SMILES的大规模预训练模型在分子表示和理解中的作用,以及它对下游分子分析任务的促进作用。研究两个基本问题。
- 利用大量的无标记数据进行预训练和强大的计算能力,学习和理解基于SMILES的分子表示的可行性。
- 应用这种预训练技术来促进多样化的下游分子分析任务。为此,作者开发了一个通用的分子表示学习和理解框架X-MOL,通过在百度PaddlePaddle平台下使用11亿分子训练数据和强大的计算能力进行大规模预训练,促进全面的分子分析任务,包括分子性质预测、化学反应分析、药物-药物相互作用预测、从头分子生成和分子优化。
二、Method
BERT、RoBERTa、ERNIE和XLNET等自监督模型通过对大规模文本语料库的预训练和对下游任务的微调,在自然语言处理中取得了显著的进步。预训练的关键是使模型理解语义信息的方法。在自然语言处理的预训练中,经常使用掩码语言模型(MLM) 和排列语言模型(PLM) 训练模型来学习整个句子的语义。作者在X-MOL中设计了一个特定的生成式预训练策略来表示和理解SMILES。
作者提出了一种生成式预训练策略:训练预训练模型,以给定的SMILES作为输入,生成同一分子的另一个有效SMILES。一旦模型能够执行这一任务,作者认为该模型不仅可以通过SMILES重构分子结构,还可以根据分子结构生成有效的SMILES表示。在X-MOL中,作者提出了一种融合了双向注意力机制与单向注意力机制的混合注意力Transformer模型(图1b),使得X-MOL在一个Transformer encoder模型上实现了Encoder-Decoder结构的效果,达到了小分子生成的目的。

三、Results
作者将下游任务分成了两类:预测任务和生成任务(图1b-d)。对于预测任务,作者总结了不同任务的具体输入格式和损失函数。对于生成任务,总结了不同任务的生成源和采样策略(表1)。

1. Prediction tasks
1.1 Molecular property prediction (classification)
分子性质预测,包括分子亲和性和ADME/T预测。在这项任务中,作者在MoleculeNet中选择了四种具有少量数据集(HIV、BACE、BBBP、ClinTox)的分类来测试X-MOL的性能。采用ROC-AUC来评估模型的性能。对于每个分类,数据被随机分成20次训练和测试数据集,平均性能为X-MOL在该特定分类上的最终性能。
结果表明,X-MOL在四个分类数据集上完全超越了现有的方法,并达到了最高的ROC-AUC值(图2a,b,c,d)。特别是,对于BBBP和ClinTox数据集,它明显优于现有的最佳模型(图2c,d)。值得注意的是,尽管基于图的分子表示有望比SMILES表示更好地捕捉分子结构,但X-MOL仍然优于基于图的分子表示模型GC (Graph convolutional model),这表明对SMILES表示的大规模预训练可以很好地理解分子结构,并且能与基于图的分子表示相当。

1.2 Molecular property prediction (regression)
分子性质预测也可以表述为带有数值标签的回归任务。作者在MoleculeNet中划分了三个基准回归数据集(ESOL、FreeSolv、Lipophilicity )和几个子数据集,以测试X-MOL在基于回归的分子性质预测方面的性能。采用均方根误差(RMSE)来评估模型的性能。结果表明,在三个回归数据集上,X-MOL完全优于现有的方法,并获得了最优秀的RMSE值(图2e,f,g)。
1.3 Chemical reaction productivity prediction
化学反应产率预测是化合物生产和药物合成领域的一项重要计算任务。与分子亲和力或性质的预测不同,这项任务的输入由几个部分组成,参与或影响化学反应的反应物、反应环境、催化剂等。采用3956个化学反应的生产率数据集,并使用随机森林进行化学反应生产率预测。作者设计了一种微调策略,以四种SMILES表示作为输入来表示化学反应的四个部分,预测的产率是输出。结果,X-MOL的平均RMSE为0.0626,显著低于随机森林的值0.078(图3a)。

1.4 Drug-drug interaction prediction
药物-药物相互作用(DDIs)可能在人体中会引起一系列意想不到的药理作用,包括一些未知的药物不良事件(ADEs)机制。作者采用经典计算模型DeepDDI作为基准,192284个DDI预测数据集,并基于所设计的DDI表示提出了一种用于DDI预测的多分类深度学习模型。作者设计了一种微调策略,以代表两种药物的两个SMILES作为输入,以这些药物对属于每一类的概率(例如吸收的增加或减少)作为输出。结果表明,X-MOL的平均准确度(ACC)为0.952,高于基线DeepDDI的0.924值(图3b)。
2. Generation tasks
从头分子生成任务可以分为两类:基于分布学习(DL)的生成和目标导向(GD)的生成。
- 基于DL生成:需要模型生成与给定训练数据中的分子相似的分子。通常是在训练过程中,模型学习训练数据的内部分布,然后对学习到的空间进行采样,获得新的分子。
- 基于GD生成:需要模型生成满足某些预定义要求(分子量、QED、logP)的分子。模型需要学习训练数据的标签与数据分布之间的对应关系,然后在生成过程中根据预定义的目标进行采样和生成分子。
2.1 Molecule generation (DL)
通常,基于Graph的生成模型被认为在分子生成任务中比基于SMILES的模型表现更好,因为基于Graph的表示明确地捕获了分子结构信息。尽管如此,作者表明通过利用大规模预训练技术,基于SMILES表示的X-MOL能够在从头分子生成中获得类似或优于现有基于Graph模型(JT-VAE、GCPN、MRNN、GraphNVP和GraphAF)的表现。结果,X-MOL在三个指标(有效性、独特性、新颖性)超越了现有的基于Graph的分子生成方法。特别是,X-MOL在有效性方面大大超过了其他方法(表2a)。结果表明,经过预训练的X-MOL能够很好地学习和理解SMILES语法,因此,它可以比现有的方法生成更多的有效分子。


2.2 Molecule generation (GD)
在GD生成任务中,主要关注生成的分子是否满足预定义的要求,根据所需属性的平均值以及生成的分子偏离目标值来评估该任务的性能。作者使用zinc250k数据集作为训练集,以QED作为目标属性微调X-MOL。训练结束后,设定QED = 0.948为生成目标,因为0.948是训练集中的最大QED, 对X-MOL生成10000个新分子进行评估。与现有的基于Graph表示的生成模型相比,检查生成的前3个分子时,X-MOL生成了所有这些分子,其设计QED值为0.948,优于JT-VAE、GCPN、MRNN和GraphAF等模型(表2b)。前五个分子如表2c所示。
2.3 Molecule optimization
作者研究了大规模的预训练和微调技术如何提高分子优化的最终性能。因此,将X-MOL与冷启动(cold-start)模型(具有与X-MOL相同的模型架构和参数,无预训练或微调)进行比较。结果表明,在分子优化过程中,X-MOL和冷启动模型都有助于提高目标分子的性能。然而,X-MOL除新颖性外,在所有方面都优于冷启动模型(图4c)。
- X-MOL在训练和测试中表现出更高的鲁棒性和稳定性(图4a)。从图4b中可以看出,由于预训练的影响,X-MOL优化后的分子与原始分子的相似度很快达到了很高的水平,但相似度逐渐降低,这与冷启动模型的性能完全相反。
- 在新颖性方面,X-MOL与冷启动模型相比存在不足,这是生成式模型的必然结果。生成模型在训练过程中记忆训练样本。因此,X-MOL得到的优化分子的新颖性低于冷启动模型(图4c)。
- 由于X-MOL在预训练阶段对SMILES的语法规则有较好的理解,因此在整个优化过程中都能保持较高的有效性。尽管冷启动模型在优化初期迅速提高了有效性,但与微调后的X-MOL相比,最终收敛仍存在差距(图4d)。

3. Visualization and Interpretation
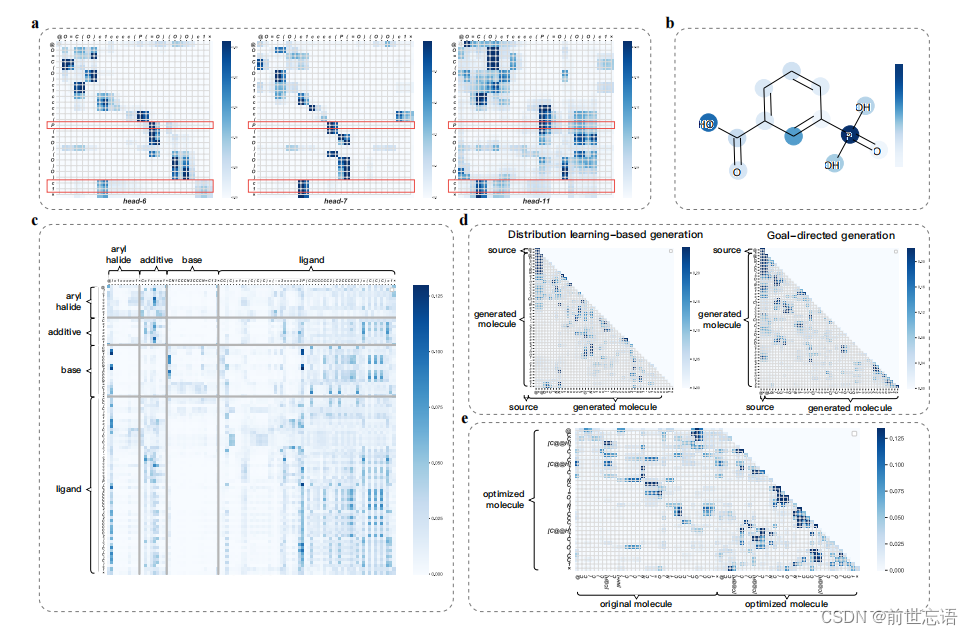
作者还研究了X-MOL在微调阶段采用的注意力机制,能够直观地检查不同层对SMILES不同部位的注意。这种注意力机制的可视化和解释能帮助理解X-MOL如何根据不同分子分析任务的SMILES表示来理解分子。
研究O=C(O)c1cccc(P(=O)(O)O)c1分子在HIV数据集的分子性质预测。学习这个分子所需的注意力权重被可视化为热图,颜色的亮度表示注意力的强度。热图的纵坐标根据SMILES表示表示输入分子中的每个字符,横坐标表示所涉及的每个字符。以SMILES表示中两个原子连接环境最复杂的字符P和最后一个c1为例,可以看出,在模型的中间层(第9层),该方法可以正确识别出P和c1的环境。对于P,头6识别了连接O的双键,头7识别了连接苯环的c,头11识别了连接O的两个单键。对于c1,头6和头7都注意到对应的开环c1,而头11除了识别开环c1外还识别了一个连接在苯环上的C。因此,X-MOL可以正确地识别两种复杂原子连接环境(图5a)。然后,模型进一步对分子的各个部分进行抽象,以显示其更高层次的特征,如后面几层所示,最后根据对分子不同部分的注意程度进行属性预测(图5b)。总之,这个例子清楚地表明,X-MOL能够重建非线性分子结构,并学习其高级特征,以线性SMILES表示的性质预测。
如图5d所示,对于不同的生成任务,右上角的注意力可视化通常是黑暗的。然而,不同的任务在注意力方面有具体的区别。在基于DL的分子生成中,模型在训练过程中学习目标分子的生成空间,新生成的字符更加关注自身生成的部分。在基于GD的分子生成中,模型在生成新的分子时,既要满足SMILES的语法规则,又要满足生成目标,这体现在注意力可视化上,即新生成的字符需要同时注意源和先前生成的部分。最后,在分子优化中,如图5e所示,可以看出模型在生成的早期更加关注原始分子,而在优化过程中逐渐从原始分子转移到新分子的生成部分。

4. Knowledge Embedding
作者进一步考虑除了SMILES表示之外的信息是否有助于提高模型在预测任务中的性能。作者提出了知识嵌入,它可以通过在输入中添加更多的嵌入组件来将特定的先验知识合并到模型中。作为探索性研究,作者设计了三种不同的知识嵌入策略(链接嵌入、环嵌入和类型嵌入)(图6a)。
- Link embedding :将SMILES中每个原子的连接信息、键和结构信息的符号结合起来。对于每个字符,对应的链接嵌入是它所连接的前一个原子的位置,而SMILES中第一个原子的链接嵌入由它自己的位置表示。通过这种方式,SMILES中的链接信息被合并。
- Ring embedding :在环嵌入中,表示开环或闭环原子的每个数字都用相应原子的位置表示。例如,指示开环的数字用闭环原子的位置表示,其他字符记录为NULL。这样,模型可以直接获取环数对所代表的连接信息和环结构信息。
- Type embedding:设计了类型嵌入,将字符分为九类,以整合其类型信息。
作者选择分子特性预测任务(HIV数据集)和DDI预测任务作为两个探索性研究。结果表明,使用所有的嵌入策略都不能提高模型在相应任务中的性能(图6b, c)。相反,这些额外的信息可能会使模型的学习更加困难。无论是否采用预训练策略,这三种嵌入策略都会在一定程度上降低模型性能。

四、Discussion
作者首次将大规模的预训练技术用于分子的理解和表示,并基于微调策略设计了用于各种下游分子分析任务的X-MOL。作者综合研究了X-MOL中的预训练策略、微调策略以及注意机制的可视化和解释。最后,X-MOL被证明在各种下游分子分析任务中取得了最先进的结果。所有这些任务都可以通过大规模的预训练和精心设计的微调统一在一个框架中,从而获得最先进的性能。
X-MOL有望进一步改进:
- 更好地定义学习SMILES语法规则的预训练策略
- 将这些预训练策略应用于其他分子表示,如基于Graph的表示
- 对其他下游分子分析任务进行微调
参考(更多细节见原文)
原文链接:X-MOL: large-scale pre-training for molecular understanding and diverse molecular analysis | bioRxivbioRxiv - the preprint server for biology, operated by Cold Spring Harbor Laboratory, a research and educational institution https://doi.org/10.1101/2020.12.23.424259
https://doi.org/10.1101/2020.12.23.424259
























 3676
3676

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










