前言
这次要写的是一本目前我看过的专业书里最厚的也是最新的一本-《人工智能与药物设计》,主要是总结一些书中所学,顺便把一些连接和网址整理在这里,更方便使用。可能写笔记的顺序会按照我的需要是不和书中的对应的,但标号还是会按照书里章节。今天就开始整理啦!加油加油~
一、机器学习基础
1.1 监督学习
1.1.1 概念
监督学习实际上就是对于有明确标签的数据进行学习,比较类似于有标准答案的考试(像是全是选择题那种考试,批改判分的时候有明确标准)。监督学习包括两种:分类问题和回归问题。
1.1.2 分类
分类问题类似于我们做过的判断题,答案是不连续的值。常见方法包括K最邻近算法和决策树。
1.1.3 回归
回归问题的答案不固定,是连续的值,常见方法有线性回归和逻辑回归。
1.2 无监督学习
1.2.1 概念
无监督学习是没有标签的学习,类似于没有标准答案的考试,通过一次次学习使得答案具有一定的偏向性。无监督学习包括:聚类、降维和自编码器。
1.2.2 无监督学习的基本算法
1.2.2.1 聚类
聚类所实现的任务是对样本进行分组,类似于学习中的分组问题。划分式聚类方法最典型的算法是K均值聚类算法,基于密度的聚类方法中DBSCAN方法最为突出。层次化聚类方法以Agglomerative和Divisive为代表算法。
1.2.2.2 降维
降维是一种缩小数据集维度的方法,它是将一个向量转化为一个维度更低的向量的方法。可以分为线性的方法(SVD和PCA)和非线性的方法(KPCA、LLE和T-SNE)
1.2.2.3 自编码器
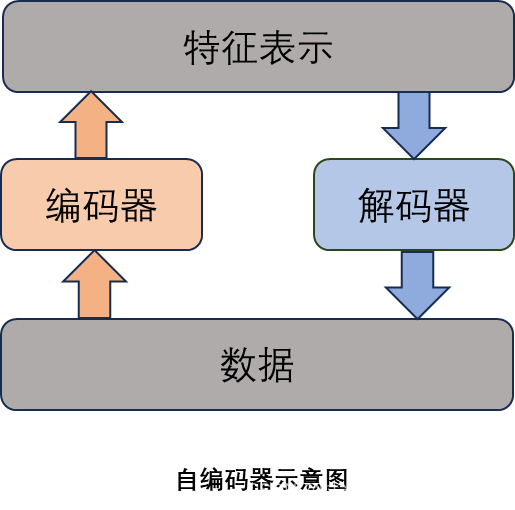
自编码器是无监督学习中的网络模型。它使得数据通过编码器和解码器,而进行非恒等变换的限制,使得隐藏层的向量作为目标输出。它有两层意义:1自编码器网络可以看作将数据进行压缩(从原来的n维输入降到m维) 2在需要的时候用损失最小的方式将数据恢复出来。
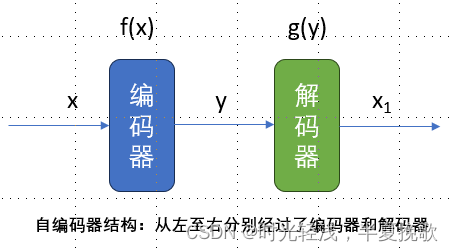
如图所示为自编码器的结构,要使得x和x1之间的误差尽可能小的前提下,y不等于x,自编码器输出的相当于是y。感觉这个图对概念解释的比较清晰,所以放在这里,供参考。
1.3 强化学习
1.3.1 概念
1.3.1.1 什么是强化学习
强化学习是一种无标签、无监督、试错的学习,类似于题海战术,不看答案,只通过批改去优化过程得到答案。
智能体对环境做出各种动作,获得反馈(奖励或惩罚)。
1.3.1.2 强化学习和马尔科夫决策过程
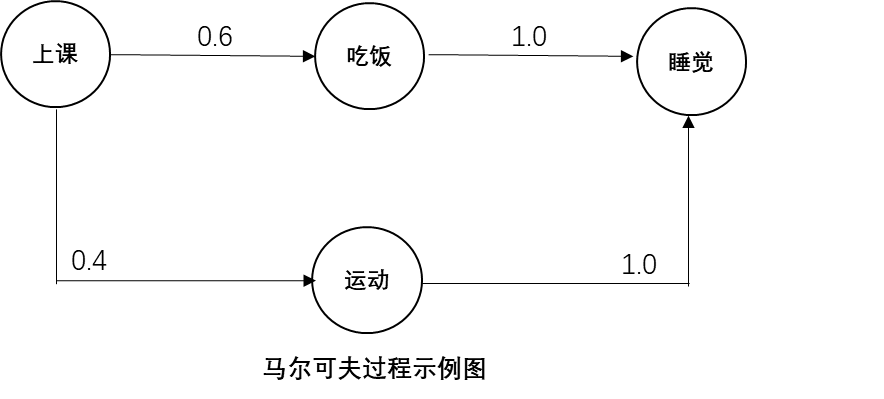
马尔可夫性:智能体的状态只有前后相邻的状态相关,即相互影响。
马尔可夫过程:由状态集和转移概率组成。如下图所示:
马尔可夫决策过程:四要素(状态集合S、动作集合A、状态转移函数P、奖励函数R)
下面的式子,策略Π表示在当前状态St时,执行动作At的概率:
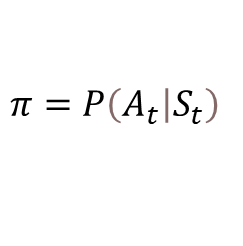
Gt表示累计汇报;γ表示衰减系数。
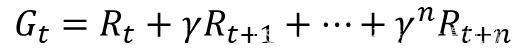
强化学习的过程示意图,如下所示:

1.3.2 有模型学习和免模型学习
1.3.2.1 有模型学习
根据真实环境构建虚拟环境,在虚拟环境和真实环境同时对模型进行训练学习。四要素完全已知。
1.3.2.2 免模型学习
直接在真实环境中进行交互,使模型学习到最优策略。四要素不完全已知(状态转移函数和奖励函数常常未知的情况)。
由于目前的研究中,大部分环境是静止的,只能提的状态是离散的,这种问题不需要估计奖励函数和状态转换函数,因此目前的强化学习模型大多是免模型学习的。
1.3.3 求解方法
策略迭代和值迭代。策略迭代需要优化策略一遍一遍迭代评估,值迭代改进了策略迭代,对价值期望进行评估,减少了迭代次数。
1.3.4 强化学习算法
蒙特卡洛学习是免学习模型,采集完整状态更新价值函数。
时序差分强化学习是免学习模型,将动态规划和蒙特卡洛采样相结合,从不完整状态估计完整状态,并更新价值。
Q-learning是经典的基于价值迭代的时序差分强化学习方法。
深度强化学习将深度学习与Q-learning结合,用深度神经网络来近似估计价值函数,并采取放回的方式对网络进行训练。
1.4 模型评估与验证
1.4.1 模型评估指标介绍
介绍了分类准确度、查准率、查全率、F1度量、ROC、AUC和代价敏感错误率。
1.4.2 模型验证方法介绍
介绍了交叉验证法、留出法、自助法和比较检验法(包括交叉验证t检验和Friedman检验)。
1.5 小结
这一章中的基本概念,我之前没用过的是强化学习的部分,因此笔记用了更多的篇幅帮助自己理解强化学习。整体来说这一张的概念还是很基础的,也还算容易理解,但好像没什么能上手的部分。
二、深度网络结构设计基础
2.1 卷积神经网络
2.1.1 卷积神经网络的组件
卷积层,是通过卷积核对图输入进行特征化的层,输入通过卷积层转化为特征。
池化层,是通过取平均或最大值对特征进行降维的层,特征通过池化层转化为更低维的特征。
激活函数,f(wx+b)其中x是我们的输入,进行线性操作是通过权重w和偏置b的线性相加,而f就是激活函数,它对输入进行的是非线性操作,常见的激活函数包括Sigmoid、ReLU和Tanh。(详细的可参考读书笔记-深度学习入门)
全连接层,为了使得通过各个层的输入的维度转化为标签所对应的维度,需要通过全连接层来实现这一操作。
2.1.2 神经网络的训练
损失函数,是计算输出和标签之间的误差后,而训练神经网络是为了使得这个误差最小化。
正则化,为了防止过拟合,在损失函数中加入一个正则项,这个正则项就可以防止训练的过程中的损失函数过拟合权重w。正则化的方法包括L1和L2,还有防止过拟合的方法包括dropout、批量归一化。(详细的可参考读书笔记-深度学习入门)
Softmax分类,softmax可以对输出进行归一化进而获得的是类别的概率,可以用交叉熵损失函数对网络的分类准确度进行评估。
2.1.3 基于卷积神经网络的图像分类
AlexNet
VGGNet
ResNet
它们是具有代表性的图像分类卷积神经网络,详细的模型细节通过网络的名字检索相应论文就好。
2.1.4 基于卷积神经网络的图像分割
这个概念是对输入的一张图像,语义分割将输出图像中的每个像素的分类标签。
FCN
U-Net
DeepLab
PSPNet
它们是具有代表性的图像分割卷积神经网络,详细的模型细节通过网络的名字检索相应论文就好。
2.2 循环神经网络
和一般神经网络不同的是,循环神经网络包括了时序关系。
2.2.1 循环神经网络结构
循环神经网络的结构是包括了一个循环输入的过程(输出又作为输入返回网络)。(详细的可参考读书笔记-深度学习进阶)
2.2.2 双向循环神经网络
影响往往不仅来自于前序输入,还与后序相关,而双向循环神经网络则在结构中包括了后序数据的传输。
2.2.3 深度循环神经网络
除了横向的循环以外,在深度上进行网络的加深,就得到了深度循环神经网络。
2.2.4 长短期记忆网络
LSTM,长短期记忆网络包括三个门:输入门、遗忘门和输出门。(详细的可参考读书笔记-深度学习进阶)
2.2.5 双向长短期记忆网络
双向长短期记忆网络包括前向的和后向的长短期记忆网络,将过去和未来时序信息相结合,使得信息输入更加完整。
2.2.6 门控循环单元
GRU,简化了LSTM,其结构仅包括两个门:重置门和更新门。(详细的可参考读书笔记-深度学习进阶)
2.2.7 基于长短期记忆网络的视频分类
举例了循环神经网络在视频分类任务中的应用。
2.3 Transformer
在自然语言处理问题中能很好地处理RNN、LSTM等可能会出现的梯度爆炸的问题。
2.3.1 自然语言处理中的Transformer
包括自注意力模块、前向传播层和编码器-解码器间的注意力层。
2.3.2 视觉任务中的Transformer
介绍了了两个有代表性的Vision Transformer和Swin Transformer。
2.4 图神经网络
可处理非欧几里得空间的数据,将数据以节点、边和邻接矩阵进行表示,然后通过不同网络比如书里举例的是图卷积神经网络(GCN)和图注意力网络(GAT)。
2.5 小结
这一章对深度学习的模型进行了科普性的介绍,包括图处理的卷积神经网络,自然语言处理的循环神经网络和Transformer,以及非欧几里得数据处理的图神经网络。
三、深度生成模型
3.1 变分自编码器
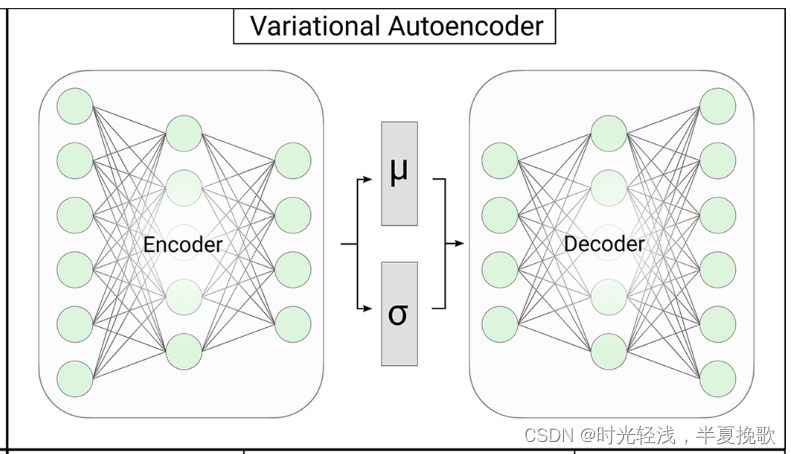
变分自编码器(VAE)可以从未标注数据中自动学到有用特征。
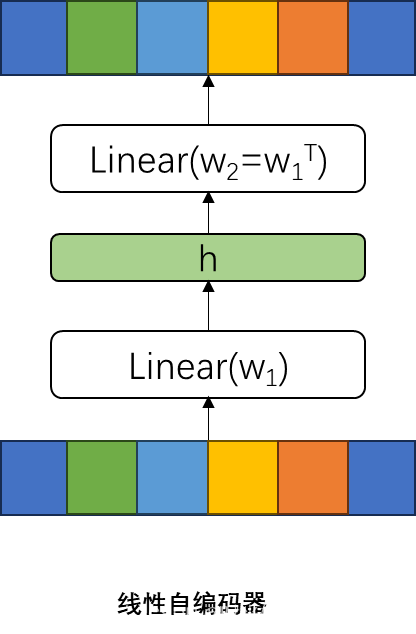
3.1.1 自编码器
编码器的部分,是通过特征抽取函数,将输入数据转化为特征。
解码器的部分,是通过特征还原数据函数,将特征还原为数据。
整个自编码器优化的目标函数是输入数据经过编码解码的输出和原输入之间的误差,需要优化这个误差到最小值。
3.1.2 隐变量生成模型
编码器中的变量x是数据带来的出现过的变量,而隐变量z是解码器中才出现的新变量。z需要还原出x的概率分布。
实际问题中需要选择合适的概率分布来刻画隐变量和条件生成过程。
3.1.3 变分自编码器
给定一组数据和一个隐变量生成模型,假设数据是由隐变量生成得到。学习的目的是估计最合适的参数,使得隐变量生成模型的似然函数最大。
在找这个最大值的参数的时候,无法直接求解该函数的最大值,所以找到该函数的下界函数,优化参数可得到下界函数的最大值,而这种优化方法得到的结果适用于原函数,这种近似优化求解的方法被称为变分法。
3.2 生成式对抗网络
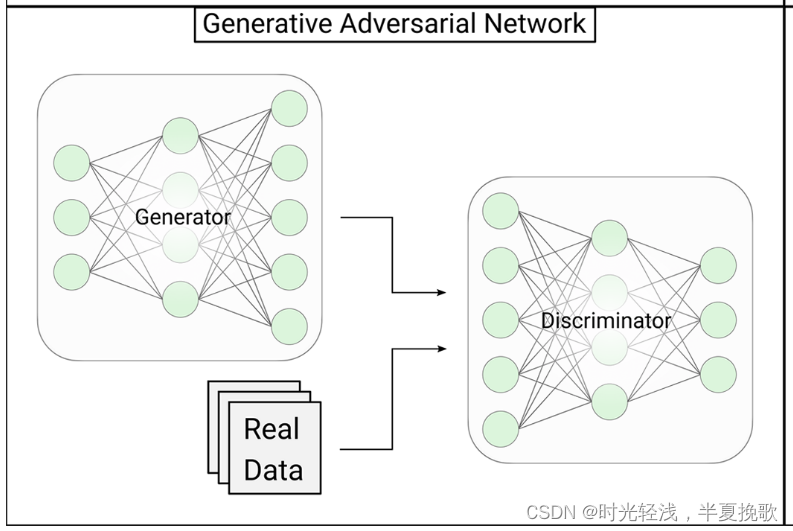
如上图所示,
生成器标记为G,输入的是高斯噪声,输出生成的样本数据。
判别器标记为D,输入的是真实数据和生成的数据,输出的是0或1(判别为真实数据/合成数据)。
生成对抗网络的目标函数是,1优化生成器,最小化生成器G生成的样本数据输入判别器D的预测值(尽可能接近0),2优化判别器,真实数据输入判别器D,D的预测值接近0,而合成数据输入判别器D,D的预测值接近1。将这两个目标合并起来就构成了生成对抗网络要优化的目标函数。
3.2.1 生成式对抗网络的理论分析
优化推到的目标函数,可得到生成对抗网络求解的本质是最小化生成器生成数据的概率分布和真实数据之间的JSD(两个分布之间距离的一种度量)。之前的VAE优化的极大似然估计(MLE),它的本质是最小化生成网络于数据之间的KL散度,而生成对抗网络本质是将极大似然估计中的KL散度换成了JSD,而JSD是对称的,用来衡量分布之间的距离更为合适。
3.2.2 Wasserstein生成式对抗网络
将分布间的度量换掉了,从JSD换成了Wasserstein距离(EMD),从而改进网络效果。这个距离又被称为动土距离,可以用三个土堆,如何搬动搬运代价最小的问题进行理解。
3.3 流生成模型
3.3.1 随机变量替换
3.3.2 标准化流
3.3.3 RealNVP网络
3.3.4 Glow
3.3.5 流模型在文本预训练表示上的应用
六、分子结构与生物活性数据
6.1 生物大分子结构数据库
实验解出来的蛋白质结构数据库RSCB PDB: https://www.rcsb.org/
AlphaFold2预测出来的蛋白质结构数据库AlphaFold DB: https://www.alphafold.ebi.ac.uk/
核酸结构数据库NDB: http://ndb-mirror-3.rutgers.edu/
蛋白质-小分子复合物结构数据库:
1)PDBbind:http://www.pdbbind.org.cn/ (按照亲和力的实验方法尽心了数据集的划分)
2)Binding MOAD: https://bindingmoad.org/ (分辨率优于2.5,但并不都提供亲和力数据)
3)BioLiP: https://zhanggroup.org/BioLiP/index.cgi (总结了其它数据库)
4)dbHDPLS: http://DeepLearner.ahu.edu.cn/web/dbDPLS/ (链接打不开)(和人类疾病相关的)
蛋白质-蛋白质复合物结构数据库:
STRING: https://cn.string-db.org/ (比较多)
PPInS: http://www.cup.edu.in:99/ppins/home.php (链接打不开)(比较早)
蛋白-核酸复合物结构数据库:
1)NPIDB:https://npidb.belozersky.msu.ru/ (以前可以打开,现在链接好像失效了)(蛋白质-DNA和蛋白质-RNA复合物结构和相互作用信息)
2)PDIdb:http://www.melolab.org/pdidb/web/content/home (蛋白质-DNA复合物结构)
3)DNAproDB:https://dnaprodb.usc.edu/ (蛋白质-DNA复合物结构)
4)PRIDB:http://pridb.gdcb.iastate.edu/RPISeq/download.php (蛋白质-RNA复合物结构)
蛋白质-多肽复合物结构数据库:
1)PepBDB:http://huanglab.phys.hust.edu.cn/pepbdb/ (多)
2)PepX:http://pepx.switchlab.org(链接打不开)(非冗余)
G蛋白偶联受体数据库:
1)GPCRdb:https://gpcrdb.org/ (结合位点和突变位点信息)
2)GPCR-EXP:https://zhanggroup.org/GPCR-EXP/ (实验值和预测值)
3)GLASS:https://zhanggroup.org/GLASS/ (有结合亲和力)
4)Human-gpDB:https://human-gpdb.lcsb.uni.lu/ (人类的)
蛋白激酶数据库:
1)KLIFS:https://klifs.net/ (人类的)
2)HomoKinase:http://www.biomining-bu.in/homokinase/(链接打不开)(同源的)
3)KinaseMD:https://bioinfo.uth.edu/kmd/ (突变和药物反应信息)
4)DKK:https://darkkinome.org/ (研究不足的激酶)
5)KinLigDB:http://www.lilab-ecust.cn/profkin/index.html (复合物结合模式)
金属酶数据库MetalPDB:http://metalpdb.cerm.unifi.it/
固有无需蛋白质结构数据库:
1)DisProt:http://original.disprot.org/
2)MoobiDB:https://mobidb.org/
其它特定功能的结构数据库:
1)ProCarbDB:http://www.procarbdb.science/procarb/ (链接打不开)(碳水化合物)
2)ADPriboDB:https://adpribodb.adpribodb.org/ (ADP核糖化)
3)DNAmoreDB:https://www.genesilico.pl/DNAmoreDB/ (DNA酶)
4)RBP2GO:https://rbp2go.dkfz.de/ (RBP)
肽类结构数据库:
1)FoldamerDB:http://foldamerdb.ttk.hu/ (具有折叠结构)
2)ConjuPepDB:https://conjupepdb.ttk.hu/ (偶联药物)
3)StraPep:http://www.isyslab.info/StraPep/ (具有生物活性)
4)DBAASP:https://dbaasp.org/home (抗菌肽及活性)
十二、小分子药物设计与优化
12.1 小分子-靶标结合亲和力预测与打分函数的设计
12.1.1 小分子把表亲和力预测与打分函数
这个部分回顾了相互作用的计算方法(FEP、TI、MM/P(G)BSA),以及打分函数(基于物理的、经验的、知识的),引出了接下来要介绍的基于人工智能的打分函数的详细介绍。
12.1.2 基于人工智能的打分函数
这个部分我整理了这些论文的原文链接和开源的代码链接:
1)RF-Score:https://doi.org/10.1093/bioinformatics/btq112
2)ID-Score:https://pubs.acs.org/doi/10.1021/ci300493w
3)ΔvinaRF20:https://onlinelibrary.wiley.com/doi/10.1002/jcc.24667
https://www.nyu.edu/projects/yzhang/DeltaVina
4)Pafnucy:https://doi.org/10.1093/bioinformatics/bty374
http://gitlab.com/cheminfIBB/pafnucy
5)InteractionGraphNet:https://pubs.acs.org/doi/10.1021/acs.jmedchem.1c01830
https://github.com/zjujdj/InteractionGraphNet/tree/master
12.1.3 基于人工智能的DTA预测模型
1)SimBoost:https://jcheminf.biomedcentral.com/articles/10.1186/s13321-017-0209-z
2)DeepDTA:https://academic.oup.com/bioinformatics/article/34/17/i821/5093245?login=true
https://github.com/hkmztrk/DeepDTA
12.1.4 问题和展望
目前领域的局限性可被概括为:1泛化能力不足,2数据集不能描述真实问题,3可解释性。接下来针对这三个局限性的小分子-靶标结合亲和力预测的打分函数仍然被期待。
12.2融合人工智能的分子对接与虚拟筛选方法
12.2.1 分子对接方法与挑战
12.2.2 机器学习与系综对接
12.2.3深度学习与结合构相预测
12.2.4 深度学习与虚拟筛选
总结
慢慢来吧,未完待续,继续努力。这篇笔记的更新顺序源自我读了哪个部分,所以有时按顺序有时不按,主打一个随意。





















 3355
3355











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








