R语言
R语言实操记录——获取包的三种渠道及安装包的三种方式
一、获取包的三种渠道
- 渠道1 、CRAN:R语言的官网,里面包含有绝大多部分领域的大部分包。
- 渠道2、Bioconductor:生物专业的相关网页,包含有大部分的专业数据分析包。
- 渠道3、Github:一部分还未被官网收录的包或者比较新的包会被发布在Github上。
二、安装包的三种方式
- 每种渠道都有自己的安装方式
2.1、CRAN
-
之前将了基于R的两种安装包的方式link,这里讲一下基于RStudio的三种安装方式。
-
三种方法就是基于之前讲的两种方法的演变,前两种是在线安装,第三种是源码安装
-
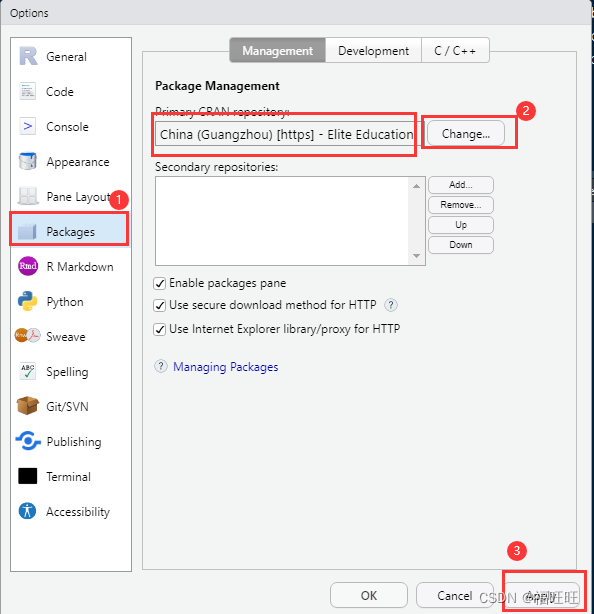
安装前可以在RStudio中的选项卡 Tools -> Global Options -> Packages 下调整镜像源(CRAN),选China的可以提高下载速度,当前镜像源崩溃时也可以用这方法来切换别的镜像源下载。(此方法是基于RStudio的,基于R的也在前面文章中介绍过)
-
-
方法一:调用函数 install.packages(“packagename”)(单引号也是一样的效果)
-
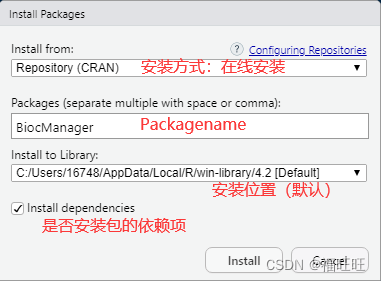
方法二:RStudio中的选项卡 Tools -> Install Packages(在线安装)
-
-
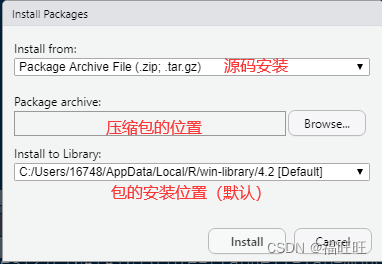
方法三:找到对应官网,下载源码压缩包(.zip,.tar.gz)后在RStudio中的选项卡 Tools -> Install Packages(源码安装)(需要自己下载依赖项)
-
-
2.2、Bioconductor
- 在线安装:
if (!require("BiocManager", quietly = TRUE))
install.packages("BiocManager")
# 安装Bioconductor的包需要用到BiocManager包来实现(::表示调用BiocManger中的install函数)
# 上面两句函数是用来搜索是否已经安装BiocManager包的,没有则安装。
# 假如已经确定安装过此包,可以直接输入下面的语句来安装包
BiocManager::install("Packagename")
- 源码安装:(同上述方法三)
- 切换镜像源:Bioconductor是外网,在下载时容易中断或速度较慢,可以切换为国内的镜像源。
- 清华镜像
- 运行下述语句就可以实现安装
options(BioC_mirror="https://mirrors.tuna.tsinghua.edu.cn/bioconductor")
2.3、Github
- 安装方式:(官网上查找对应的包,下面一般会给出安装语句)
install.packages("devtools")
# 预先安装devtools包
devtools::install_github('Creatorname/Packagename')
- 示例:安装monocle3包
devtools::install_github("cole-trapnell-lab/monocle3")
注意
- 一般在安装Bioconductor包或者Github包时会提问是否需要更新其他包,这里选择否
- 原因:包之间都存在着依赖关系,但是由于开发包的时间不同,基于的版本号也不同,更新可能会导致包之间依赖关系的崩溃,所以建议不要全部更新,而是只更新需要的包。






















 2万+
2万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








