实验:糖尿病预测
1,实验目的
1、通过数据探索技术完成对数据的认识
2、通过数据的描述性统计分析完成热销商品的分析并用可视化图标进行展示
3、通过数据预处理进行数据转换以满足构建预测模型的要求
4、采用决策树算法进行模型构建并对结果进行说明(自己编程实现或调用python第三方包均可)
5、采用朴素bayes算法进行模型构建并对结果进行说明(自己编程实现或调用python第三方包均可)
6、采用knn算法进行模型构建并对结果进行说明(自己编程实现或调用python第三方包均可)
7、采用随机森林等提升树算法进行模型构建并对结果进行说明(自己编程实现或调用python第三方包均可)
8、进行模型评估并选出最优的算法。
2,实验步骤
背景:根据美国疾病控制预防中心的数据,现在美国1/7的成年人患有糖尿病。但是到2050年,这个比例将会快速增长至高达1/3。现在我们可以通过UCL机器学习数据库里一个糖尿病公共数据集来了解利用机器学习如何完成糖尿病预测
变量:
Pregnancies:怀孕次数
Glucose:葡萄糖测试值
BloodPressure:血压
SkinThickness:皮脂厚度
Insulin:胰岛素
BMI:身体质量指数
DiabetesPedigreeFunction:糖尿病遗传函数
Age:年龄
Outcome:糖尿病标签
1、数据探索
1.1导入包及读取数据
import numpy as np import pandas as pd import matplotlib.pyplot as plt import seaborn as sns import missingno as msno from sklearn import metrics from sklearn.linear_model import LogisticRegression from sklearn.metrics import classification_report from sklearn.impute import SimpleImputer as Imputer from sklearn.metrics import confusion_matrix,accuracy_score,f1_score,roc_auc_score,recall_score,precision_score,roc_curve from sklearn.tree import DecisionTreeClassifier from sklearn.model_selection import KFold from sklearn.discriminant_analysis import LinearDiscriminantAnalysis from sklearn.naive_bayes import GaussianNB from sklearn.model_selection import cross_val_score from sklearn.neighbors import KNeighborsClassifier from sklearn.model_selection import train_test_split from sklearn.ensemble import RandomForestClassifier, GradientBoostingClassifier from sklearn.model_selection import cross_validate from sklearn.metrics import roc_curve, auc data = pd.read_csv('E:../diabetes.csv', encoding='gbk') # 输入的数据文件
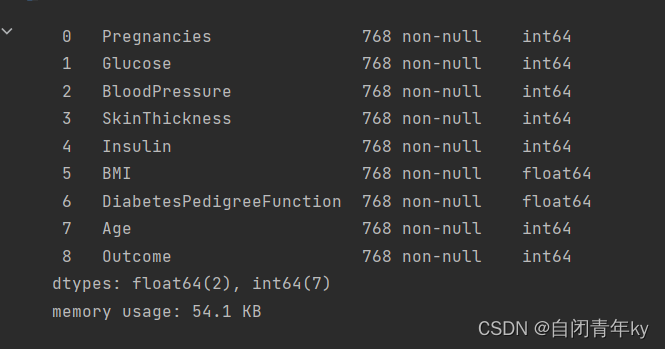
1.2对数据的基本认识
data.info()
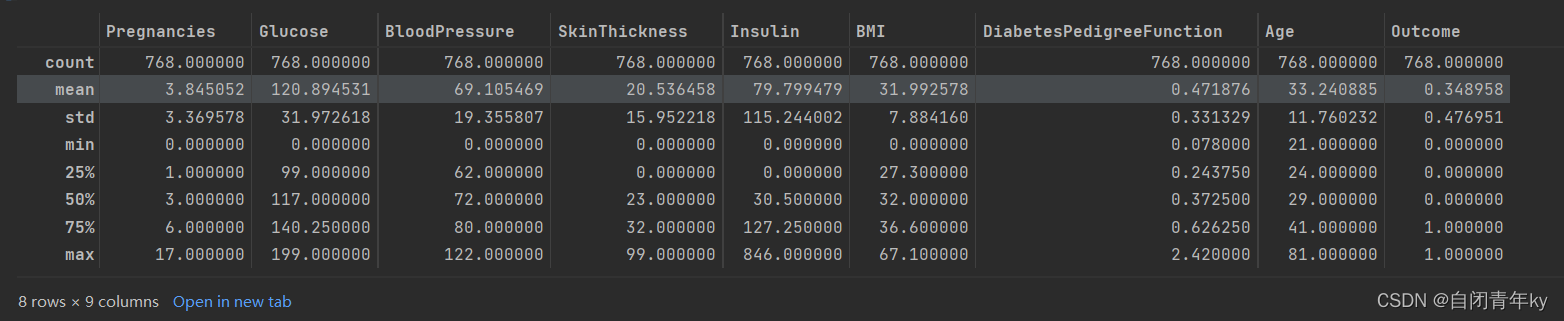
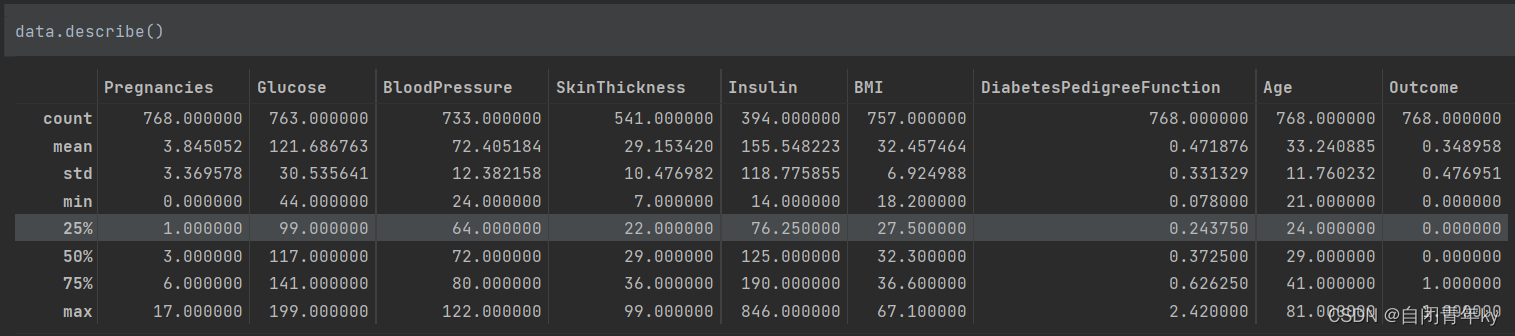
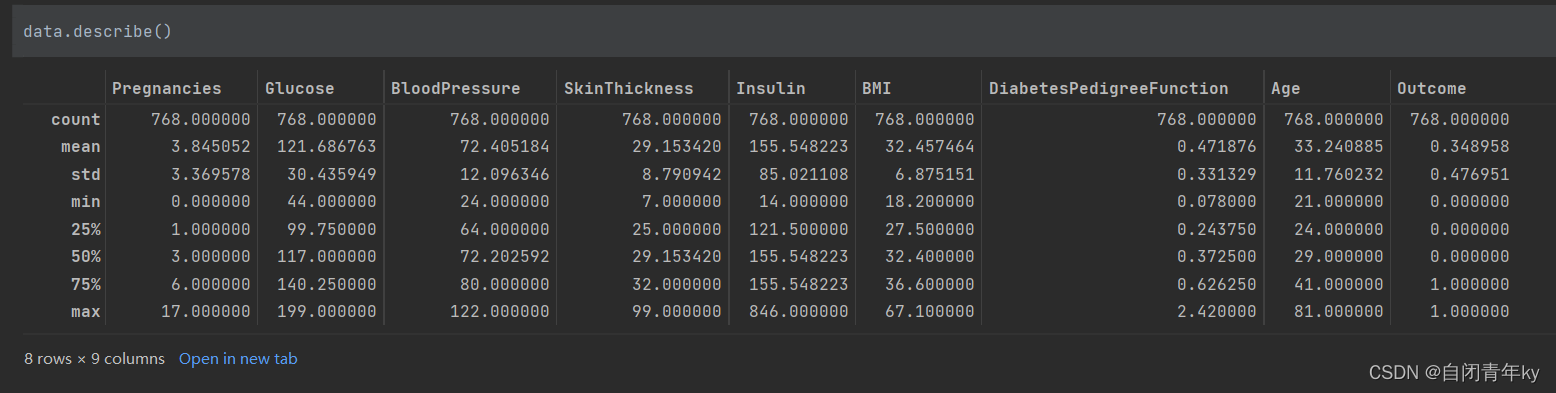
#查看数据矩阵 data.describe()
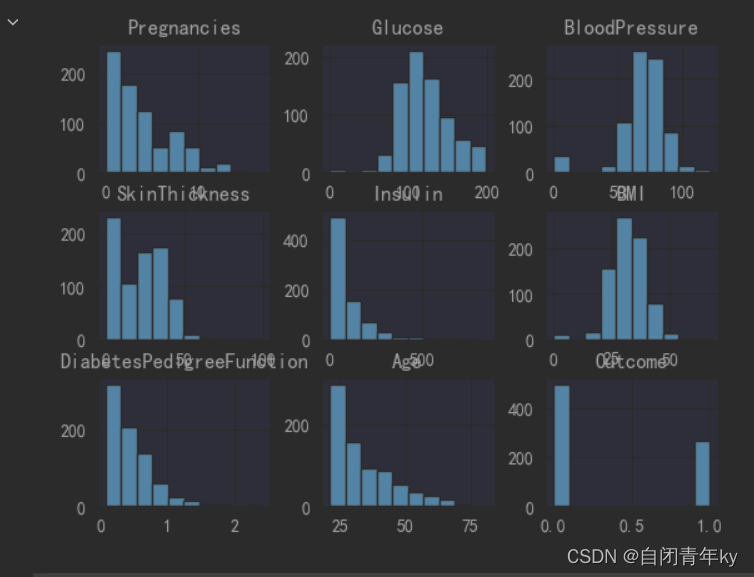
1.3数据描述性统计分析
# 使用柱状图的方式画出标签个数统计 data.hist() plt.show()
# 使用 sns.pairplot 进行可视化分析 plt.scatter(data.Insulin,data.Glucose) plt.xlabel("Insulin") plt.ylabel("Glucose") plt.show()
2,数据预处理
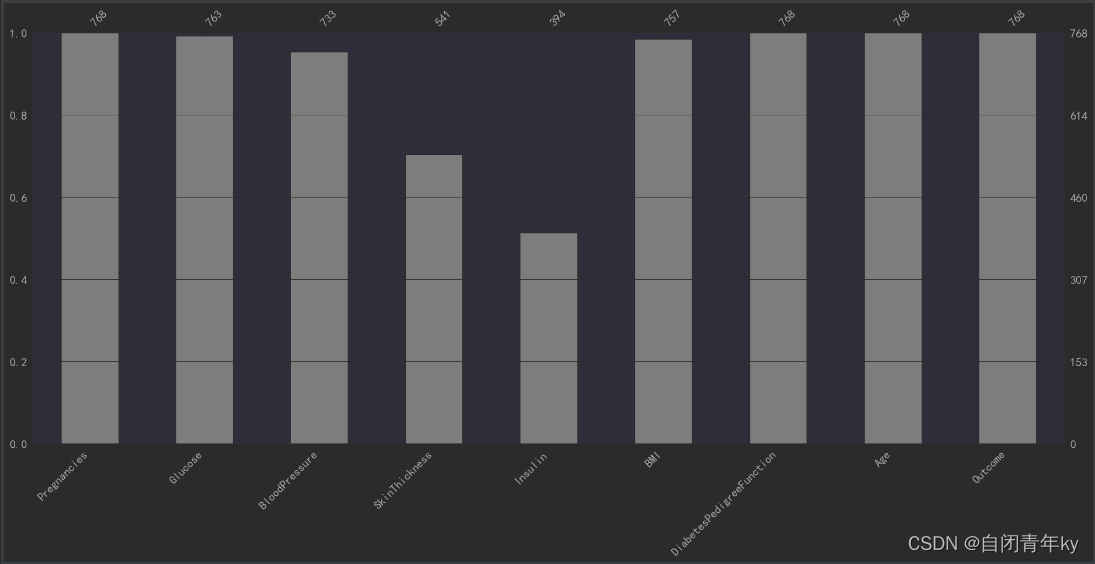
#使用SimpleImputer缺失值分析、可视化及处理 # 异常值处理(用空值替换) colume = ['Glucose', 'BloodPressure', 'SkinThickness', 'Insulin', 'BMI'] data[colume] = data[colume].replace(0,np.nan) # 可视化 import missingno as msno p=msno.bar(data) plt.show()
# 导入插补库 from sklearn.impute import SimpleImputer # 对数值变量的缺失值处理,采用均值插补的方法来填充缺失值 imr = SimpleImputer(missing_values=np.nan, strategy='mean') # 插补 data[colume] = imr.fit_transform(data[colume])
3、模型构建
(1)数据特征与标签分离
把数据切分为特征x和标签y
x = data.drop("Outcome", axis=1) y = data.Outcome(2)特征工程
使用sklearn包中 ExtraTreesClassifier算法进行特征选取
from sklearn.ensemble import ExtraTreesClassifier model = ExtraTreesClassifier() model.fit(x,y)数据标准化
from sklearn.preprocessing import StandardScaler scaler = StandardScaler() scaler.fit(data) data=scaler.transform(data)(4)决策树模型构建
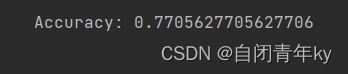
#决策树 from sklearn.model_selection import train_test_split #导入 train_test_split 函数 from sklearn import metrics #导入scikit-learn模块以计算准确率 # 将数据集分成训练集和测试集 X_train,X_test,y_train,y_test = train_test_split(x,y,test_size=0.3, stratify=y) # 创建决策树分类器对象 clf = DecisionTreeClassifier(criterion='entropy')# 训练模型 clf = clf.fit(X_train,y_train)# 使用训练好的模型做预测 y_pred = clf.predict(X_test) # 模型的准确性 print("Accuracy:",metrics.accuracy_score(y_test, y_pred))
(5)朴素bayes模型构建
X_train,X_test,y_train,y_test = train_test_split(x,y,test_size=0.3, stratify=y, random_state=20) from sklearn.metrics import accuracy_score, classification_report from sklearn.naive_bayes import GaussianNB from sklearn.metrics import confusion_matrix NB = GaussianNB() NB.fit(X_train,y_train) predict_results =NB.predict(X_test) # 模型评估目标 print("Accuracy:",accuracy_score(predict_results, y_test))

(6)knn算法模型构建
# knn from sklearn.neighbors import KNeighborsClassifier X_train,X_test,y_train,y_test = train_test_split(x,y,test_size=0.3, stratify=y) # 保存不同k值测试集准确率 training_accuracy = [] # 保存不同k值训练集准确率 test_accuracy = [] # try n_neighbors from 1 to 10 neighbors_settings = range(1, 11) for n_neighbors in neighbors_settings: # build the model knn = KNeighborsClassifier(n_neighbors=n_neighbors) knn.fit(X_train, y_train) # record training set accuracy 保存训练集准确率 training_accuracy.append(knn.score(X_train, y_train)) # record test set accuracy 保存测试集准确率 test_accuracy.append(knn.score(X_test, y_test)) plt.figure() plt.plot(neighbors_settings, training_accuracy, label="training accuracy") plt.plot(neighbors_settings, test_accuracy, label="test accuracy") plt.ylabel("Accuracy") plt.xlabel("n_neighbors") plt.legend() # 给图像加上图例 plt.show()
由图所知n大致取8的时候拟合结果最接近
knn = KNeighborsClassifier(n_neighbors=8) knn.fit(X_train, y_train) from sklearn.metrics import confusion_matrix y_pred = knn.predict(X_test) print('Accuracy:{}'.format(metrics.accuracy_score(y_pred, y_test)))
(7)提升树模型构建
from sklearn.ensemble import GradientBoostingClassifier x_train,x_test,y_train,y_test=train_test_split(x,y,test_size=0.3, stratify=y, random_state=20) gbrt=GradientBoostingClassifier(random_state=1) gbrt.fit(x_train,y_train) print("Accuracy:",metrics.accuracy_score(y_test,gbrt.predict(x_test)))
(8)roc图
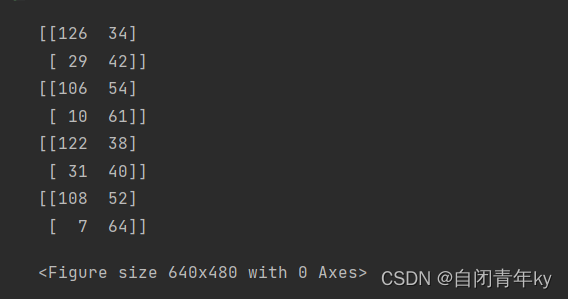
X_train, X_test, y_train, y_test = train_test_split(x, y, test_size=0.3, random_state=8675309) # 绘制roc曲线 def calculate_auc(y_test, pred): print("auc:", roc_auc_score(y_test, pred)) fpr, tpr, thersholds = roc_curve(y_test, pred) roc_auc = auc(fpr, tpr) plt.plot(fpr, tpr, 'k-', label='ROC (area = {0:.2f})'.format(roc_auc), color='blue', lw=2) plt.xlim([-0.05, 1.05]) plt.ylim([-0.05, 1.05]) plt.xlabel('False Positive Rate') plt.ylabel('True Positive Rate') plt.title('ROC Curve') plt.legend(loc="lower right") plt.plot([0, 1], [0, 1], 'k--') plt.show() # 使用Yooden法寻找最佳阈值 def Find_Optimal_Cutoff(TPR, FPR, threshold): y = TPR - FPR Youden_index = np.argmax(y) # Only the first occurrence is returned. optimal_threshold = threshold[Youden_index] point = [FPR[Youden_index], TPR[Youden_index]] return optimal_threshold, point # 计算roc值 def ROC(label, y_prob): fpr, tpr, thresholds = roc_curve(label, y_prob) roc_auc = auc(fpr, tpr) optimal_threshold, optimal_point = Find_Optimal_Cutoff(TPR=tpr, FPR=fpr, threshold=thresholds) return fpr, tpr, roc_auc, optimal_threshold, optimal_point # 计算混淆矩阵 def calculate_metric(label, y_prob, optimal_threshold): p = [] for i in y_prob: if i >= optimal_threshold: p.append(1) else: p.append(0) confusion = confusion_matrix(label, p) print(confusion) TP = confusion[1, 1] TN = confusion[0, 0] FP = confusion[0, 1] FN = confusion[1, 0] Accuracy = (TP + TN) / float(TP + TN + FP + FN) Sensitivity = TP / float(TP + FN) Specificity = TN / float(TN + FP) return Accuracy, Sensitivity, Specificity # 多模型比较: models = [('KNN', KNeighborsClassifier()), ('GN', GaussianNB()), ('DT', DecisionTreeClassifier(random_state=0)), ('GBDT', GradientBoostingClassifier(random_state=1))] # 循环训练模型 results = [] roc_ = [] for name, model in models: clf = model.fit(X_train, y_train) pred_proba = clf.predict_proba(X_test) y_prob = pred_proba[:, 1] fpr, tpr, roc_auc, Optimal_threshold, optimal_point = ROC(y_test, y_prob) Accuracy, Sensitivity, Specificity = calculate_metric(y_test, y_prob, Optimal_threshold) result = [Optimal_threshold, Accuracy, Sensitivity, Specificity, roc_auc, name] results.append(result) roc_.append([fpr, tpr, roc_auc, name]) df_result = pd.DataFrame(results) df_result.columns = ["Optimal_threshold", "Accuracy", "Sensitivity", "Specificity", "AUC_ROC", "Model_name"] # 绘制多组对比roc曲线 color = ["darkorange", "navy", "red", "green", "yellow", "pink"] plt.figure() plt.figure(figsize=(10, 10)) lw = 2 plt.plot(roc_[0][0], roc_[0][1], color=color[0], lw=lw, label=roc_[0][3] + ' (AUC = %0.3f)' % roc_[0][2]) plt.plot(roc_[1][0], roc_[1][1], color=color[1], lw=lw, label=roc_[1][3] + ' (AUC = %0.3f)' % roc_[1][2]) plt.plot(roc_[2][0], roc_[2][1], color=color[2], lw=lw, label=roc_[2][3] + ' (AUC = %0.3f)' % roc_[2][2]) plt.plot(roc_[3][0], roc_[3][1], color=color[3], lw=lw, label=roc_[3][3] + ' (AUC = %0.3f)' % roc_[3][2]) plt.plot([0, 1], [0, 1], color='black', lw=lw, linestyle='--') plt.xlim([0.0, 1.0]) plt.ylim([0.0, 1.05]) plt.xlabel('False Positive Rate') plt.ylabel('True Positive Rate') plt.title('Receiver operating characteristic Curve') plt.legend(loc="lower right") plt.show()
(9)模型评估并选取最优算法模型
略


























 1799
1799











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








