一、第一步安装环境
pip install dicom2nifti二、第二步写代码
现在我有一个文件夹里面都是dicom序列需要转换

import dicom2nifti
if __name__ == '__main__':
dicom_path = "HKMTEVPH/3EIKEE1O"
dicom2nifti.convert_directory(dicom_path,'./')
结果就会保存在当前目录下:
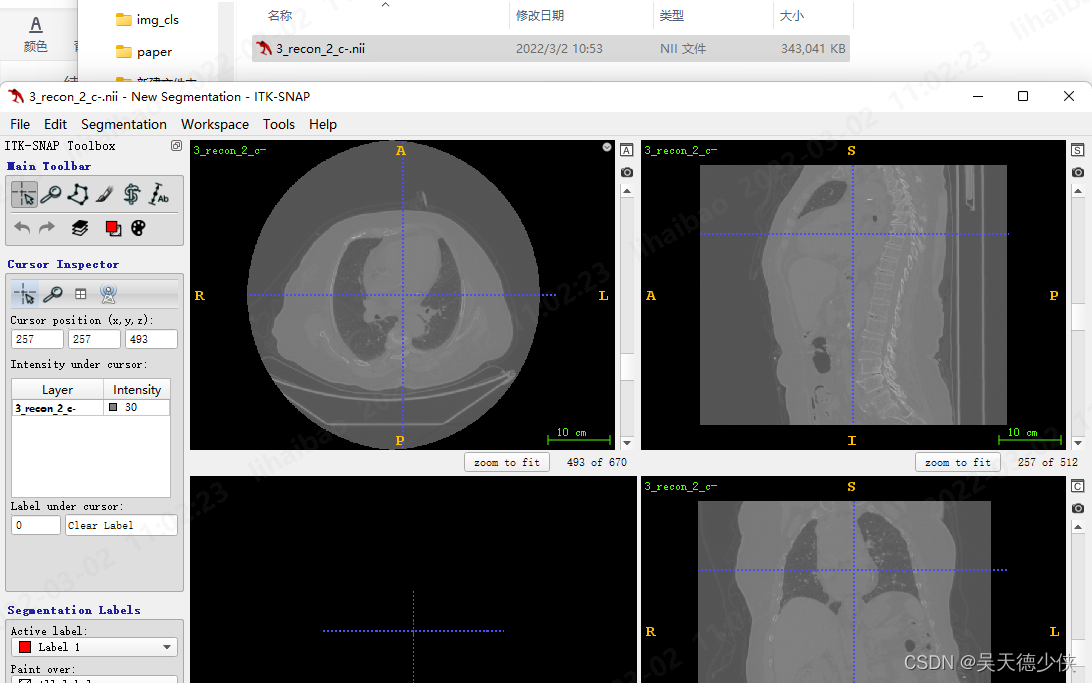
解压之后,使用itk-snap软件打开:
pip install dicom2nifti现在我有一个文件夹里面都是dicom序列需要转换

import dicom2nifti
if __name__ == '__main__':
dicom_path = "HKMTEVPH/3EIKEE1O"
dicom2nifti.convert_directory(dicom_path,'./')
结果就会保存在当前目录下:

解压之后,使用itk-snap软件打开:

 5672
5672











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?


