大多数医学成像设备以复杂的 DICOM 格式(后缀 .dcm)的变体存储图像。许多科学工具希望医学图像以更简单的 NIfTI 格式(后缀 nii.gz)存储。事实上,我们做深度学习基本都是使用的 nii.gz 格式或者 nii 格式。
那么,如何将 dicom 格式转换为 Nifti 格式,是我们做研究的第一个难题。
本次推荐一个特别好用的转换工具(dcm2niix),可以批量转换你的 dicom 数据。当然,你要是不满足一种方案,直接拉到文章最后,查看转换工具汇总,总有一个适合你
文章目录
dcm2niix 简介
dcm2niix 是一款由 NTRC团队开发的医学图像转换工具,用于将数字成像与通信(DICOM)格式的医学图像转换为更加通用的神经影像学信息交换(NIfTI)格式。该软件直接读取磁共振成像(MRI)和计算机断层扫描(CT)数据,并把它们转换为 NIfTI 格式。除此之外,dcm2niix 还能够处理其他信息,例如医院信息化系统(HIS)中的元数据、图像方向和标签等。
NTRC 团队还开发了 MRICron 软件,这是一款 NIfTI 格式的看图软件,而且它可以方便地调用 dcm2niix 进行图像转换。dcm2niix 的出现使得神经影像学数据的转换和处理更加高效、准确和便捷。
dcm2niix 使用方法-简单版
为了让各位看官对 dcm2niix 。有个初步印象和了解。在这里,先简单介绍一下如何使用。后面再跟深入的讲解安装和使用细节~
简单讲,使用分为 图形界面版和命令版
图形界面版

图形界面版需要先下载 MRICron。在上图左边设置好输出文件的命名格式,输出文件夹等参数,把dcm文件夹拖到右边空白处即可完成转换。
命令行版
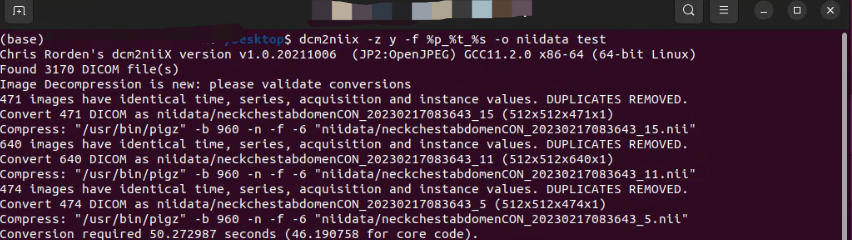
对于使用 shell 命令不太熟,数据量少的朋友,可以使用图形界面版,鼠标点点点就可以了。当然计算机玩家还是推荐命令行版。
dcm2niix 图形界面安装和使用
图形界面版的安装
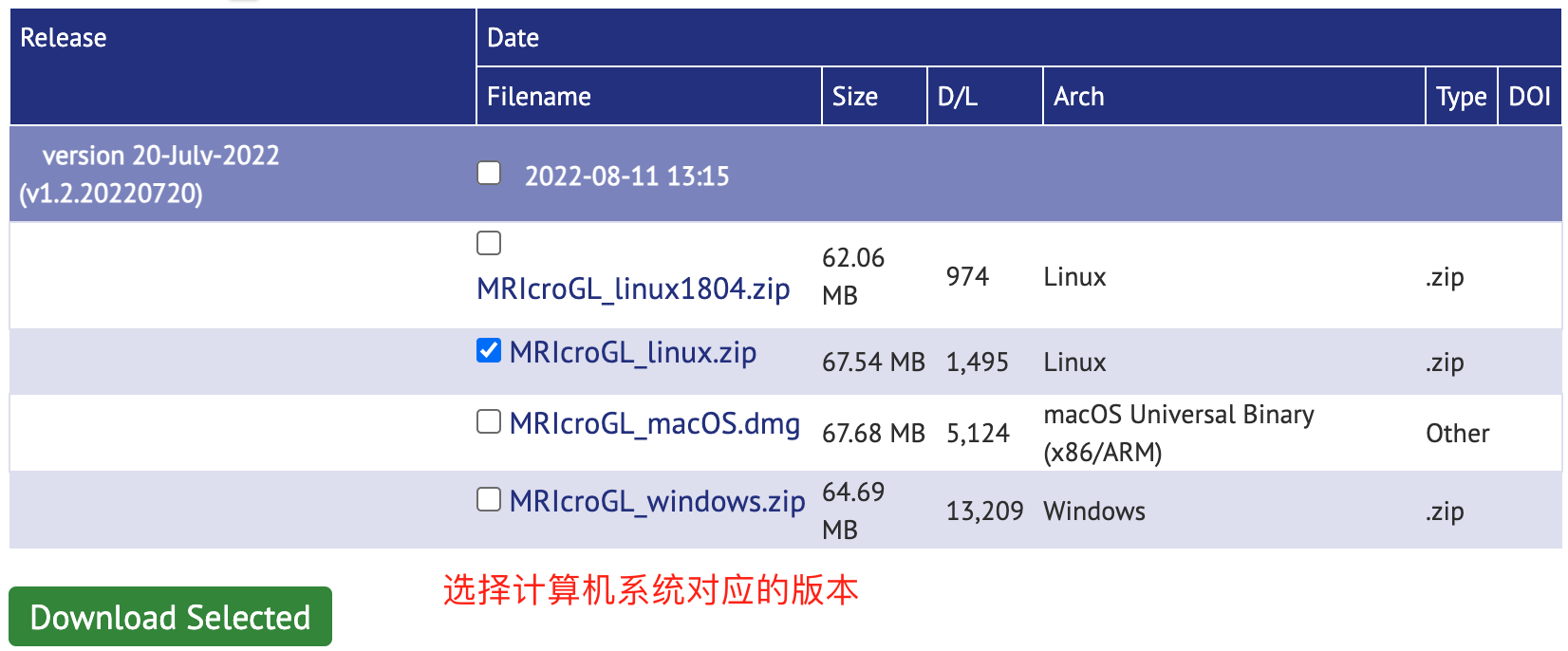
官方推荐下载 MRIcroGL 版本,是 MRICron 的升级版本。如下图,选择对应的操作系统下载安装即可
软件打开界面如下图👇

这个界面我还没研究透。还是用 MRICron 比较顺手。
使用方法
MRIcroGL包括一个简单的图形用户界面,用于控制dcm2niix。启动 MRIcroGL 后,选择 Import/ConvertDICOMtoNIfTI菜单项。
界面如下图所示。

复选框设置输出是压缩(创建.nii.gz文件)还是不压缩(创建.nii文件)。然后,您可以使用图片中描述的格式指定所需的输出文件名。
当您调整所需的输出名称时,软件将显示一个示例,说明您的文件将如何显示。您还可以选择是否要将输出保存到特定的文件夹中,或者是否应该在与输入DICOM文件相同的文件夹中创建NIfTI图像。
最后一步是拖放要转换为应用程序的文件夹。注意,界面总是以交互方式显示命令行,因此,当您通过选择复选框与界面交互时,您正在创建一个文本命令,您可以将其复制并粘贴到您喜欢的脚本中。
注意:我在 mac 中使用 MRIcroGL 转 Nii 时,会提示没有有效的DICOM文件,但是我换成 MRICron 就能成功。奇了怪了,搞不懂,遇到同样问题的话,你就使用 MRICron。
MRICron的下载链接,软件示意图👇

图形界面版比较简单,就介绍这么多
dcm2niix 命令行版
命令行版的安装
有几种方法可以安装 dcm2niix
- 有 conda,
conda install -c conda-forge dcm2niixon Linux, MacOS or Windows. - 有 pip,
python -m pip install dcm2niixon Linux, MacOS or Windows.
这两种方法安装最简单,但不一定有效。比如,我在 mac 上使用这两种方法安装后,转 nii 时,总会提示下图错误

但是在 linux 上这样安装是可用的。
如果上述安装方法不行,试试以下两种方法,稍微复杂一些。
- 下载 Github Releases 版本,提供了最新的可编译的执行文件。

下载对应的版本并解压,把可执行文件放入/Users/xxx/opt/anaconda3/bin/dcm2niix
为什么放在这个地方?计算机咱也不太懂,不过我们之前不是使用 conda 安装过 dcm2niix 吗,它就放在了这个位置。我想的是它自动安装的版本可能不太好用,我就用最新版本替换掉它,所以放在了此处,实际证明是可行的。

我们可以通过命令 which dcm2niix获取地址,并使用刚下载的 dcm2niix 替换掉它

- 运行以下命令获取Linux、Macintosh或Windows的最新版本
如果你不想从上述网站进去下载,也可以直接使用如下命令下载
curl -fLO https://github.com/rordenlab/dcm2niix/releases/latest/download/dcm2niix_lnx.zip (linux版)
curl -fLO https://github.com/rordenlab/dcm2niix/releases/latest/download/macos_dcm2niix.pkg (macos版)
curl -fLO https://github.com/rordenlab/dcm2niix/releases/latest/download/dcm2niix_win.zip (windows版)
如果是计算机玩家,还可以 build from source((using make or cmake)). 详细流程自行浏览 dcm2niix github
dcm2niix 命令行详细使用说明
dcm2niix 命令中唯一必填的参数是:需要转换的 DICOM 文件夹的位置,该参数始终作为最后一个参数进行提供。例如,dcm2niix ~/dicomdir 将会转换文件夹 “dicomdir” 中的所有 DICOM 文件。但是,你可以指定一些可选参数来影响转换结果。
因此,一个典型的转换命令可能是 dcm2niix -o ~/niftidir -f %p_%s -g y ~/dicomdir。
在这个例子中,软件会在 dicomdir 中查找 DICOM 图像并将转换后的 NIfTI 文件保存在 niftidir 中,输出文件将被压缩,并包含协议名称 (%p) 和序列号 (%s) 作为文件名。
下面是详细的参数列表(在终端直接输入 dcm2niix 命令来查看所有命令的列表)。以下是各个参数的说明:

- -1…-9 压缩级别(1为最快,9为最慢但会产生更小的文件,默认为6)。
- -b: BIDS sidecar:(“-b y”或“-b n”, 默认为 y)如果选择“yes”,则软件将生成与NIfTI图像同名的一种Brain Imaging Data Structure文件。该文件采用JSON文本格式,提供了有关扫描的附加信息,这些信息无法保存在NIfTI头文件中
-baBIDS sidecar的匿名化:(“-ba y”或“-ba n”)如果选择“no”,则BIDS文件将包含编码在DICOM文件中的参与者个人信息。这些识别信息可能会危及参与者的隐私。此选项仅影响BIDS sidecar(见b)。

- -f 输出文件名:(例如“-f myMR”, 默认 ‘%f_%p_%t_%s’)dcm2niix允许您指定转换后的 NIfTI 文件的命名方式。该方法可能一开始看起来很复杂,但它提供了非常大的灵活性。
一个简单的文件名可能是“exp03subj09”。
在这种情况下,这个参与者的第一个序列将被命名为exp03subj09.nii,
接下来是exp03subj09a.nii,然后是exp03subj09b.nii等等。
但是
dcm2niix还允许您将相关信息的存在和位置标注到文件名中。
考虑您指定“exp03subj09%p%s”
在这种情况下,协议的名称和序列号(存储在DICOM文件中)将成为文件名的一部分。
在这种情况下,输出文件的名称可能是exp03subj09fmri1.nii、
exp03subj09t12.nii
和exp03subj09fmri3.nii。
您可以按任意顺序组合尽可能多的修饰符。
以下是特殊修饰符的列表:
%a:插入天线(线圈)编号。例如,“myName%a”的输出文件名将生成“myName1”,“myName2”等,每个线圈一个。请注意,大多数扫描都会将所有线圈的数据组合在一起,在这些情况下,该选项将被忽略。例如,大多数将所有线圈的数据组合在一起的扫描通常只称为“myName”。
%d:插入系列描述(0008,103E)。例如,使用“myName%d”转换的回波平面图像会产生“myNameEPI”
%e:插入回波数。例如,具有两个回波时间的序列使用输出文件名“myName%e”进行转换,将产生“myName1”和“myName2”。请注意,大多数MRI序列仅使用单个回波时间,在这种情况下,您只会得到“myName1”。
%f:插入输入文件夹名。例如,使用输入文件夹“/usr/Subj22”和输出文件名“myName%f”将导致输出文件命名为“myNameSubj22.nii”。
%i:插入病人ID(DICOM标签0010,0020)。例如,“myName%i”的输出文件名将把病人ID命名为“ID123”的图像转换为“myNameID123.nii”
%m:制造商名称。 例如,“myName%m”的输出文件名将把来自GE扫描仪的图像转换为“myNameGE.nii”,而来自Philips的图像将变为“myNamePh.nii”,而Siemens将变为“myNameSi.nii”,否则制造商不可用(“myNameNA.nii”)。 (需要2015年或之后的dcm2nii版本)。
%n:插入受试者姓名(DICOM标签0010,0010)。例如,“myName%n”的输出文件名将把来自John Doe的图像转换为“myNameJohnDoe.nii”。如果您的参与者名称仅使用英文字母,则此选项最有效。对于其他欧洲语言,您可能会发现它进行一些基本转换(“Müller”将变成“Muller”)。对于非欧洲语言,您会发现此选项不满意。也许以后的版本可以支持DICOM标签0008,0005。
%p:插入协议名称(DICOM标签0018,1030)。例如,“myName%p”的输出文件名将把协议命名为T1的图像转换为“myNameT1.nii”
%q:插入序列名称(DICOM标签0018,1020)。例如,使用输出文件名“myName%q”将旋转回波序列转换为“myNameSE.nii”(新功能,在2015年8月30日版本中)。
%s:插入系列(DICOM标签0020,0011)。例如,使用输出文件名“myName%s”将第二个序列转换为“myName2.nii”。如果您想将系列号补零,请插入所需的数字数(0..9)。例如,在转换11个序列时应用过滤器“m%s”将创建文件,这些文件将导致简单的按字母排序出现问题,例如“m1.nii,m11.nii,m2.nii...m9.nii”的区别在于指定“m%3s”将有助于排序(例如“m001.nii,m002.nii...m011.nii”)。
%t:插入会话日期和时间(DICOM标签0008,0021和0008,0030)。例如,“myName%t”的输出文件名将把会话开始于2014年1月13日下午1:23的图像转换为“myName20140113132322.nii”
%z:插入序列名称(0018,0024),因此使用“myName%z”转换的T1扫描可能会产生“myNameT1”。
- -h 显示帮助屏幕
- -m: 合并2D切片:(n/y or 0/1/2, default 2) [no, yes, auto])如果选择,同一系列的图像将堆叠到单个NIfTI图像中. 默认就好了,3维数据直接在同一个 nii 里面。
- -o: 输出文件夹(不给,就会直接放在input folder)
- -z: 是否压缩,(y/o/i/n/3, default n) [y=pigz, o=optimal pigz, i=internal:zlib, n=no, 3=no,3D] 一般会选择压缩(-z y)
参数很多,但常用的转换命令如下(至少满足我的需求)
dcm2niix -z y -f %i_%d_%s -o niftidir dcmdir
表示将 dcmdir 的所有图像转换到 niftidir 并压缩图像,重命名为 %i_%d_%s
dcm2niix 我已经用了 2年以上了,团队里面转换都是用这个,转换快,需要的操作少。
但是朋友们刚开始接触需要花一点时间了解,我都做到这份上了,学习起来应该很容易吧。

你要是觉得这个工具不ok, 别急走!!!我还能在推荐10个
其他工具推荐
- dinifti: dinifti 下载链接, 用C语言编写的,专注于Siemens数据的转换
- dicm2nii: 用Matlab编写的。Matlab语言使得这个工具非常易于脚本化。它试图保留扩散梯度信息。dicm2nii 下载链接

- dicom2nifti: 看起来很有前途:可使用命令行,也可以在python中调用 dicom2nifti 下载链接。

- dcmstack: Python编写的。它具有一个巧妙的功能,可以让你在NIfTI文件中存储/查看DICOM元头数据。dcmstack下载链接

- mrconvert: MRtrix工具mrconvert是一个有用的通用图像转换器,并能够良好地处理DTI数据
- mri_convert: mri_convert下载链接是流行的FreeSurfer软件包的一部分。根据我的有限经验,这个工具适用于GE和Siemens数据,但在Philips 4D数据集上失败。
以下是不同工具处理数据所需花费的时间。dcm2niix为对照组,表里的时间是相对dcm2niix而言的

这么多工具,总有一个适合你吧。
文章持续更新,可以关注微信公众号【医学图像人工智能实战营】获取最新动态,一个关注于医学图像处理领域前沿科技的公众号。坚持已实践为主,手把手带你做项目,打比赛,写论文。凡原创文章皆提供理论讲解,实验代码,实验数据。只有实践才能成长的更快,关注我们,一起学习进步~
我是Tina, 我们下篇博客见~
白天工作晚上写文,呕心沥血
觉得写的不错的话最后,求点赞,评论,收藏。或者一键三连


























 1942
1942

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










