-基于R语言实例:预测海藻的数量
目录
问题描述与目标
描述:某些高浓度的有害藻类会对河流生态环境造成巨大影响。
目标:能够监测并在早期对海藻的繁殖进行预测以提高河流质量。
案例背景:针对这一问题,在大约一年的时间内,在不同时间收集了多条河流的水样,对于每个水样,测定了它们的不同化学性质以及7种有害藻类的存在频率。还有一些其他的数据,如季节、河流大小和水流的速度。
研究动机:本案例研究的主要动机之一是化学监测价格便宜,并且易于自动化。而通过分析生物样品来识别水中的藻类要涉及显微镜检验,需要训练有素的工作人员,因此既昂贵又缓慢。因此构建一个可以基于化学性质来准确预测藻类的模型来建立监测有害藻类的廉价自动化系统。
第一步、加载包和数据框以及读取海藻数据
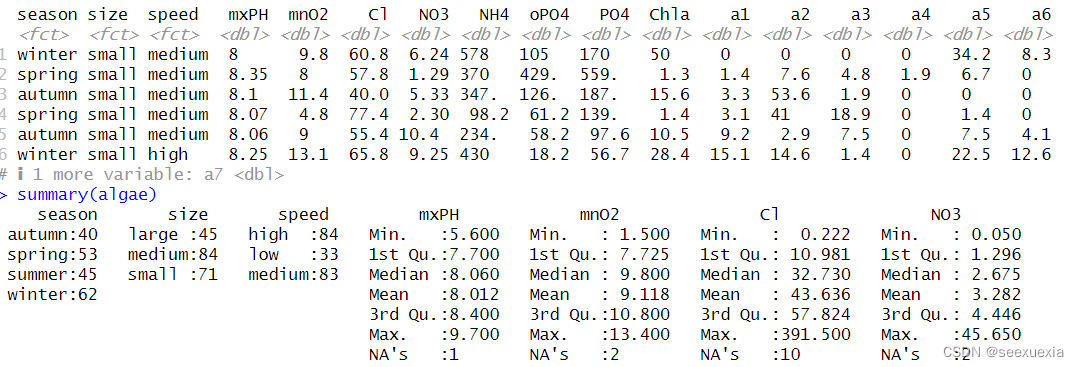
数据框可以看做一种有列名称的矩阵或者表格,它是存储R数据表的一种理想的数据结构。函数head讲显示数据框的前6行。其中,library()用于导入包,head()函数用于读取包中数据集,summary()函数:用于获取描述性统计量,可以提供最小值、最大值、四分位数和数值型变量的均值,以及因子向量和逻辑型向量的频数统计等。
autumn 40:表示秋季测量的数量有40次,lst Qu:表示四分之一中位数,Median:表示中位数,3rd Qu:表示四分三中位数,NA’S:表示由于某些原因没有采集到的数据。a1表示第一种有害海藻出现的频率名义变量
season : 季节 size : 河流规模 speed : 流速
mxPH : 最大PH值 mnO2 : 最小含氧量 Cl : 平均氯化物含量
NO3 : 平均硝酸盐含量 NH4 : 平均氨含量oPO4 : 平均正磷酸盐含量 PO4 : 平均硝酸盐含量 Chla : 平均叶绿素含量
第二步:处理数据的缺失问题
先将含有缺失值的数据查找出来
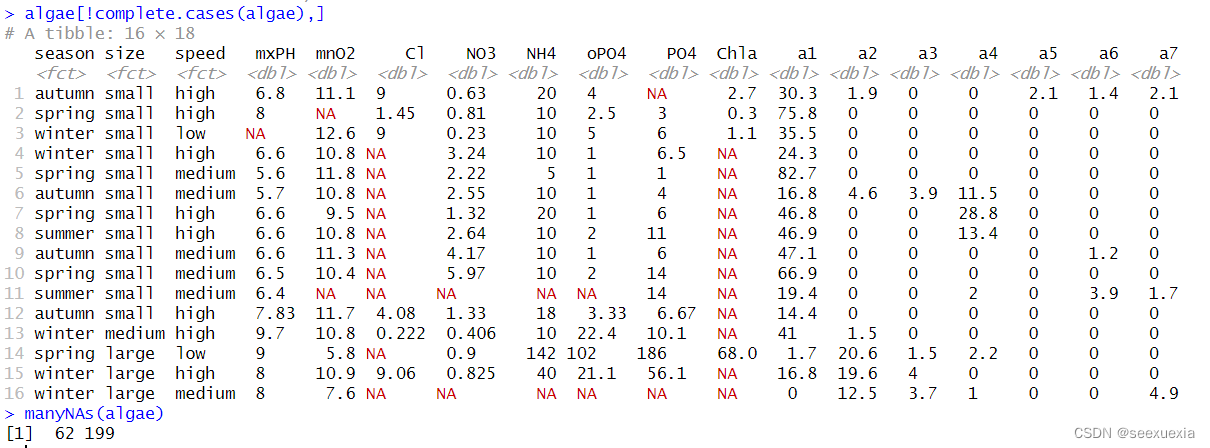
algae[!complete.cases(algae),]:返回含有缺失值的数据并给他们排序。参数 !complete.cases(algae) 是一个逻辑向量,它表示哪些行不是完整的案例。
manyNAs():函数manyNAs()的功能是找出缺失值个数大于20%的行
第一种:将含有缺失值的数据删除
剔除缺失值的操作非常容易实现,特别当缺失值在数据当中所占的比例比较小的时候,这个选择比较合理。
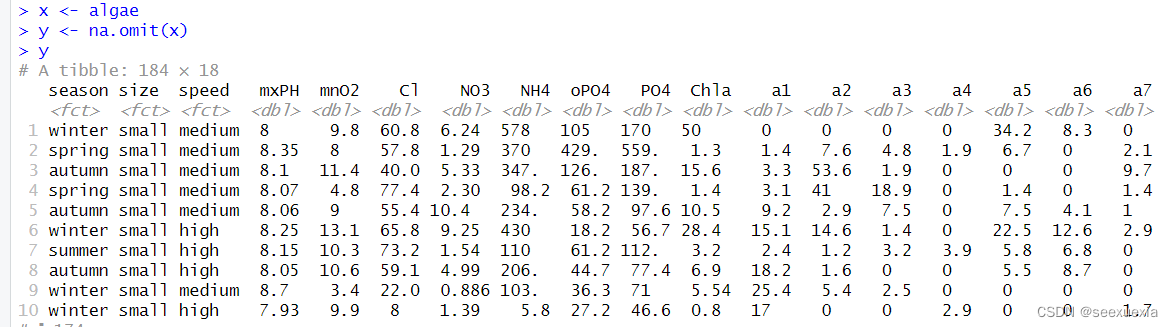
na.omit():将含有缺失值数据删除
这样我们就剔除了源数据中的所有含有缺失值的记录。但同时问题也浮出来了:数据分析的前提是有效数据越多越好。若果数据中含有缺失值的记录比例不低,那么这种做法会导致剩余数据的分析结果出现很大的偏差;
第二种:通过变量的相关关系填补缺失值
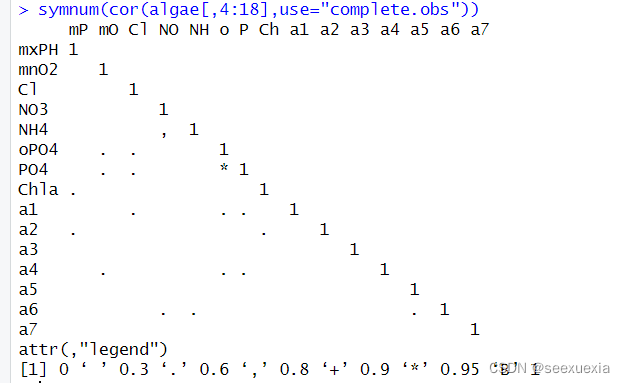
R 内置函数 cor 来计算 algae 数据框中第 4 到第 18 列之间的 Pearson 相关系数。然后我们将该相关系数矩阵传递给另一个名为 symnum 的自定义函数。这个函数会根据其内部实现来处理传入的相关系数矩阵,以改进输出的结果。
use="complete.obs" 参数告诉 cor 函数,在计算相关系数时只使用那些没有缺失值的数据点。这意味着如果有任何一列包含 NA 值,那么这一列将会被完全忽略,不会影响其他列的相关性计算。
0~0.3,用‘表示;0.3~0.6,用.表示;0.6~0.8,用,表示;0.8~0.9,用+表示;0.9~0.95用*表示。
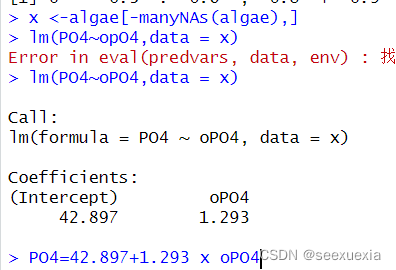
x <-algae[-manyNAs(algae),]:创建一个新的数据框,其中不包括那些包含大量缺失值的列。函数lm()可以用来获取形如Y=β0+β1χ1+…+βnχn的线性模型。上述结果得到的线性模型是:PO4 = 42.897 + 1.293 * oPO4。如果这两个变量不是同时有缺失值,那么通过这个模型计算这些变量的缺失值。
第三种方法:探索案例直接相似性来填补缺失值。
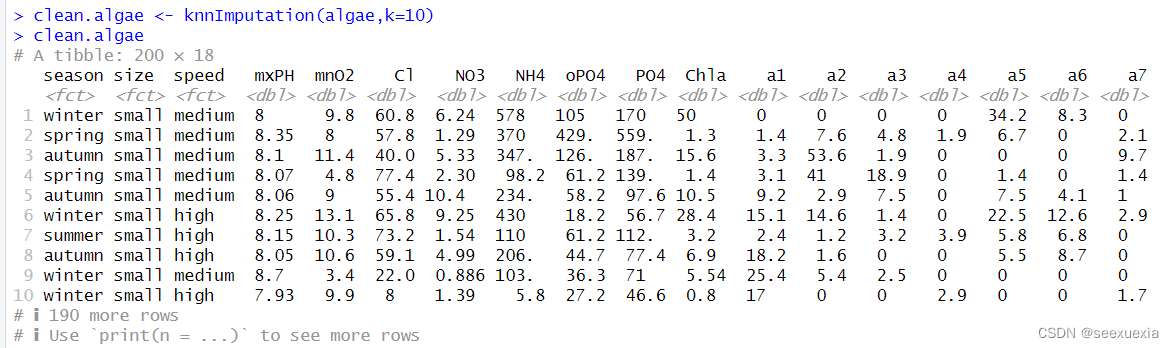
假设如果两个水样是相似的,其中一个水样在某些变量上有缺失值,那么该缺失值很可能与另外一个水样的值是相似的。为了使用这种直观的方法,首先定义相似性的概念。相似性经常由描述观察值的多元度量空间的变量所定义。在文献中有许多度量相似性的指标,常用的就是欧氏距离。knnImputation函数用欧氏距离来寻找距离个案最近的k个邻居,用中位数填补缺失值。
第三步:利用多元线性回归模型对数据进行预测
数据的模型简单的说就是一个函数,输入就是我们之前的数据algae,而输出就是我们预期的一些值。本案例的主要研究目的是预测140个水样中7种海藻的出现频率。假设海藻频率是数值型数据,因此可以考虑进行回归分析。简单地说,预测任务是建立一个模型来找到一个数值变量和一组解释变量的关系。这个模型既可以根据未来解释变量的值来预测目标变量,也可以帮助更好地理解问题中各个变量之间的相互联系。
多元线性回归模型
多元线性回归模型是最常用的统计数据分析方法,该模型给出了一个有关目标变量与一组解释变量关系的线性函数。
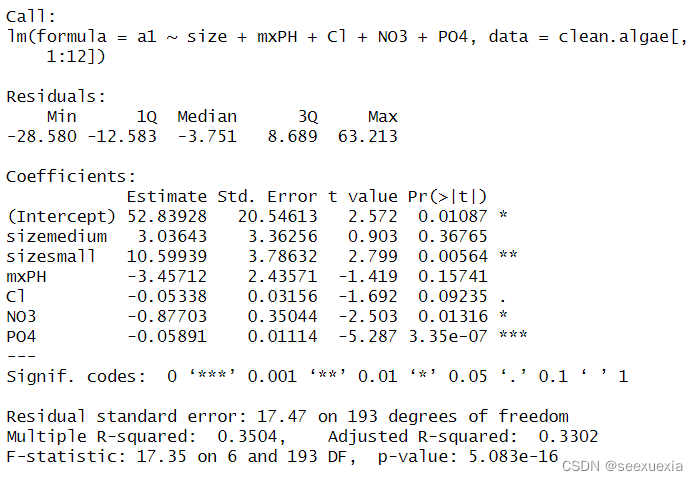
> lm.a1 <- lm(a1 ~ .,data = clean.algae[,1:12])
> summary(lm.a1)
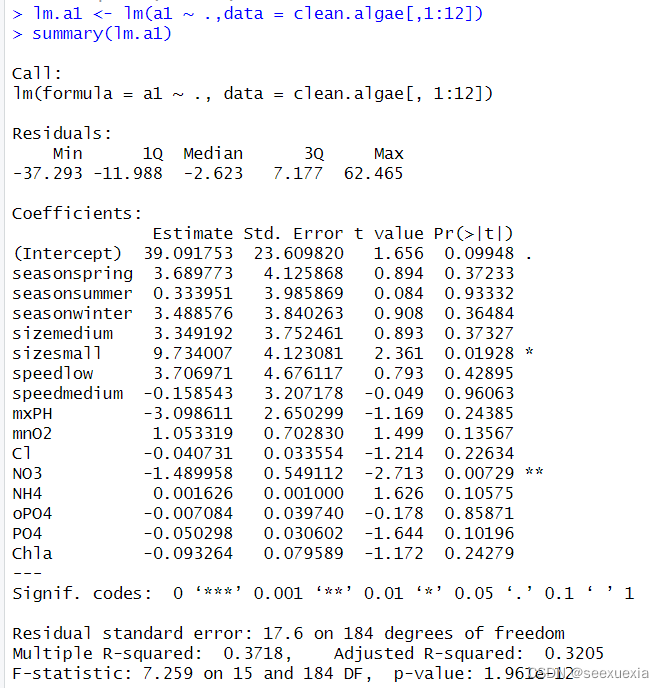
创建了一个新的线性模型对象 lm.a1 并将其赋值为对 clean.algae 数据框中第 1 到第 12 列之间的数据进行拟合的线性模型的结果。在 R 中,lm 函数是用于构建线性回归模型的内置函数。它可以接受两个主要参数:公式 (formula) 和数据 (data)。在这段代码中,我们使用了 . 符号作为公式参数,这意味着我们要预测的目标变量是 a1,而所有其他变量都是独立变量。data 参数指定了我们希望使用的数据数据框,也就是 clean.algae。clean.algae[,1:12] 就是从 clean.algae 数据框中选择前 12 列的结果。
R如何处理3个名义变量。当像上面一样进行模型构建时,R会生成一组的辅助变量,即对每一个有k个水平的因子变量,R会生成k-1个辅助变量。这些辅助变量的值为0或者1.当辅助变量的值为1,表明该因子出现,同时表明其他所有辅助变量的值为0。如果所有的k-1个变量的值都是0,则表明因子变量的取值为第k个剩余的值。在以上的汇总结果中,可以看到R为因子变量season生成了3个辅助变量(seasonspring, seasonsummer和seasonwinter)。如果某个水样的season变量的取值为”autumn”,则所有3个赋值变量的值将全部为零。
R^2表明模型与数据的吻合度,即数据所能解释的数据变差的比例。R^2越接近1(几乎100%地解释模型数据的变差)就说模型模拟得很好;R^2越小,说明模型拟合得越差。
此处为0.3205,还不是很理想,所以需要精简回归模型。
首先用anova函数提供模型拟合的方差序贯分析。
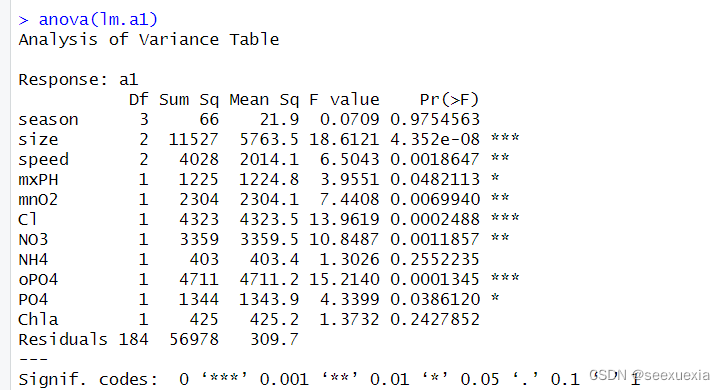
从图中可见,season对减少模型拟合误差的贡献最小,将其删除。然后再做一次线性回归模型。
lm2.a1<-update(lm.a1,.~.-season) #更新
summary(lm2.a1)
anova(lm.a1,lm2.a1)

尽管误差平方和减少了(-449),但显著性只有0.695,说明两个模型不同的可能性为30%,应该再次消元。使用step向后消元法。
final.lm<-step(lm.a1)
summary(final.lm)
最后的R平方仍然不理想,说明在此案例,应用线性回归模型并不合适。
第四步:模型预测
回归模型的预测性能是通过将目标变量的预测值与实际值进行比较得到的,并从这些比较中计算某些平均误差的度量。一种度量方法是平均绝对误差(MAE)。下面描述如何获得线性回归的平均绝对误差。第一步,获取需要评价模型预测性能的测试集个案的预测值。在R中,要获得任何模型的预测,就要使用函数predict()进行预测。函数predict()是一个泛型函数,它的一个参数为需要应用的模型,另一个参数为数据的测试集,输出结果为相应的模型预测值。
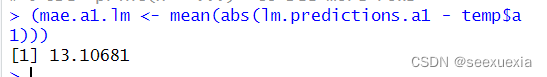
lm.predictions.a1 <- predict(final.lm, clean.algae)
# 计算平均绝对误差
(mae.a1.lm <- mean(abs(lm.predictions.a1 - temp$a1)))
# 可视化查看模型的预测值
old.par <- par(mfrow = c(1,2))
plot(lm.predictions.a1, algae[,'a1'], main="Linear Model", xlab="Predictions", ylab="True Values")
abline(0,1,lty=2)
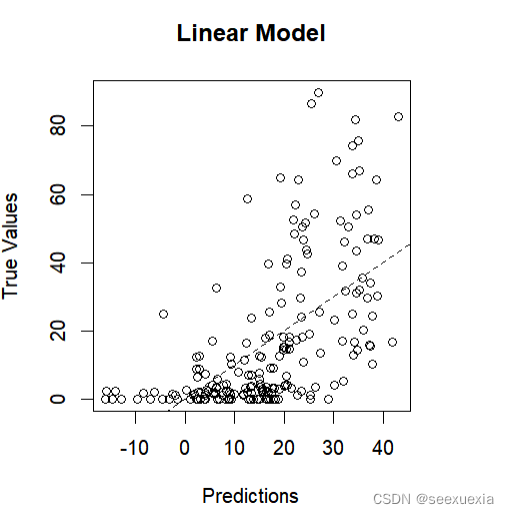
在理想的情况下,模型对所有的个案做出正确的预测时,图中的所有圈应该在虚线上,这条虚线是通过函数abline(0,1,lty=2)来绘制的。这条虚线穿过坐标系的原点,代表x坐标和y坐标相等的点集。图中圆圈的x坐标和y坐标分别代表目标变量的预测值和真实值,如果他们相等,那么这些圆圈就会落在这条理想的直线上。但从上图可以看到,情况并非如此!
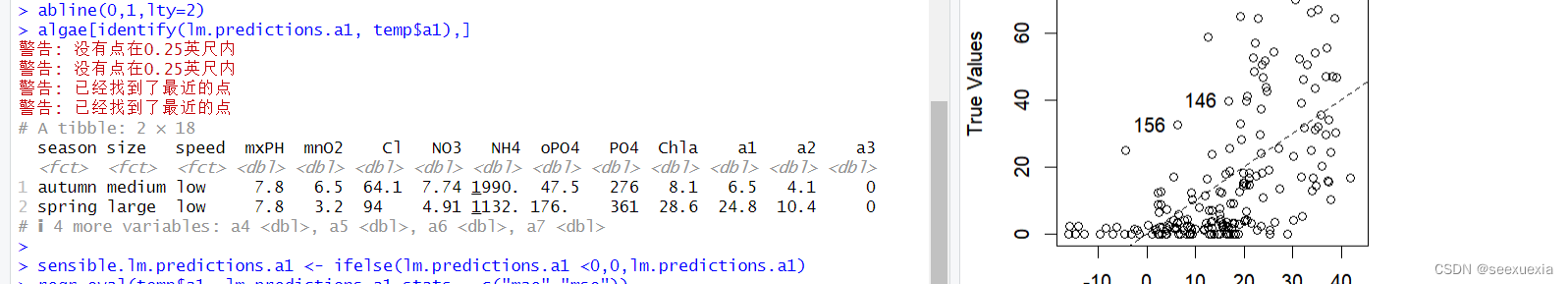
可以用函数identify()来检查哪些预测特别差的样本点,该函数可以让用户通过互动方式点击图形中的点
# identify()
plot(lm.predictions.a1, temp$a1,main="Linear Model", xlab="Predictions", ylab="True Values")
abline(0,1,lty=2)
algae[identify(lm.predictions.a1,temp$a1),]
运行上面的代码,并在图形上点击,然后右击结束交互过程后,应该看到相应于所点击的圆圈的海藻数据框的行数据–因为这里用函数identify()得到的向量来索引海藻数据框。
//本文只是作者个人理解,如果有不同意见,欢迎随时提出
本文参考链接:DMwR-note-01-预测海藻数量(四) | In Web3.0 We Trust (chenyuqing.github.io)





















 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








