安装和使用Entrez Direct软件
#到home目录下
cd
#等同于用~/ ,~/ 表示你的home目录
cd ~/
#创建/local/app目录来存放需要安装的程序
mkdir -p ~/local/app
#进入app目录
cd ~/local/app
获取entrez direct 工具包
#大写 -O 是使得curl 命令去识别url上的文件名(作为下载后的文件名字)
curl ftp://ftp.ncbi.nlm.nih.gov/entrez/entrezdirect/edirect.zip -O
#上一条命令等同于
curl ftp://ftp.ncbi.nlm.nih.gov/entrez/entrezdirect/edirect.zip -o edirect.zip
解压这个工具包
unzip edirect.zip查看新工具
cd edirect
ll
配置环境变量
echo "export PATH=\$PATH:\$HOME/local/app/edirect" >> $HOME/.bashrc
source ~/.bashrc
esearch -help
Option h is ambiguous (help, http, https)
Query Specification
-db Database name
-query Query string
Document Order
-sort Result presentation order
Date Constraint
-days Number of days in the past
-datetype Date field abbreviation
-mindate Start of date range
-maxdate End of date range
....................................................
#.bashrc 这个文件主要保存个人的一些个性化设置,如命令别名、路径等
运行 einfo
einfo -help
einfo 7.40
Database Selection
-db Database name
-dbs Get all database names
Data Summaries
-fields Print field names
-links Print link names
Field Example
<Field>
<Name>ALL</Name>
<FullName>All Fields</FullName>
.........................................................................
抓取描述信息,然后查看它们
einfo -dbs > einfo-dbs.txt
more einfo-dbs.txt
einfo -db sra > einfo-sra.txt
more einfo-sra.txt
运行esearch
esearch -help
esearch -db nucleotide -query PRJNA257197
#-db是指定数据库类型,而query是跟着你要搜索的关键词。
#在nucleotide这个数据库(database,简称db)里用关键词PRJNA257197搜索
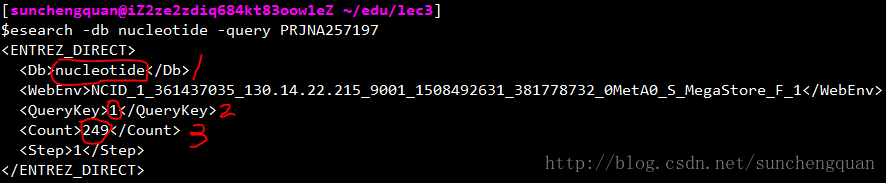
1:数据库类型为nucleotide,我们esearch的时候就指明了,我们要在nucleotide这个数据库=里搜索
2:搜索关键词数量:1(因为我们只输入了一个关键词,就是PRJNA257197)
3:搜索得到的条目有249个
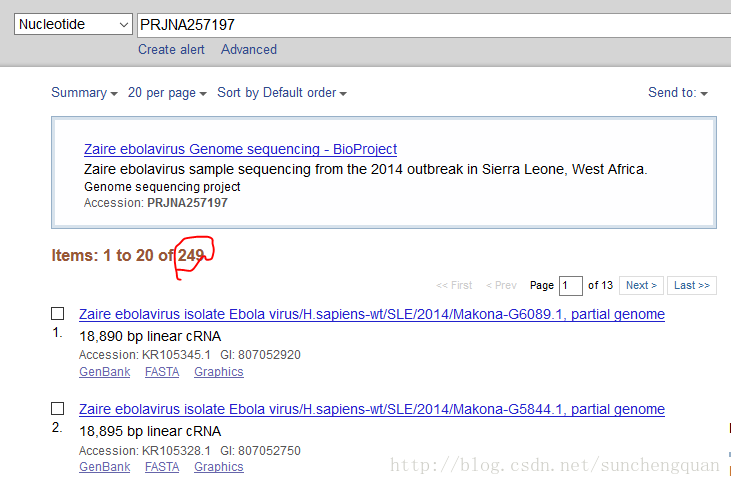
这一步相当于你到NCBI的首页,做了下图的操作:
并点了Search
看!果然是249条检索结果
抓取nucleotides数据
esearch -db nucleotide -query PRJNA257197 | efetch -format fasta > ~/edu/lec3/ebola.fasta看一下这个文件里有多少条序列
cat ~/edu/lec3/ebola.fasta | grep ">" | wc -l
249
cat ~/edu/lec3/ebola.fasta | wc -l
67603
less ~/edu/lec3/ebola.fasta
以GenBank格式获取数据
esearch -db nucleotide -query PRJNA257197 | efetch -format gb > ~/edu/lec3/ebola.gb
less ~/edu/lec3/ebola.gb
看一下这个文件里有多少条序列
cat ~/edu/lec3/ebola.gb | grep ^LOCUS | wc -l
249

























 1250
1250

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








