在进行医学图像器官分割时,无论我们是用传统方法还是深度学习,最后肯定要用到手动分割的区域作为金标准来验证我们方法的精度。但是手动标注器官的轮廓是十分耗时的,这里我介绍一个比较简单的标注方法,以肺分割作为例子。
我们使用ITK-snap作为分割标注工具。
当切片间距离比较小时,比如1mm,一个患者的完整HRCT序列通常在200张以上,如果让医生一一进行分割区域标注是非常耗时乏味的。因此,我们可以使用最简单的阈值法比如OSTU,加上基本的形态学操作对肺进行粗分割得到粗略的分割掩码,在此基础上再手动的对分割结果进行精分割,就能快速的得到一批金标准。
假设我们要对以下这张切片进行手动标注

首先打开粗分割结果,segmentation—>open segmentation。

由于这个掩码值为255,所以我们选择255的画笔进行修补,画笔大小和形状都可以修改,左键画图,右键去除。

假设下图为手动分割结果

最后将分割后的结果保存即可,segmentation—>save XXX as。
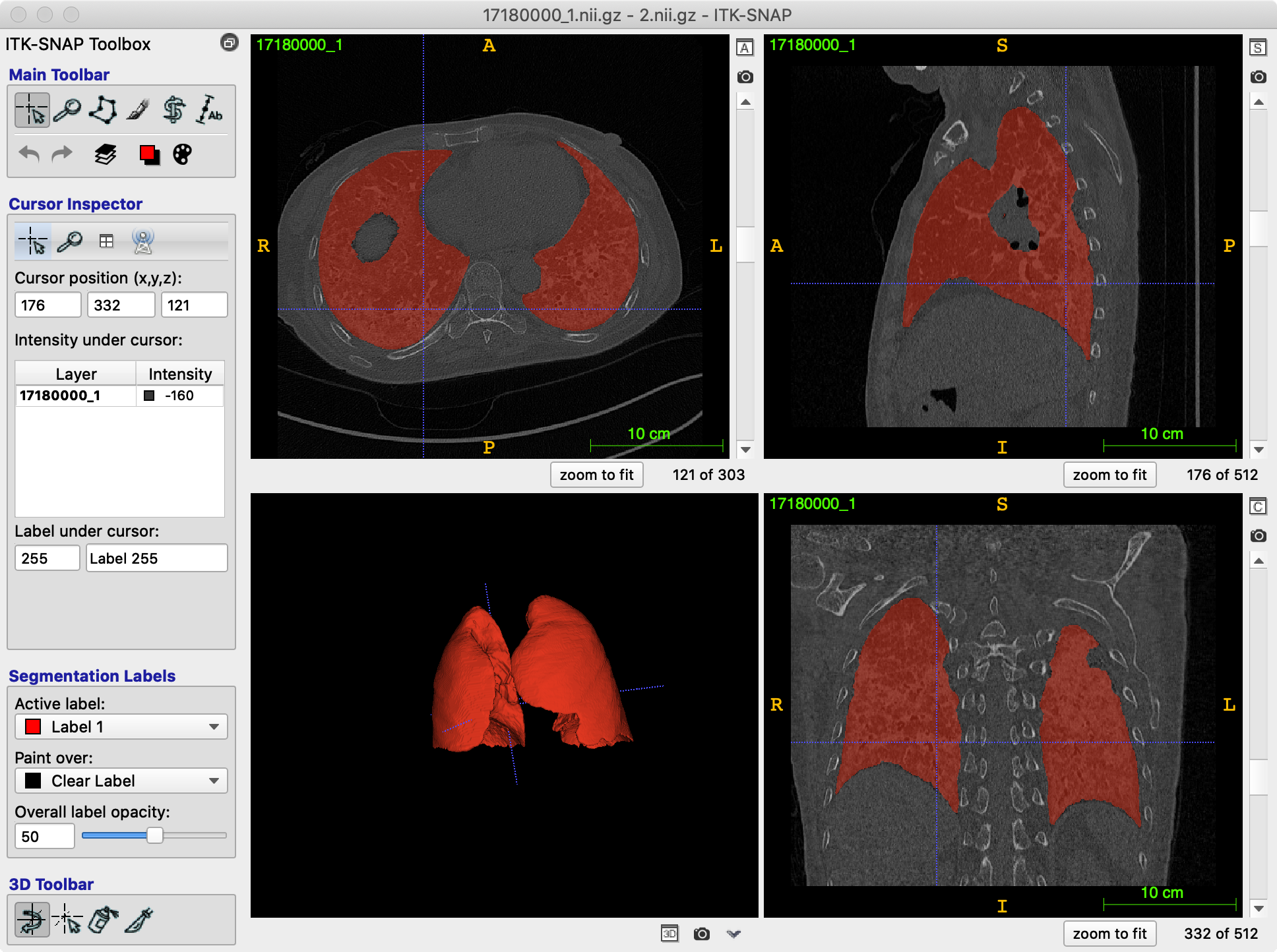
ITK-snap不能直接对原图进行三维显示,只能显示分割结果。因此我们首先打开原图,然后载入分割结果。然后在左下角将Continuous Update打钩就可以显示三维分割结果了。


有的时候分割结果可能有多个器官,我们需要进行不同颜色的显示,或者我们想改变某个分割器官的显示颜色。
只要打开label editor,改变激活标签的颜色就行,比如我这里的激活标签是Label 255。























 1万+
1万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








