L-M方法全称Levenberg-Marquardt方法,是一种非线性最小二乘优化算法,它通过同时利用高斯牛顿方法和梯度下降方法来解决非线性最小二乘问题。其核心思想是在每次迭代中,根据当前参数估计计算目标函数的梯度和海森矩阵,并使用这些信息来更新模型参数。
LM算法适用于解决各种非线性最小二乘问题,例如数据拟合、无约束非线性优化等。LM算法相对于其他算法的优势在于其自适应调整步长的能力,使得模型更容易收敛到最优解,并且可以避免梯度爆炸或消失的问题。
最近公司一直在研究大气污染物的检测技术,对采集数据后要进行拟合算法处理,试了很多,在机械机构和电子电路上也做了很多尝试,昨天对这个LM算法也在软件上测试了下,被逼无奈的,哈哈哈。其实数学原理,我是一窍不通。

以下是F = A * exp(t * B) 的LM算法的代码解释:
python代码实现LM算法
python
import numpy as np
from scipy.optimize import leastsq
# 定义目标函数
def func(x, p):
A, B = p
return A * np.exp( * )
# 定义残差函数
def residuals(p, y, t):
return y - func(t, p)
# 初始化参数
p0 = [1, 1]
t = np.linspace(0, 1, 10)
y = func(t, [2, -1]) + 0.2 * np.random.randn(len(t))
# 使用LM算法拟合模型
plsq = leastsq(residuals, p0, args=(y, t), ftol=1e-15, xtol=1e-15)
# 输出拟合结果
print(plsq[0]) # [1.99170234, -1.01074562]
上述代码中,我们首先定义了目标函数 func 和残差函数 residuals,然后初始化了待拟合的数据 t 和 y,以及初始参数值 p0。最后,我们使用 leastsq 函数对模型进行拟合,并将拟合结果输出到控制台中。
在使用LM算法时,需要预设收敛精度(ftol和xtol等参数),以及控制最大迭代次数的限制来保证计算效率和避免出现意外循环情况。
C++ 代码实现LM算法
#include <cmath>
#include <Eigen/Dense>
#include <unsupported/Eigen/NonLinearOptimization>
using namespace Eigen;
// 定义目标函数
struct Func {
int operator()(const VectorXd& x, VectorXd& fvec) const {
double A = x[0], B = x[1];
for (int i = 0; i < fvec.size(); ++i) {
fvec[i] = A * exp(x[2] * i) - B;
}
return 0;
}
};
// 初始化参数和数据
int main() {
VectorXd x(3);
x[0] = 2.0;
x[1] = 1.0;
x[2] = -1.0;
VectorXd y(10);
for (int i = 0; i < y.size(); ++i + 0.2 * std::rand()/RAND_MAX;
}
// 定义 LM 问题并求解
NumericalDiff<Func> numerical_diff;
LevenbergMarquardt<NumericalDiff<Func>> lm(numerical_diff);
lm.parameters.ftol = 1e-15;
lm.parameters.xtol = 1e-15;
lm.parameters.maxfev = 1000;
lm.minimize(x, y);
// 输出拟合结果
std::cout << "A = " << x[0] << ", B = " << x[1] << ", t = " << x[2] << std::endl;
return 0;
}
以上代码中,我们使用 Eigen 库来计算矩阵的逆和乘积,使用 NumericalDiff 类计算函数的一阶导数,使用 LevenbergMarquardt 类求解 LM 问题。其中,minimize 函数接收待拟合数据 y 和初始参数向量 x 作为输入,并可以自动调整 LM 算法的缩放因子 λ 和 μ,同时支持设置迭代次数限制、收敛精度等参数来控制算法行为。
以上两种是实现方法是人工智能生成的,有兴趣的小伙伴可以测试跑跑看。
自己测试过得代码实现LM算法:
#include "stdafx.h"
#include <cstdio>
#include <vector>
#include <opencv2/core/core.hpp>
#include <fstream>
#include <string>
using namespace std;
using namespace cv;
const double DERIV_STEP = 1e-4; // 拟合精度
const int MAX_ITER = 100; // 最大循环次数
void LM(double(*Func)(const Mat &input, const Mat params), // function pointer
const Mat &inputs, const Mat &outputs, Mat& params);
double Deriv(double(*Func)(const Mat &input, const Mat params), // function pointer
const Mat &input, const Mat params, int n);
// The user defines their function here
double Func(const Mat &input, const Mat params);
int main()
{
// For this demo we're going to try and fit to the function
// F = A*exp(t*B)
// There are 2 parameters: A B
int num_params = 2;
// Generate random data using these parameters
int total_data = 1410;
Mat inputs(total_data, 1, CV_64F);
Mat outputs(total_data, 1, CV_64F);
// //load observation data
// for (int i = 0; i < total_data; i++) {
// inputs.at<double>(i, 0) = i + 1; //load year
// }
// //load America population
// outputs.at<double>(0, 0) = 8.3;
// outputs.at<double>(1, 0) = 11.0;
// outputs.at<double>(2, 0) = 14.7;
// outputs.at<double>(3, 0) = 19.7;
// outputs.at<double>(4, 0) = 26.7;
// outputs.at<double>(5, 0) = 35.2;
// outputs.at<double>(6, 0) = 44.4;
// outputs.at<double>(7, 0) = 55.9;
for (int i = 0; i < total_data; i++)
{
inputs.at<double>(i, 0) = i * 0.05;
}
ifstream ifstxt;
ifstxt.open("shuju.txt");
string strline;
int iout = 0;
while (getline(ifstxt,strline))
{
outputs.at<double>(iout, 0) = stoi(strline.c_str());
iout++;
}
ifstxt.close();
///
// Guess the parameters, it should be close to the true value, else it can fail for very sensitive functions!
Mat params(num_params, 1, CV_64F);
//init guess
params.at<double>(0, 0) = 14000;
params.at<double>(1, 0) = -0.05;
LM(Func, inputs, outputs, params);
printf("Parameters from LM: %lf %lf\n", params.at<double>(0, 0), params.at<double>(1, 0));
system("pause");
return 0;
}
double Func(const Mat &input, const Mat params)
{
// Assumes input is a single row matrix
// Assumes params is a column matrix
double A = params.at<double>(0, 0);
double B = params.at<double>(1, 0);
double x = input.at<double>(0, 0);
return A*exp(x*B);
}
//calc the n-th params' partial derivation , the params are our final target
double Deriv(double(*Func)(const Mat &input, const Mat params), const Mat &input, const Mat params, int n)
{
// Assumes input is a single row matrix
// Returns the derivative of the nth parameter
Mat params1 = params.clone();
Mat params2 = params.clone();
// Use central difference to get derivative
params1.at<double>(n, 0) -= DERIV_STEP;
params2.at<double>(n, 0) += DERIV_STEP;
double p1 = Func(input, params1);
double p2 = Func(input, params2);
double d = (p2 - p1) / (2 * DERIV_STEP);
return d;
}
void LM(double(*Func)(const Mat &input, const Mat params),
const Mat &inputs, const Mat &outputs, Mat& params)
{
int m = inputs.rows;
int n = inputs.cols;
int num_params = params.rows;
Mat r(m, 1, CV_64F); // residual matrix
Mat r_tmp(m, 1, CV_64F);
Mat Jf(m, num_params, CV_64F); // Jacobian of Func()
Mat input(1, n, CV_64F); // single row input
Mat params_tmp = params.clone();
double last_mse = 0;
float u = 1, v = 2;
Mat I = Mat::ones(num_params, num_params, CV_64F);//construct identity matrix
int i = 0;
for (i = 0; i < MAX_ITER; i++)
{
double mse = 0;
double mse_temp = 0;
for (int j = 0; j < m; j++)
{
for (int k = 0; k < n; k++)
{//copy Independent variable vector, the year
input.at<double>(0, k) = inputs.at<double>(j, k);
}
r.at<double>(j, 0) = outputs.at<double>(j, 0) - Func(input, params);//diff between previous estimate and observation population
mse += r.at<double>(j, 0)*r.at<double>(j, 0);
for (int k = 0; k < num_params; k++) {
Jf.at<double>(j, k) = Deriv(Func, input, params, k); //construct jacobian matrix
}
}
mse /= m;
//printf("%lf\n", mse /= m);
params_tmp = params.clone();
Mat hlm = (Jf.t()*Jf + u*I).inv()*Jf.t()*r; //calculate deta
params_tmp += hlm; //update value
for (int j = 0; j < m; j++) {
r_tmp.at<double>(j, 0) = outputs.at<double>(j, 0) - Func(input, params_tmp);//diff between current estimate and observation population
mse_temp += r_tmp.at<double>(j, 0)*r_tmp.at<double>(j, 0);//diff square sum
}
mse_temp /= m;//diff square sum
Mat q(1, 1, CV_64F);
q = (mse - mse_temp) / (0.5*hlm.t()*(u*hlm - Jf.t()*r));
double q_value = q.at<double>(0, 0);
if (q_value > 0)
{
double s = 1.0 / 3.0;
v = 2;
mse = mse_temp;
params = params_tmp;
double temp = 1 - pow(2 * q_value - 1, 3);
if (s > temp)
{
u = u * s;
}
else
{
u = u * temp;
}
}
else
{
u = u*v;
v = 2 * v;
params = params_tmp;
}
// The difference in mse is very small, so quit
if (fabs(mse - last_mse) < 1e-5) // 这个值得大小,影响循环跳出,计算精度(考虑计算时间问题)
{
printf("%d %lf\n", i, mse);
break;
}
//printf("%d: mse=%f\n", i, mse);
//printf("%d %lf\n", i, mse);
last_mse = mse;
}
}
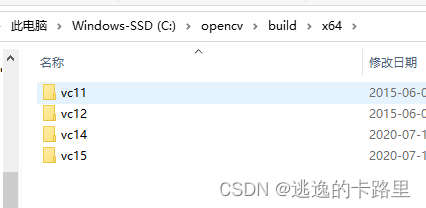
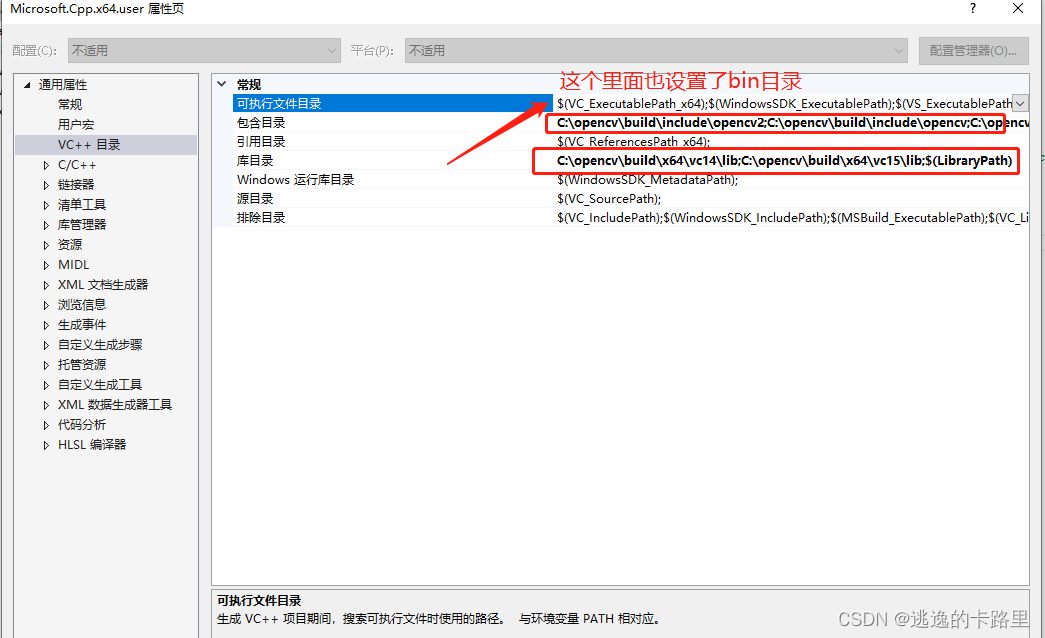
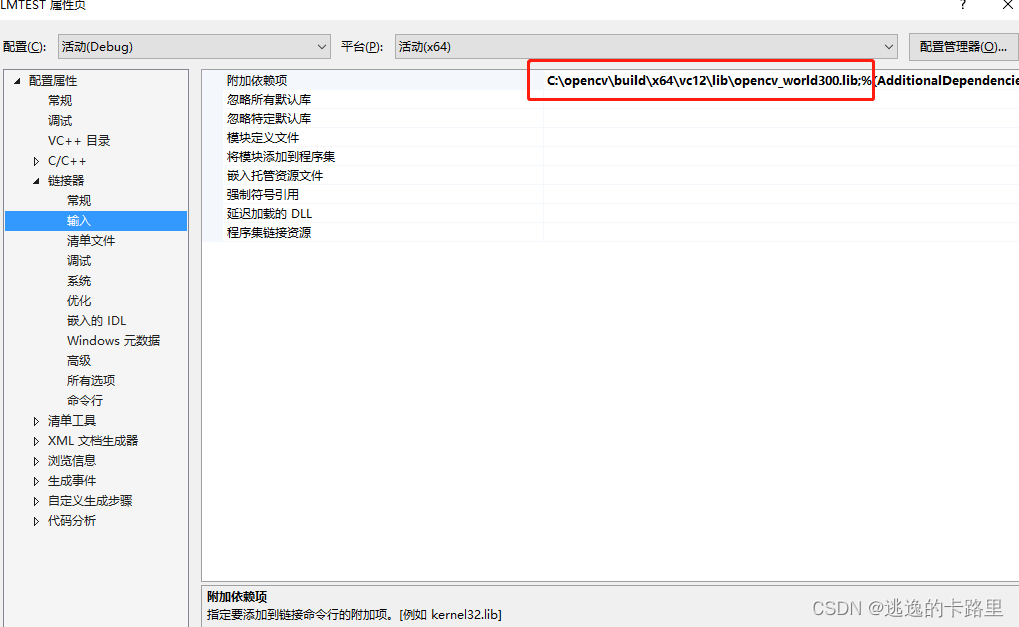
在配置#include <opencv2/core/core.hpp>,这个的时候会遇到一些问题,小伙伴可以在网上自己搜下解决办法,
下面是我再vs2015中设置的截图:电脑上安装两个版本OpenCV,设置有点乱

























 3万+
3万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










