VIPdb简介
VIPdb(Variant Impact Predictor database)是一个专门收集和总结变异影响预测工具(Variant Impact Predictors,简称VIPs,也称为Variant Effect Predictors,简称VEPs)的数据库。这些工具用于帮助科研人员和医生从基因组中检测到的数百万遗传变异中识别出可能致病的变异。
VIPdb第二版汇集了过去25年开发的VIPs,提供了它们的特性、ClinGen校准分数、CAGI评估结果、出版详情、访问信息以及引用模式。在2019年的基础上,VIPdb v2新增了186个VIPs,总计达到403个。VIPs主要设计用来预测遗传变异的影响,包括单核苷酸变异、插入和删除变异、结构变异、非同义/终止变异、同义变异、剪接变异和调控区域变异等。

VIPdb v2地址: https://genomeinterpretation.org/vipdb.html
预印版文章: https://www.biorxiv.org/content/10.1101/2024.06.25.600283v1.full.pdf
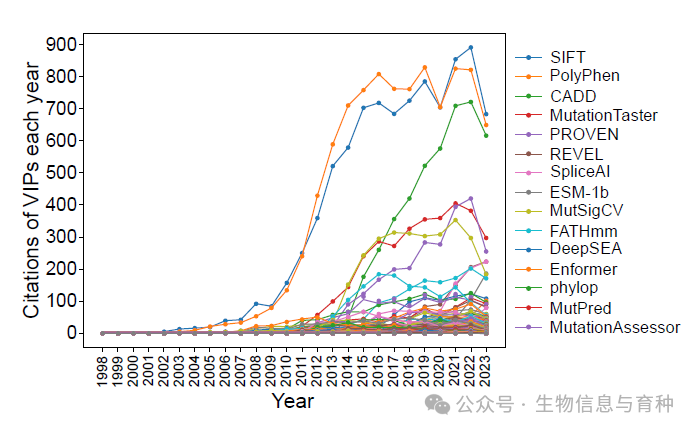
Citation trend of 274 core VIPs (1998 to 2023)

Citation trend of the top 15 most cited core VIPs in the year 2023
致病变异/有害变异与植物育种
VEP 更多用于人类基因组变异注释,已知的变异信息形成了强大的数据库。在动植物研究领域,应用最多的是snpEff、Annovar等工具,这些一开始也只是用在人类基因组上。
所以,植物生物信息研究的很多方法都来自于人类,有害突变也不例外。有害突变在植物中的研究很少,但很重要,尤其是对自交不亲和或自交衰退比较严重的作物而言,有人认为考虑有害突变才是GS的正确之道(存疑)。这是一个新的研究思路,大家都可去尝试,只是发表了大作不要忘了感谢我~
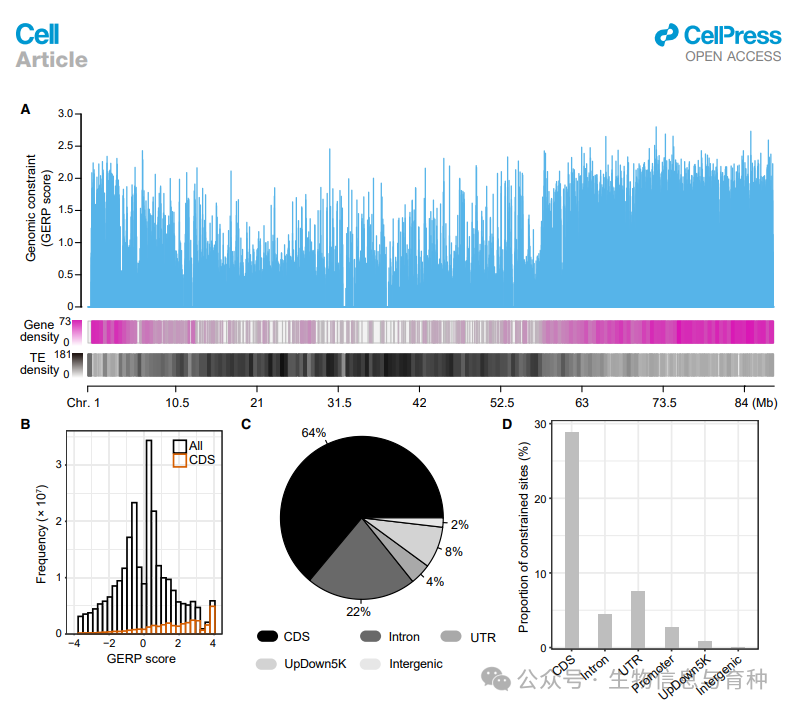
比如,2023年黄三文院士通过这个思路绘制首个马铃薯有害突变二维图谱,发表Cell(Phylogenomic discovery of deleterious mutations facilitates hybrid potato breeding)。而更早前(2021)一位韩国思密达研究了大豆驯化过程中有害突变的模式,发表在NC(The patterns of deleterious mutations during the domestication of soybean)。他们都是用的一种基因组进化速率分析(Genomic Evolutionary Rate Profiling,GERP)的方法,该方法/工具已经发表14年了(Identifying a high fraction of the human genome to be under selective constraint using GERP++),一开始也是用在人类基因组上。
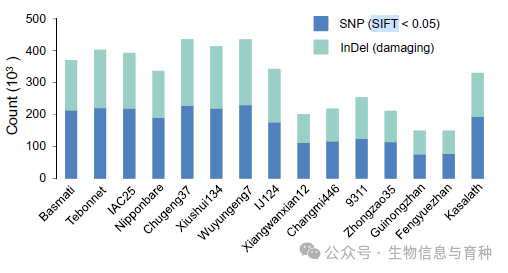
前几天,黄学辉老师发表Science的文章里(Genomi__c investigation of 18,421 lines reveals the genetic architecture of rice),也用SIFT来定量评估了突变对基因功能可能产生的影响。
所以很多植物上的研究方法其实一点都不新鲜,很多都是拿来主义,极少有专门针对动植物基因组特点开发的分析算法和软件,tassel算是一个典范。相比于人类医学研究,落后了太多。
记得今年在某次南京召开的学术会议上,黄院士做了优薯计划报告,提到了有害突变图谱鉴定这种“新”方法,严校长问(大意):如何知道鉴定的有害突变是全面的?还有更好的方法吗?这个问题当时在作物界,应该很少人能回答吧。看看,生信算法和工具有多重要。如果审稿人是做生信的,也很较真,那么CNS可能没那么顺利发表。
有害突变对植物育种研究,尤其是GS,可能是一个值得探索的方向,大家也可以去尝试。如果看了我们的推文启发你选育出新品种,也不要忘了回来感谢我呀~
附Cell作者吴教授的GERP分析代码:
Github地址: https://github.com/yywyaoyaowu/SolEvo_PotatoDele
❝
声明:本人对大佬及其大作没有任何的不敬和质疑之意,只是作为一个例子来说明植物育种研究的生信方法开发任重道远,请勿过分解读。
作者:生物信息与育种,若要及时了解动态信息,请关注同名微信公众号:生物信息与育种。





















 96
96

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








