Core idea
LLE is inherently a non-linear dimensionality reduction strategy
即局部线性嵌入算法。该算法是针对非线性信号特征矢量维数的优化方法,这种维数优化并不是仅仅在数量上简单的约简,而是在保持原始数据性质不变的情况下,将高维空间的信号映射到低维空间上,即特征值的二次提取。
Charateristics: neighborhood-preserving
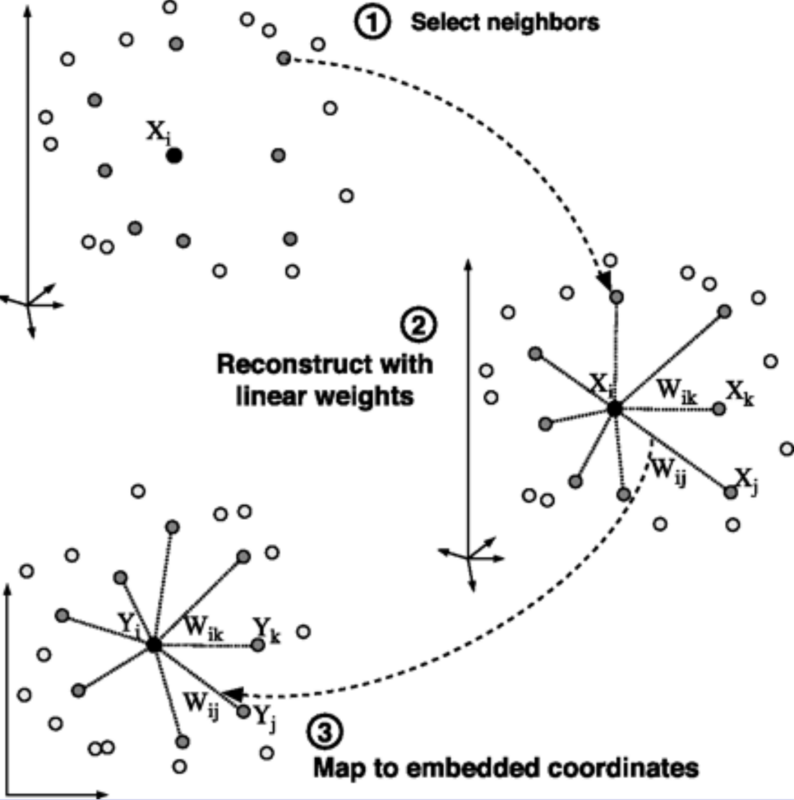
Steps
Select neighbors
Reconstruct with linear weights
Map to embedded coordinates
Basic formulas

How to translate this problem to eigenvalue solution
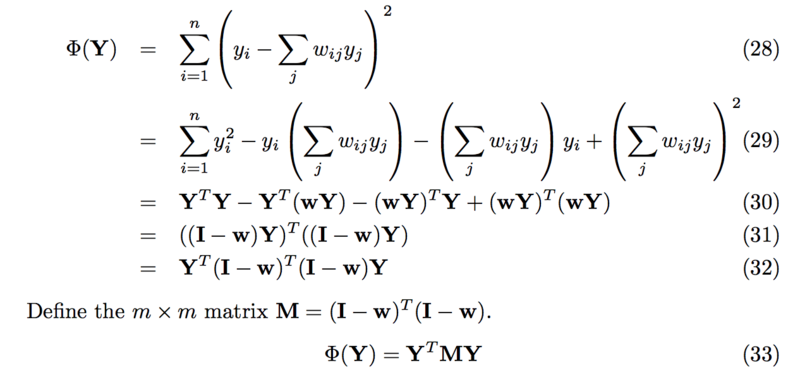
Use a Lagrange multiplier
Graph Embedding
寻找neighborhood:直接用Graph的邻接结构表示neighborhood
计算linear weights:直接用邻接矩阵W
生成embedding:计算矩阵M特征值,当节点数为n,embedding为q维时,取[n-q, n-1]的特征向量为embedding结果
Code
simple_code:
Python
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets, manifold
from mpl_toolkits.mplot3d import Axes3D
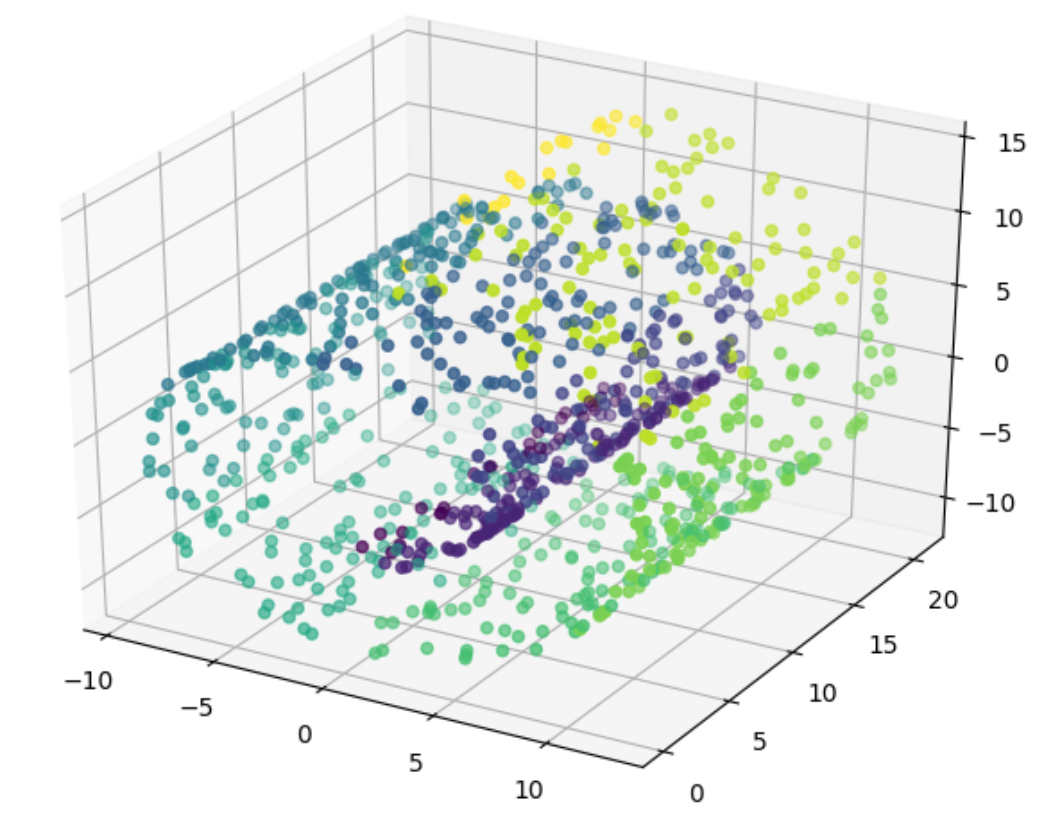
swiss_roll = datasets.make_swiss_roll(n_samples=1000)
X = swiss_roll[0]
Y = np.floor(swiss_roll[1])
d = 3
k = 999
fig_original = plt.figure('swiss_roll')
ax = Axes3D(fig_original)
ax.scatter(X[:, 0], X[:, 1], X[:, 2], marker='o', c=Y)
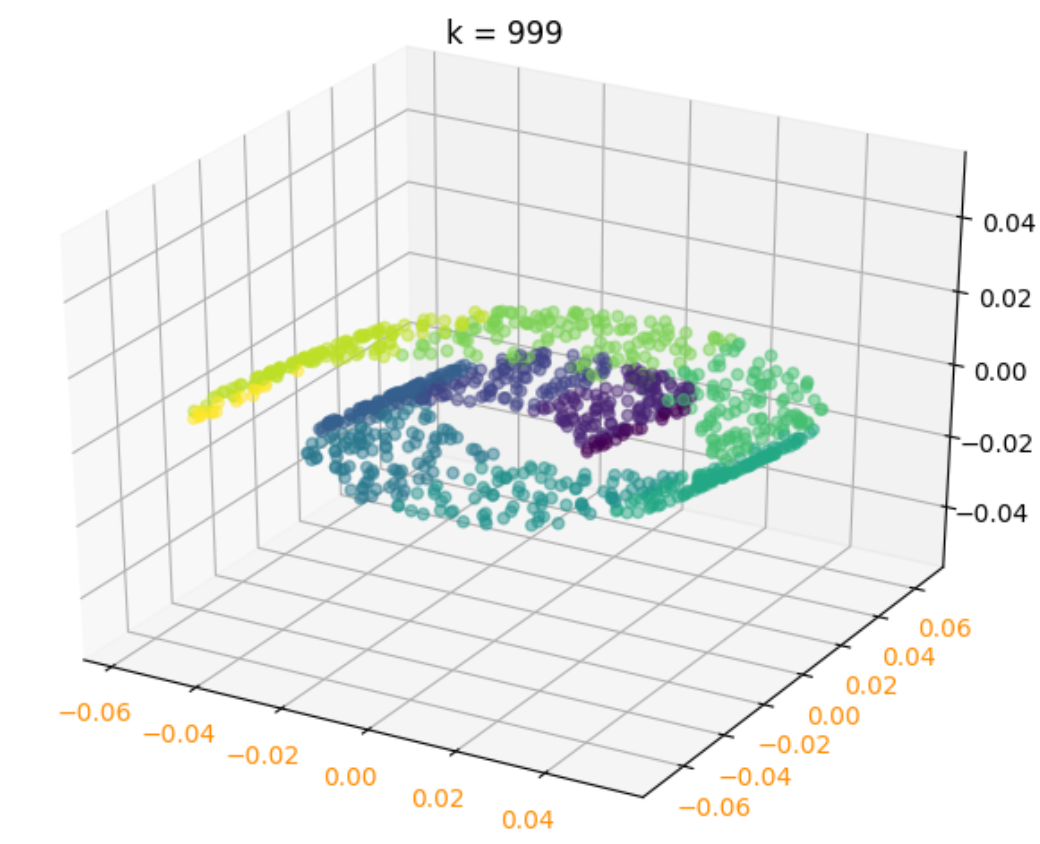
LLE = manifold.LocallyLinearEmbedding(n_components=d, n_neighbors=k, eigen_solver='auto')
X_r = LLE.fit_transform(X)
fig = plt.figure('LLE')
ax = Axes3D(fig)
ax.scatter(X_r[:, 0], X_r[:, 1], marker='o', c=Y, alpha=0.5)
ax.set_title("k = %d" % k)
plt.xticks(fontsize=10, color='darkorange')
plt.yticks(fontsize=10, color='darkorange')
plt.show()
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
importnumpyasnp
importmatplotlib.pyplotasplt
fromsklearnimportdatasets,manifold
frommpl_toolkits.mplot3dimportAxes3D
swiss_roll=datasets.make_swiss_roll(n_samples=1000)
X=swiss_roll[0]
Y=np.floor(swiss_roll[1])
d=3
k=999
fig_original=plt.figure('swiss_roll')
ax=Axes3D(fig_original)
ax.scatter(X[:,0],X[:,1],X[:,2],marker='o',c=Y)
LLE=manifold.LocallyLinearEmbedding(n_components=d,n_neighbors=k,eigen_solver='auto')
X_r=LLE.fit_transform(X)
fig=plt.figure('LLE')
ax=Axes3D(fig)
ax.scatter(X_r[:,0],X_r[:,1],marker='o',c=Y,alpha=0.5)
ax.set_title("k = %d"%k)
plt.xticks(fontsize=10,color='darkorange')
plt.yticks(fontsize=10,color='darkorange')
plt.show()
another version:
Python
import numpy as np
import matplotlib.pyplot as plt
from sklearn import datasets, decomposition, manifold
from mpl_toolkits.mplot3d import Axes3D
def load_data():
'''
从sklearn中读取swiss_roll数据集
:return:
'''
swiss_roll = datasets.make_swiss_roll(n_samples=1000)
return swiss_roll[0], np.floor(swiss_roll[1])
def LLE_components(*data):
X, Y = data
for n in [3, 2, 1]: # 最终的降维目标
lle = manifold.LocallyLinearEmbedding(n_components=n) # sklearn的LLE方法
lle.fit(X)
print("n = %d 重建误差:" % n, lle.reconstruction_error_)
def LLE_neighbors(*data):
X, Y = data
Neighbors = [1, 2, 3, 4, 5, 15, 30, 100, Y.size - 1] # 可以选择的几个邻域值
fig = plt.figure("LLE", figsize=(9, 9))
for i, k in enumerate(Neighbors):
lle = manifold.LocallyLinearEmbedding(n_components=2, n_neighbors=k, eigen_solver='dense')
'''
eigen_solver:特征分解的方法。有‘arpack’和‘dense’两者算法选择。
当然也可以选择'auto'让scikit-learn自己选择一个合适的算法。
‘arpack’和‘dense’的主要区别是‘dense’一般适合于非稀疏的矩阵分解。
而‘arpack’虽然可以适应稀疏和非稀疏的矩阵分解,但在稀疏矩阵分解时会有更好算法速度。
当然由于它使用一些随机思想,所以它的解可能不稳定,一般需要多选几组随机种子来尝试。
'''
X_r = lle.fit_transform(X)
ax = fig.add_subplot(3, 3, i + 1)
ax.scatter(X_r[:, 0], X_r[:, 1], marker='o', c=Y, alpha=0.5)
ax.set_title("k = %d" % k)
plt.xticks(fontsize=10, color="darkorange")
plt.yticks(fontsize=10, color="darkorange")
plt.suptitle("LLE")
plt.show()
X, Y = load_data()
fig = plt.figure('data')
ax = Axes3D(fig)
ax.scatter(X[:, 0], X[:, 1], X[:, 2], marker='o', c=Y)
LLE_components(X, Y)
LLE_neighbors(X, Y)
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
importnumpyasnp
importmatplotlib.pyplotasplt
fromsklearnimportdatasets,decomposition,manifold
frommpl_toolkits.mplot3dimportAxes3D
defload_data():
'''
从sklearn中读取swiss_roll数据集
:return:
'''
swiss_roll=datasets.make_swiss_roll(n_samples=1000)
returnswiss_roll[0],np.floor(swiss_roll[1])
defLLE_components(*data):
X,Y=data
fornin[3,2,1]:# 最终的降维目标
lle=manifold.LocallyLinearEmbedding(n_components=n)# sklearn的LLE方法
lle.fit(X)
print("n = %d 重建误差:"%n,lle.reconstruction_error_)
defLLE_neighbors(*data):
X,Y=data
Neighbors=[1,2,3,4,5,15,30,100,Y.size-1]# 可以选择的几个邻域值
fig=plt.figure("LLE",figsize=(9,9))
fori,kinenumerate(Neighbors):
lle=manifold.LocallyLinearEmbedding(n_components=2,n_neighbors=k,eigen_solver='dense')
'''
eigen_solver:特征分解的方法。有‘arpack’和‘dense’两者算法选择。
当然也可以选择'auto'让scikit-learn自己选择一个合适的算法。
‘arpack’和‘dense’的主要区别是‘dense’一般适合于非稀疏的矩阵分解。
而‘arpack’虽然可以适应稀疏和非稀疏的矩阵分解,但在稀疏矩阵分解时会有更好算法速度。
当然由于它使用一些随机思想,所以它的解可能不稳定,一般需要多选几组随机种子来尝试。
'''
X_r=lle.fit_transform(X)
ax=fig.add_subplot(3,3,i+1)
ax.scatter(X_r[:,0],X_r[:,1],marker='o',c=Y,alpha=0.5)
ax.set_title("k = %d"%k)
plt.xticks(fontsize=10,color="darkorange")
plt.yticks(fontsize=10,color="darkorange")
plt.suptitle("LLE")
plt.show()
X,Y=load_data()
fig=plt.figure('data')
ax=Axes3D(fig)
ax.scatter(X[:,0],X[:,1],X[:,2],marker='o',c=Y)
LLE_components(X,Y)
LLE_neighbors(X,Y)





















 1920
1920











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








