目录
总结
前言
在学习一篇医学影像处理的综述,从中挑几篇觉得感兴趣的进行深度学习。第一篇是:Chen S , Ma K , Zheng Y . Med3D: Transfer Learning for 3D Medical Image Analysis[J]. 2019. 即三维医学图像预训练模型MED 3D(MedicalNet)
3D 医学成像中的数据采集和注释困难,作者汇总了来自多个医学挑战的数据集,构建具有多种模式、靶器官和病理学的 3DSeg-8 数据集。为了提取一般医学三维(3D)特征,设计了一个名为 Med3D 的异构 3D 网络来共同训练多域 3DSeg-8,从而制作一系列预训练模型。
综述:S. K. Zhou et al., "A Review of Deep Learning in Medical Imaging: Imaging Traits, Technology Trends, Case Studies With Progress Highlights, and Future Promises," in Proceedings of the IEEE, vol. 109, no. 5, pp. 820-838, May 2021, doi: 10.1109/JPROC.2021.3054390.
一、介绍
作者的主要工作是构建一个大型的 3D 医疗数据集,并用这些数据训练一个基础模型,以解决其他医学问题。
第一步,从不同的医学领域收集了许多具有不同成像方式、靶器官和病理表现的小规模数据集,以构建一个相对较大的 3D 医学图像数据集。
第二步,建立了一个名为 Med3D 的编码-解码分割网络,该网络使用上述大规模 3D 医学数据集进行了异构训练。还提出了一种多分支解码器来解决不完整的注释问题。
与基于 Kinetics 视频数据集的 3D 模型和从头训练的模型相比,我们还将提取的 Med3D 通用编码器转移到肺分割、结节分类和 LiTS 竞赛。实验证明了基于我们提出的预训练模型的训练收敛效率和准确性(见图 1)

作者工作的贡献总结如下:(1)我们提出了一种针对3D多域医学数据的异构Med3D网络,即使在数据域分布差异很大的情况下也可以提取一般的3D特征。 (2) 我们将 Med3D 模型的主干转移到三个新的 3D 医学图像任务中。 而且,我们通过大量实验证实了 Med3D 的有效性和效率。 (3) 为方便社区复现我们的实验结果并将 Med3D 应用到其他应用程序中,我们将发布我们的 Med3D 预训练模型和相关源代码,地址:https://github.com/Tencent/MedicalNet.
二、方法
作者工作的动机是用相对较大的 3D 医疗数据集训练高性能 DCNN 模型,该模型可用作主干预训练模型,以提升由于训练数据不足的其他任务的效果。为了达到这个目标,作者设计了一个包含三个主要步骤的处理工作流程,如图 2 所示。第一步,从不同的医学成像模式中收集了几个名为 3DSeg-8 的公开可用的 3D 分割数据集,具有各种扫描区域、靶器官和病理,例如:磁共振成像 (MRI) 和计算机断层扫描 (CT)。然后,对具有相同空间和强度分布的所有数据进行归一化。第二步,训练一个 DCNN 模型,即 Med3D,来学习特征。该网络为每个特定数据集提供一个共享编码器和八个简单的解码器分支。第三步,从预训练的 Med3D 模型中提取的特征被转移到其他医疗任务中,以提高网络性能。

1.数据选择和归一化
数据是从八个不同的 3D 医学分割数据集中收集的,为了以后的方便,将组合数据集称为 3DSeg-8。 从分割数据集中选择数据而不是分类数据的原因主要有两个方面:首先,与可以从数千个对象类别中学习一般特征表示的自然图像分析不同,医学成像分析在有限的身体区域上进行操作,该区域有很多更少的对象类。 在分类任务中从一小组类别中学习可能会导致泛化能力差。 其次,与自然图像标签相比,分类标签对于 3D 医学数据的监督信息变得更弱,因为标签可能只对应于存在于一小部分体数据中的组织/器官,这可能会阻碍图像的神经网络学习过程。 因此,从这八个分割数据集中选择数据,并相信从分割任务中学习组织/器官差异可以产生更好的代表性特征。
3DSeg-8 的原始体数据来自不同的模态(MRI 和 CT)、不同的扫描区域(例如大脑、心脏、前列腺、脾脏等)以及多中心,从而产生了大量的数据特征,例如 3D 中的 3 个空间分辨率和像素强度范围。为了减轻可能阻碍网络训练过程的数据变化问题,我们使用预处理模块处理数据,该模块完成空间和强度分布的归一化。
空间归一化。由于不同的扫描仪或不同的采集协议,医疗体积很常见,体素间距不均匀。这种间距是指图像中两个像素之间的物理距离,在不同域之间几乎完全不同。更重要的是,这样的间距信息无法从 CNN 中学习到。空间归一化(即,将体积重新采样到固定分辨率)通常用于减少体素间距变化的影响。为了避免过度插值,我们根据各自的域将每个体积内插到中值间距,以保持每个域中目标的空间特征。
由于每个域的目标器官在整个图像中所占的比例不同,经过空间归一化后,随机裁剪了从目标边界框的两倍到整个体积的大小变化的区域,以确保训练数据中完全包含目标。
强度归一化。在不同的成像方式下,医学图像的像素值范围差异很大。为了消除像素值异常值的副作用,特别是对于CT模态 (例如,CT中的金属),我们对每个图像中的所有像素值进行排序,并将强度截断到0.5到99.5个百分位数的范围。
2.Med3D 网络
目前流行的预训练网络,如 ResNet和预激活 ResNet,已被证明可以有效地转移到其他视觉任务,因为它们提供了一个通用的编码器,涵盖了低和高在其他领域也有帮助的级别功能。因此,在 Med3D 网络的设计中,我们利用成熟网络的现有特征提取架构,专注于探索用我们独特的 3DSeg-8 数据训练 Med3D 的最佳方式。由于目标是通过训练分割网络来学习通用特征表示,因此采用了通用的编码-解码分割架构,其中编码器可以是任何基本结构。在这项工作中,采用 ResNet 模型族作为编码器的基本结构,并进行少量修改以允许网络使用 3D 医学数据进行训练。 3DSeg-8 数据和自然图像分割任务中的数据之间的一大区别是缺少多器官分割,其中每个数据集中只有特定的器官/感兴趣的问题被注释为前景类,而其他的则留作背景。例如,肝脏/肿瘤数据集中只有肝脏分割掩码可用,并且附近的胰腺被标记为背景。同样,只有胰腺被标记为胰腺/肿瘤数据集中的前景。这种不完整的注释信息可能会混淆网络,并且训练过程不会收敛。由于在大规模 3D 医学数据中详细注释完整的器官图谱在技术上是不可能的,我们提出了一种多分支解码器来解决不完整的注释问题。如上面的图 2 所示,将编码器与八个特定的解码器分支连接,其中每个分支对应于 3DSeg-8 的一个特定数据集。在训练阶段,每个解码器分支只处理从相应数据集中提取的特征图,其余分支不参与其优化过程。此外,每个解码器分支共享使用一个卷积层来组合来自编码器的特征的相同体系结构。为了计算网络输出和真实值之间的差异,直接将特征图从解码器上采样到原始图像大小。这种简单的解码器设计使网络可以专注于训练通用编码器。在测试阶段,解码器部分被移除,剩余的编码器可以转移到其他任务。
3.迁移学习
作者的目标是建立一个通用的 3D 骨干网络,该网络可以转移到其他医疗任务中,以获得比从头开始训练更好的性能。 为了验证已建立的 Med3D 网络的有效性和多功能性,我们在分割和分类领域进行了三个不同且彻底的实验。
肺分割。 Med3D 只看到局部感受野中的不同组织,并且从未受到肺注释的监督。 为了验证 Med3D 生成的特征在大感受野(整个人体)的肺分割任务中是否具有通用性,将 Med3D 中的编码器部分作为特征提取部分,然后在全身分割肺,然后是三组 的 3D 解码器层。 第一组解码器层由内核大小为(3, 3, 3)、通道数为256(用于将特征图放大两倍)的转置卷积层和(3 , 3, 3) 内核大小和 128 个通道。 其余两组解码器层与第一组相似,只是网络的每层通道数逐渐增加一倍。 最后,使用 (1, 1, 1) 内核中的卷积层生成最终输出,通道数对应类别数。
肺结节分类。 为了进一步验证 Med3D 在非分割任务上的通用性,如图 2 所示,我们将 Med3D 的编码器部分转移到结节分类器作为特征提取器,平均池化操作和全连接层具有 (1, 1, 1) 添加内核大小以对结果进行分类。 与人体器官(例如脾脏和胰腺)不同,肺结节是一种尺寸小得多的微观结构。 此外,肺结节具有边界模糊、形态变形大、纹理信息丰富等特点,与正常人体器官相比存在较大差异。 将 Med3D 模型转移到肺结节任务可以帮助检查 Med3D 产生的特征是否也适用于微观组织。
LiTS 挑战赛。 为了评估 Med3D 预训练模型在实际应用中的性能,我们将 Med3D 模型应用于 LiTS 比赛中的肝脏分割。 肝脏是原发性肿瘤发展的常见部位。 与肝脏肿瘤和邻近组织的低强度对比是准确分割肝脏的一大障碍。 肝脏是与左心房相同的单一连接区域。 受 2018 年 3D 心房分割比赛第一名获得者的方法的启发 ,采用两阶段分割网络来分割肝脏。

如图3所示,首先,在整个图像中粗略地分割肝脏以获得目标的感兴趣区域(ROI)。 在这个阶段,将从 Med3D 预训练的主干转移作为编码器部分,然后是具有 (1, 1, 1) 内核的卷积层,通道数为两个(肝脏与背景)。 调整图像大小后,我们将图像输入到粗分割网络中,以 32 次下采样提取特征。 然后,通过双线性插值方法将特征图上采样到原始图像大小。
其次,根据第一阶段的结果对肝脏目标区域进行裁剪,并再次对目标进行子分割,从而获得最终的肝脏分割结果。这个阶段主要关注仔细的肝脏轮廓分割。为了在特征图中获得更密集的尺度信息并获得更大的感受野,从 Med3D 预训练的主干嵌入到最先进的 DenseASPP 分割网络中。该网络以密集的方式连接一组多孔卷积层,从而生成多尺度特征,这些特征不仅覆盖更大的尺度范围,而且在不显着增加模型大小的情况下密集覆盖该尺度范围。并且,我们将所有 2D 内核替换为相应的 3D 版本。由于第一步的结果在真实值和肝脏目标预测之间不可避免地存在偏差,为了提高精细分割模型的鲁棒性,随机扩展肝脏目标区域,然后使用包括旋转和平移在内的两种增强方法对其进行处理。
三、实验
作者进行了几个实验来探索所提出的 Med3D 模型的性能。首先,展示了实验的详细设置以及不同数据集设置的一些比较结果。接下来,转移预训练的 Med3D 编码器来初始化其他医疗任务的网络,并将结果与从头训练网络和预训练网络与动力学数据进行比较。最后,将预训练的 Med3D 编码器与 DenseASPP 网络连接起来,并使用单个模型展示了肝脏分割任务的最新性能。
1.实验细节
3DSeg-8 数据集。 3DSeg-8 是来自八个公共医疗数据集的聚合数据集。它通过 CT 或 MR 扫描覆盖不同的器官/组织,如表 1 所示。从每个成员数据集中,我们随机选择 90% 的数据作为训练集,其余 10% 作为测试集。为了提高网络的鲁棒性,我们使用了三种数据增强技术,包括平移、旋转和缩放,设置如下:在目标边界框 10% 边距内的任意方向平移数据;在 [-5, 5] 内旋转数据;并在原始大小的 [0.8, 1.2] 倍范围内缩放数据。

网络架构。采用 ResNet 系列(具有 10、18、34、50、101、152 和 200 的层)和预激活 ResNet-200 架构作为 Med3D 网络的主干。为了使网络能够使用 3D 医疗数据进行训练,对骨干网络进行了如下修改:1)由于单通道体积输入,将第一个卷积层的通道数从 3 更改为 1; 2)用3D版本替换所有2D卷积核; 3)将块 3 和 4 中的卷积核的步幅设置为 1,以避免对特征图进行下采样,并且使用具有 r = 2 率的扩张卷积层用于以下层相同的目的; 4)用一个 8 分支解码器替换全连接层,其中每个分支由一个 1x1x1 卷积核和一个相应的上采样层组成,将网络输出扩展到原始维度。
训练。由于每个成员数据集都有不同数量的训练数据,从最大的数据集中获取所有训练数据并随机扩充其余数据以生成均匀分布的平衡训练数据集。我们使用交叉熵损失和标准 SGD 方法优化网络参数,其中学习率设置为 0.1,动量设置为 0.9,权重衰减设置为 0.001。
训练数据量级的影响。视觉系统的性能随着训练数据的数量成对数增加。为了评估训练数据大小在医学成像领域的影响,我们分别用 10%、20%、40%、80% 和 100% 的训练数据训练 Med3D 网络(ResNet-152 主干),并使用相同的测试设置来比较性能。从图 4 可以看出,当训练数据利用率为 100% 时,所有任务的 Dice 得分最高。当训练数据量只有 10% 或 20% 时,由于过拟合问题,模型性能急剧下降。当训练数据的数量增加时,所有的实验都呈现出相同的改善趋势,这表明 Med3D 的性能和训练数据的大小也存在相关性。

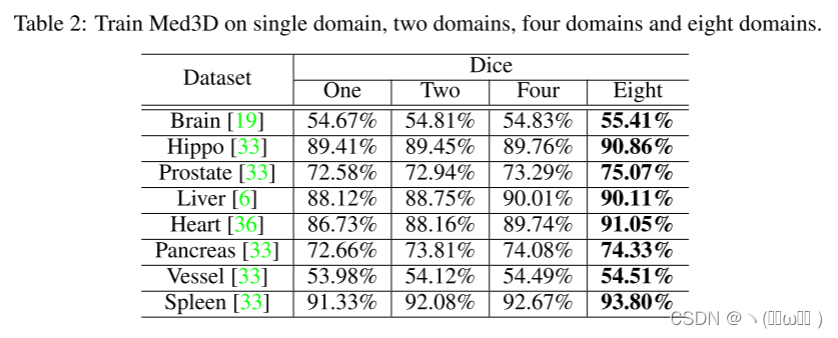
训练集多样性的影响。设置了一组比较实验,以使用来自不同数量数据集的数据来研究 Med3D 的性能。例如,使用单独的成员数据集训练 Med3D,并将结果与来自两个、四个和八个成员数据集的结果进行比较。使用不同的颜色来标记具有两个和四个成员数据集的实验的组设置。如表 2 所示,当所有成员数据集都用于训练时,Med3D 达到了最高性能。这是因为数据种类可以提供补充信息并提高整体性能。
2.迁移学习实验
在之前的实验中,研究了 3DSeg-8 数据集上的 Med3D 性能(ResNet-152 主干)。结果表明,当使用由八个域组成的完整 3D 医学数据集进行训练时,Med3D 实现了最佳分割性能。在本节中,将探索预训练的 Med3D 网络在未知数据集上的性能,以验证学习特征的可迁移能力。
3DSeg-8 数据集由来自八个不同数据集的器官/组织组成,这些数据集具有适度的尺度变化。为了清楚地揭示预训练网络被概括为规模和任务不变,对来自肺区域的数据进行了两个实验,肺分割内脏数据集和肺结节恶性肿瘤数据集 LIDC。还将使用预训练 Med3D 的结果与使用预训练动力学模型 (Kin) 的结果以及从头训练 (TFS) 的结果进行比较。
肺分割任务。选择 Visceral 数据集,因为它包含丰富的肺分割注释,用于 4 种不同模式的数据。全书共80册。选择 72 卷用于训练,8 卷用于测试。训练和测试数据都包含 4 种模态。在训练期间,对所有比较候选者使用相同的分割架构(ResNet 系列)。主要区别在于如何初始化分割网络,使用预训练的 Med3D、预训练的 Kin 或 TFS。在使用预训练模型进行训练时,使用 Adam从 0.001 的学习率开始优化模型参数,而 TFS 的学习率设置为 0.01。如表 3 所示,所有结果都遵循相同的模式,即 Med3D 网络的性能比 Kin 网络好得多,而 TFS 网络是最差的。这证实了最初的假设,即时间视频数据捕获的 3D 信息与医疗体积之间存在差距。为了在 3D 医学数据上获得最佳性能,最好从捕获有关人体生理结构信息的特征开始。图 5 中的子图展示了不同初始化的分割任务的训练曲线。可以看出,在一定的数据量和足够的训练迭代下,三个网络都收敛到了稳定的损失。然而,Med3D 网络可以将损失推到比其他两个更低的水平,并显示出更快的收敛速度。


肺结节分类任务。LIDC数据集收集了来自1,010名患者的胸部ct扫描,每个ct扫描的结节由四名放射科医生注释。根据lidc-idri的规则,结节的恶性程度分为五个级别,从良性到恶性。我们将 “1”,“2”,“3” 合并为良性,将 “4”,“5” 合并为恶性,以减少主观不确定性。此任务的目标是比较使用不同初始化方法的网络性能。我们通过将完整连接的层附加到编码器来将骨干网更改为分类体系结构。使用1,050个结节进行训练,99个结节进行测试。优化参数与前面的任务一致。从表3中可以看出,基于预先训练的Med3D网络的结果大大超过了基于Kin和TSF的结果。这证明了Med3D学习功能的有效性,这也有助于分类任务。而且,当网络深度逐渐增加时,Med3D的性能也随之提高。相反,当网络复杂性较高时,Kin和TFS网络的性能均下降。结合图6所示的证据,在经过足够长的训练时期后,不同网络的训练损失降低到相似的水平,可以得出结论,从Med3D网络中提取的特征对于具有较小数据集的分类任务是更好的概括的数据,而其他两种方法则显示出过拟合问题。与图5类似,图6还提供了反映 fl 网络收敛速度和难度等级的训练损失曲线。从图中可以看出,从头开始训练的模型比使用Med3D预训练权重初始化的模型具有更高的 fl 功能和收敛速度慢得多。由于此任务是一个简单的二进制分类任务,因此可以想象从头开始训练的网络可能难以收敛到复杂的分类任务。

3.LiTS 挑战赛
已经证明,与自然场景视频和从头开始训练的网络相比,预训练的Med3D网络在分割和分类任务中对没见过的数据具有更好的有效性和泛化。这让我们想知道Med3D是否可以在具有挑战性的任务 (例如LiTS挑战) 上进一步提高网络性能,其中从头开始训练的3D网络通常比使用自然图像上的预训练模型的2D或2.5D方法具有更差的性能。LiTS挑战数据集具有201次增强的腹部ct扫描,其进一步分为具有131次扫描的训练集和具有70次扫描的测试集。仅将训练数据注释提供给公众,而测试数据注释则与主机保密。任务是分割肝脏和肝脏肿瘤。由于两个原因,它被称为具有挑战性的任务。首先,训练数据数量少。其次,使用不同的扫描仪和协议从不同的临床部位收集数据,导致数据质量,外观和间距发生很大变化。使用ResNet-152主干建立了相同的分段网络,并使用预先训练的Med3D对其进行了初始化。在训练过程中,使用训练数据的平均间距值对所有数据进行标准化,因为某些卷没有提供间距信息。还用从-200到250 Hounsfield单位的窗口宽度来归一化强度。所有的训练超参数都与之前的分割实验相同。如表4所示,Med3D网络无需任何后处理即可获得94.6% 的得分,这与使用集成技术的最新网络非常接近。还将Dice结果与纯3D网络 (例如V-net,Kin和TFS) 进行了比较,并表明Med3D的性能优于具有较大利润的方法。

总结
作者构建了一个大规模的 3D 医学数据集 3DSeg-8,并提出了一个新的框架来用这些数据训练 Med3D 网络。
从 Med3D 网络中提取的特征被证明是有效的和泛化的,并且可以用作具有小训练数据集的其他任务的预训练特征。
与使用自然视频训练或从头开始训练的网络相比,Med3D 网络取得了更好的结果。








 本文介绍了Med3D网络,通过构建3DSeg-8数据集,使用异构3D网络进行多域训练,以提取通用的3D医学特征。实验表明,Med3D在肺分割、结节分类和LiTS挑战中表现出色,证实了其在小数据集任务中的迁移学习能力。
本文介绍了Med3D网络,通过构建3DSeg-8数据集,使用异构3D网络进行多域训练,以提取通用的3D医学特征。实验表明,Med3D在肺分割、结节分类和LiTS挑战中表现出色,证实了其在小数据集任务中的迁移学习能力。
















 1350
1350

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








