文章题目:
Benchmarking scRNA-seq imputation tools with respect to network inference highlights
中文题目:
关于网络推理的scRNA序列插补工具基准突出了高稀疏性水平下的性能缺陷
说明
这是一篇关于插补后scRNA-seq数据是否带来假阳性的讨论性质文章,对目前的几种基于不同原理的插补scRNA-seq算法进行了仿真数据集的测试,同时作者提出插补后数据集本身的生物edge被破坏,插补带来的评价指标提升是虚假edge这个设定是很吸引人的。
这篇文章目前并没有发表,只有在预印阶段,后续发表的话我会更新本帖。
文章链接
正文
任何一个文章实际上都在阐明的立场 内核是 Bulk-RNAseq和Sc-RNAseq二者的区别,为什么要做到SC(single cell)级别 而不直接使用Bulk呢。
总结一句话就是:
目前人们使用转录组学数据推断基因调控网络,用以获得重要的生物学见解和想法。Bulk-rna-seq数据中可能包括不同的细胞(子)类型、处于不同细胞周期阶段的细胞或获得某些遗传突变的细胞亚群。
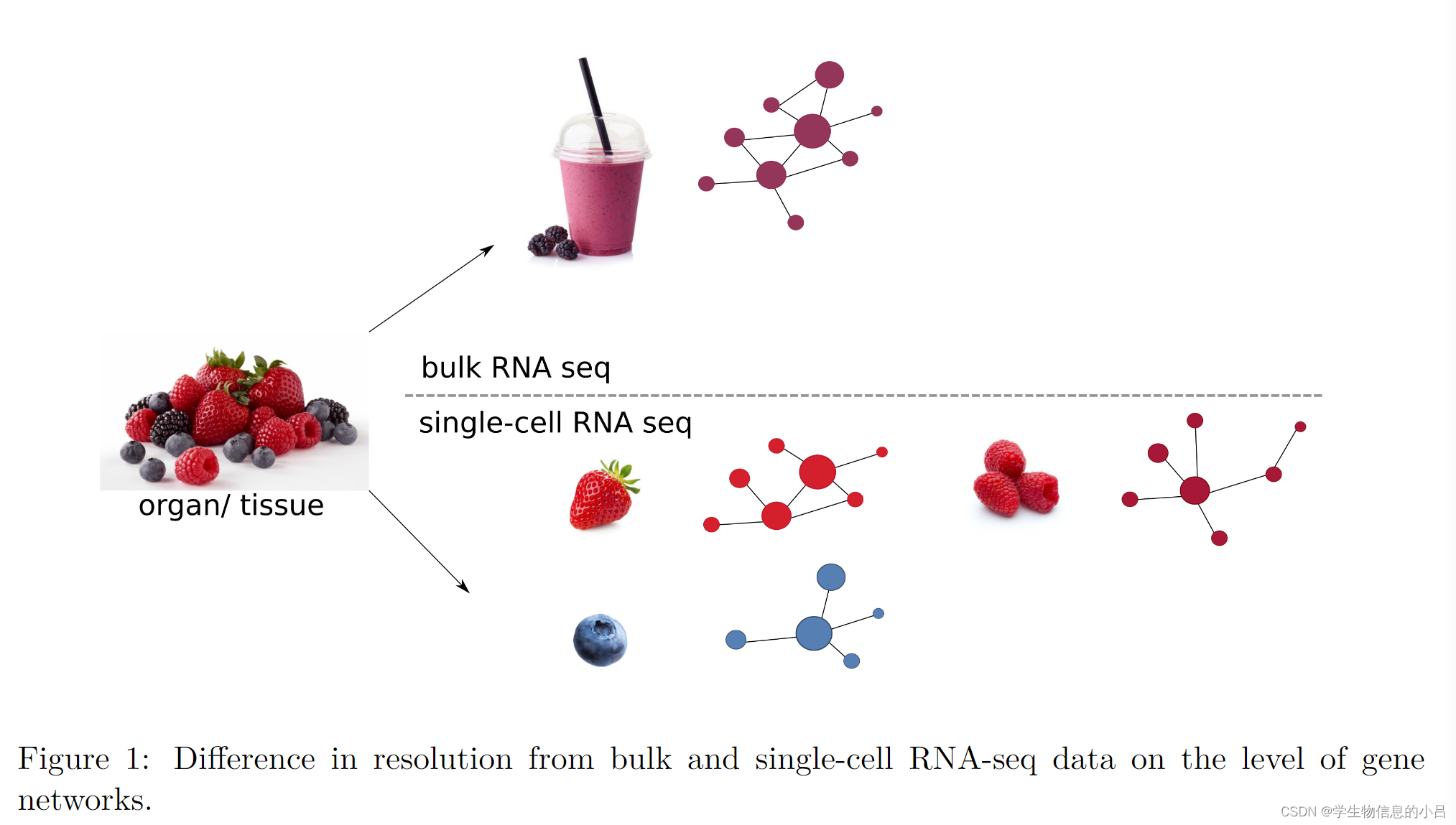
而scRNA-seq分别提取和分析每个细胞的转录组。单细胞转录组数据中的细胞间异质性有望大大提高对细胞过程的理解,从而揭示特定于细胞类型或细胞簇的基因调控网络,并且迄今为止仍处于埋藏状态。例如,scRNA序列有助于识别以前被掩盖的细胞类型,揭示细胞群体内的细胞动力学,并揭示发育轨迹。
这就好像如图1所示的 一个水果组织 有蓝莓 草莓 树莓等多种水果(因为技术手段我们只能直接提取一堆水果而不是一个个拿取水果)
但是如果做Bulk-rna-seq的话 就好像做了一杯混合莓果汁一样 但是我们真正想看到的是各类水果自身的特定转录组,来做特定水果的后续分析。
因此 使用scRNA-seq数据最主要是实现两点:
- 细胞类型的鉴定
- 推断基因相关网络的可能性
scRNA-seq数据本身因为其稀疏性导致了大量dropout(根据不同测序平台以及样本质量)出现。
基因表达谱中的“0”由两种来源:
- 生物“0” 就是真实情况下的记忆布表达现象。
- 技术“0” 来源可能是来自于上游的测序过程中mRNA未捕获成功,或者来源于不同捕获后的反转录或者PCR等不成功,基因表达谱出现特定基因部分表达部分不表达的情况
因此,scRNA-seq数据的这些特殊特征可能会干扰前面所述的目标,特别是在细胞簇的基因调控网络的推断方面。
除了总体聚类性能外,还研究了数据插补对特定细胞簇表达谱的影响。由于每种细胞类型都有一组特定的标记基因,数据插补不应改变或破坏这些表达谱,因为它们在推断细胞类型特定的网络结构中起着核心作用,但最近的研究表明,数据插拔后,它们的可繁殖性(reproduce)降低了。(这个问题详见我另一篇帖子:插补带来的假阳性引入问题)
scRNA-序列数据集内部的网络本身不容被破坏,因此需要一个ground Truth,即一个Gold数据集来违网络评估性能的参考依据。该数据集内只包含生物“0”。
如何构造Gold data?
术语Gold指的是 不包含基于测序错误的0点的单细胞数据。
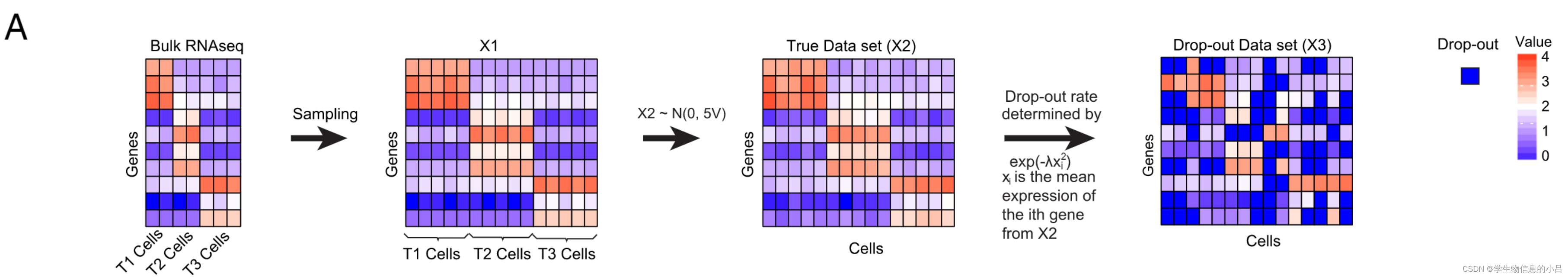
Gold构造使用Bulk-RNA-seq数据集,从Bulk-RNA-seq数据集中做采样,加入噪声的原理。
首先说明,本文的数据集构造方法来源于2019年发布的文章:

具体模拟策略:
Gold数据集
首先由T1细胞、T2细胞和T3细胞三种类型的细胞组成的bulk RNA-seq数据矩阵开始,数据矩阵X1是通过对不同类型细胞的原始数据分别重新采样得到的。然后,数据矩阵中的每个元素 (xij) 都受到正态分布 N(0, 5V) 的扰动(可以理解为加入噪声)(V 是批量 RNA-seq 数据中重复基因的标准差向量),真实数据集 X2生成(有噪声才叫真实数据集)。最后,使用指数函数在 X2 中引入 dropout 事件,从而产生 dropout 数据集 X3。 产生Gold数据集之后,使用WGCNA校验黄金数据集,以确保生成的Gold保留了大量RNA序列数据集的自然相关结构。
reference数据集
根据Gold数据集创建 dropout程度不等的数据集:
其中包含 55% 到 99% 的零,这类似于当前 scRNA-seq 方法的零比例。
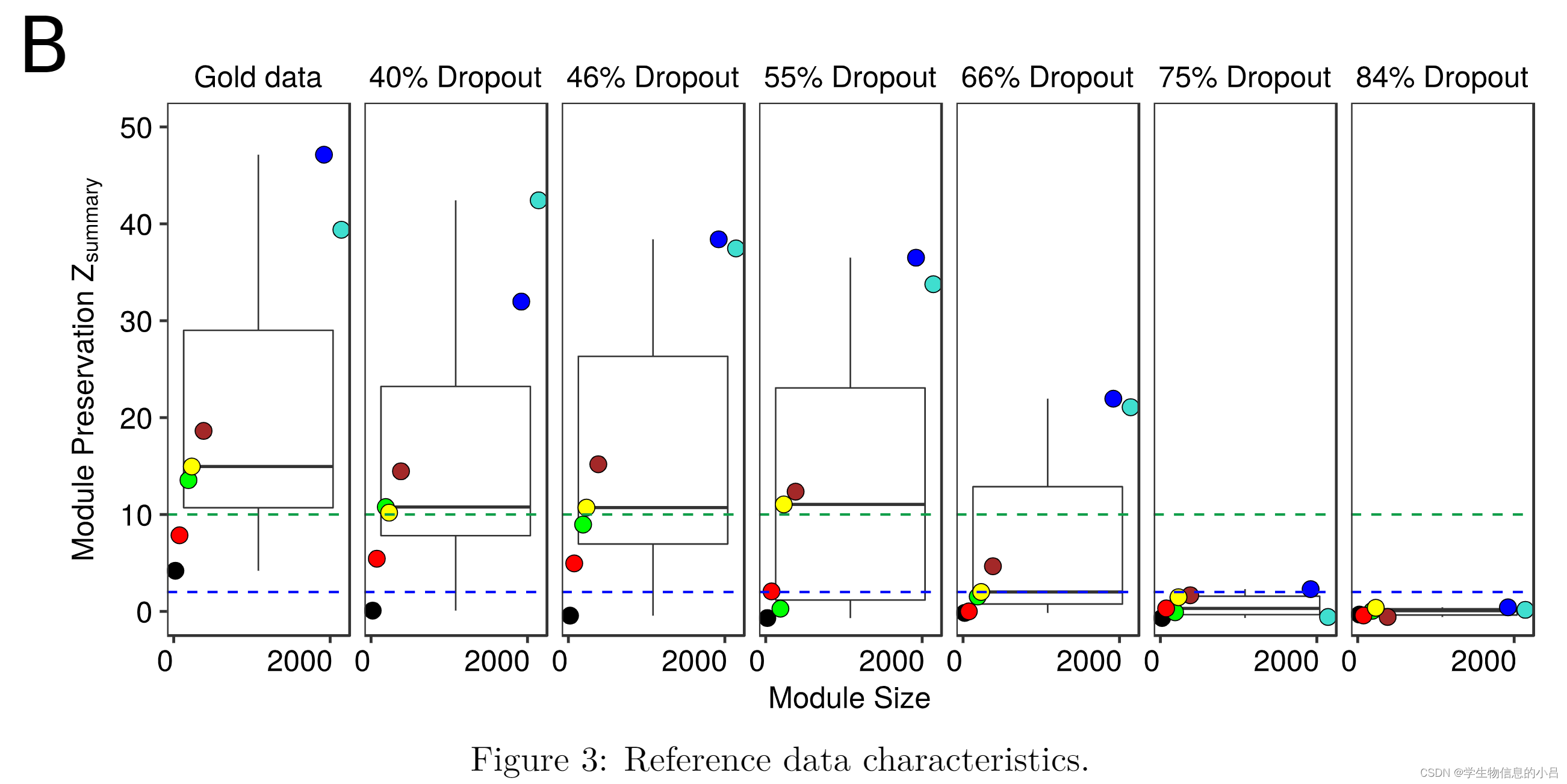
上图所示的箱型图显示了使用WGCNA对Gold data以及不同数据集data的统计结果说明,使用Z-summary来度量不同模块的强度。
该度量表明来自参考数据集的一组相关基因(模块)在测试数据集中的保存程度。在这里,所有人为丢弃的数据集都与Gold-data进行了比较。由于 Zsummary 依赖于模块大小,我们将Gold data与自身进行比较作为参考。
Zsummary 值低于 2 的模块被认为根本不保留,其中大于 10 的值表示强保留。两个阈值之间的模块被认为是适度保留的。
上图中不同的dropout率对应的Z-summary图片中,在高dropout率下,模块保留几乎没有,但是在中低水平的dropout率下,模块保存尚可。
因此,这样构造gold-data和reference-data的方法
- 在表达值分布方面类似于真实的生物数据集
- 保留其原始批量 RNA-seq 祖先的自然相关结构
- 被证明具有随着更高水平的辍学减少网络保存(与真实情况符合)。
imputation对基因网络的影响
使用插补方法
| 方法 | 原理 |
|---|---|
| DrImpute | 基于平滑 |
| SAVER | 基于模型 |
| ENHANCE | 基于低秩矩阵 |
| DCA | 使用神经网络结构 |
| scNPF | 从数据集中提取基因相关网络信息的同时进行插补 |
| DISC | 属于神经网络类,但以半监督方式执行插补。 |
所有工具都在单细胞社区中得到高度使用和接受
模块的保留
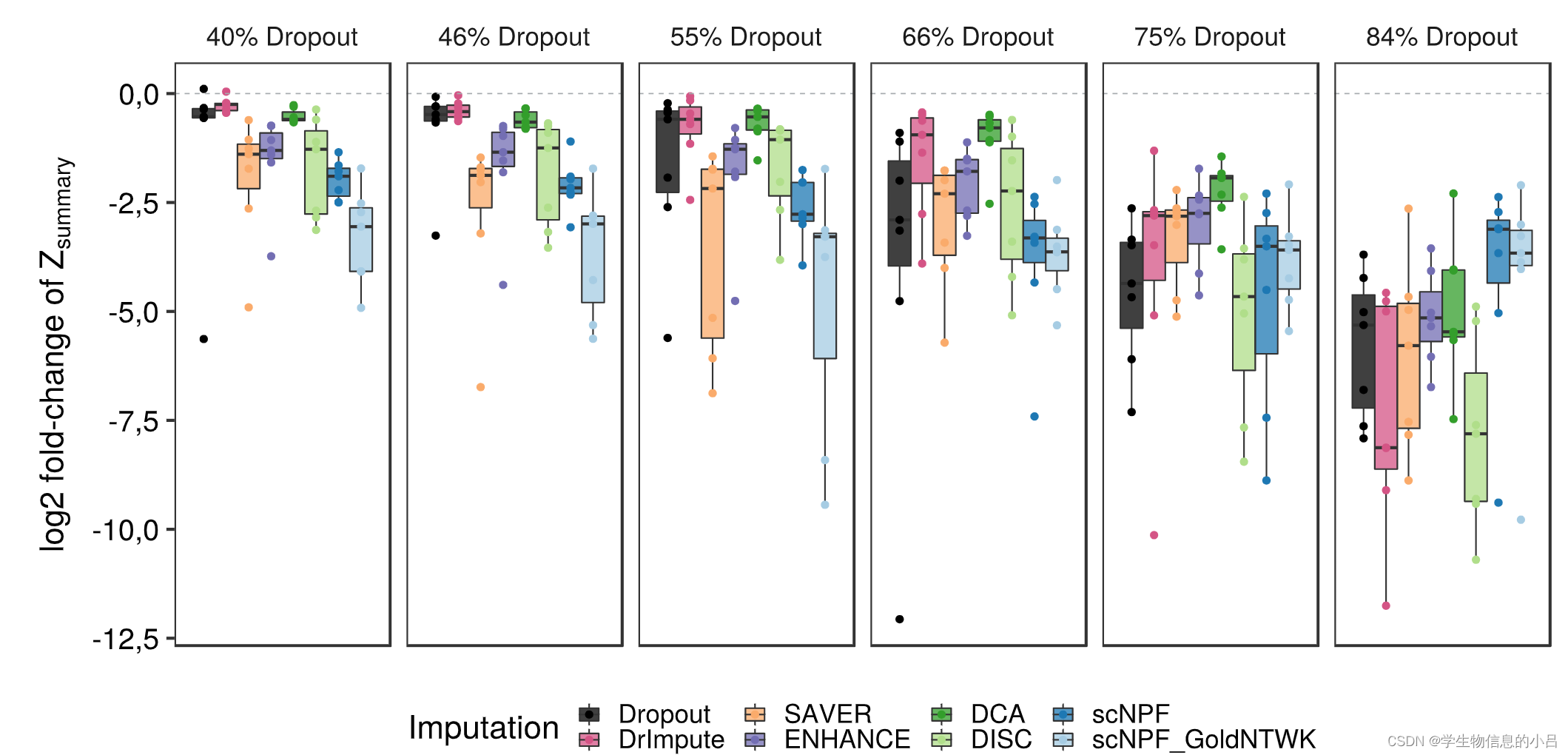
上图:模块式 Zsummary log-2 倍数变化 (log2-FC),它反映了插补前后模块保存与黄金数据集的差异
黑色模块代表dropout后没有做插补的数据与gold作比较,得到的Z-summary值。
其他颜色代表各种插补后的数据集与gold作比较,得到的Z-summary值。
结论
| dropout比率 | 结论 |
|---|---|
| 低水平的 dropout | 网络推理在没有插补的情况下仍然可以正常工作,只有 DrImpute 和 DCA 产生了与未插补数据相当的 log2-FC。所有其他工具的表现都要差得多。 |
| 在中间级别的 dropout (55-66%) | 没有插补的网络推理受到的影响越来越大,但仍然可行,DrImpute 和 DCA 极大地改进了 log2-FC。 SAVER、ENHANCE 和 DISC 导致与未估算数据相似的 log2-FC 分布,而 scNPF 的两种变体甚至表现更差。 |
| 高dropout水平 | 没有一个插补工具能够使用足够的 log2-FC 进行网络推理。 |
总之,从模块保存的角度来看,使用 DrImpute 和 DCA 的插补有助于对具有低到中等丢失水平的数据集进行网络推理,而其他几种工具严重损害了这些dropout的推理。然而,对于高dropout比率的数据集,没有任何插补工具显示出令人信服和有希望的结果。
edge的恢复
除了推断插补对网络推断的影响,也要分析插补方法恢复黄金数据基因相互作用的能力。
首先计算Gold 数据集和各个 dropout后的reference数据集的TOM重叠矩阵
TOM矩阵介绍
目的:使用TOM重叠矩阵去量化不同gene之间的真实相关性
人为设定一个阈值,该阈值作为判定是否是真实生物相关性的标准:
阈值是根据拓扑重叠矩阵 (TOM) 的平均值和标准差计算的,其中 1 * 标准差 + 平均值 为阈值,高于此阈值的权重才被视为现有的生物edge (需要保留)
而后在插补后数据判定恢复真实生物edge的结果。
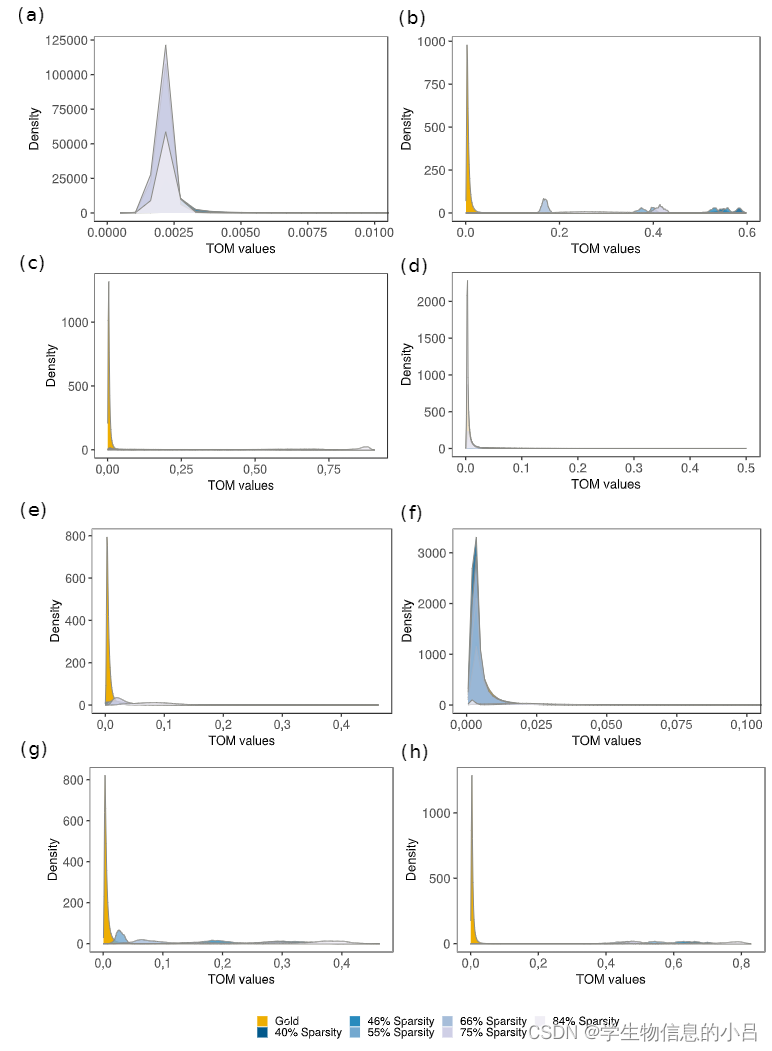
合成数据集插补前后 TOM 值的密度
通过黄金数据β值将插补前后的表达数据转换为TOM值后,在边缘恢复分析期间检测并比较正边缘。颜色对应于数据的原始信息内容(最浅的蓝色 - 信息较少,稀疏度最高)。 (a) dropout后没有插补数据,(b) DrImpute,© SAVER,(d) ENHANCE,(e) DCA,(f) DISC,(g) scNPF,(h) scNPF Gold。与黄金数据相比,某些工具,例如 DrImpute (b) 和两种 scNPF 方法 (g+h) 产生高 TOM 值,DCA (e) 和 ENHANCE (d) TOM 密度接近gold-data。
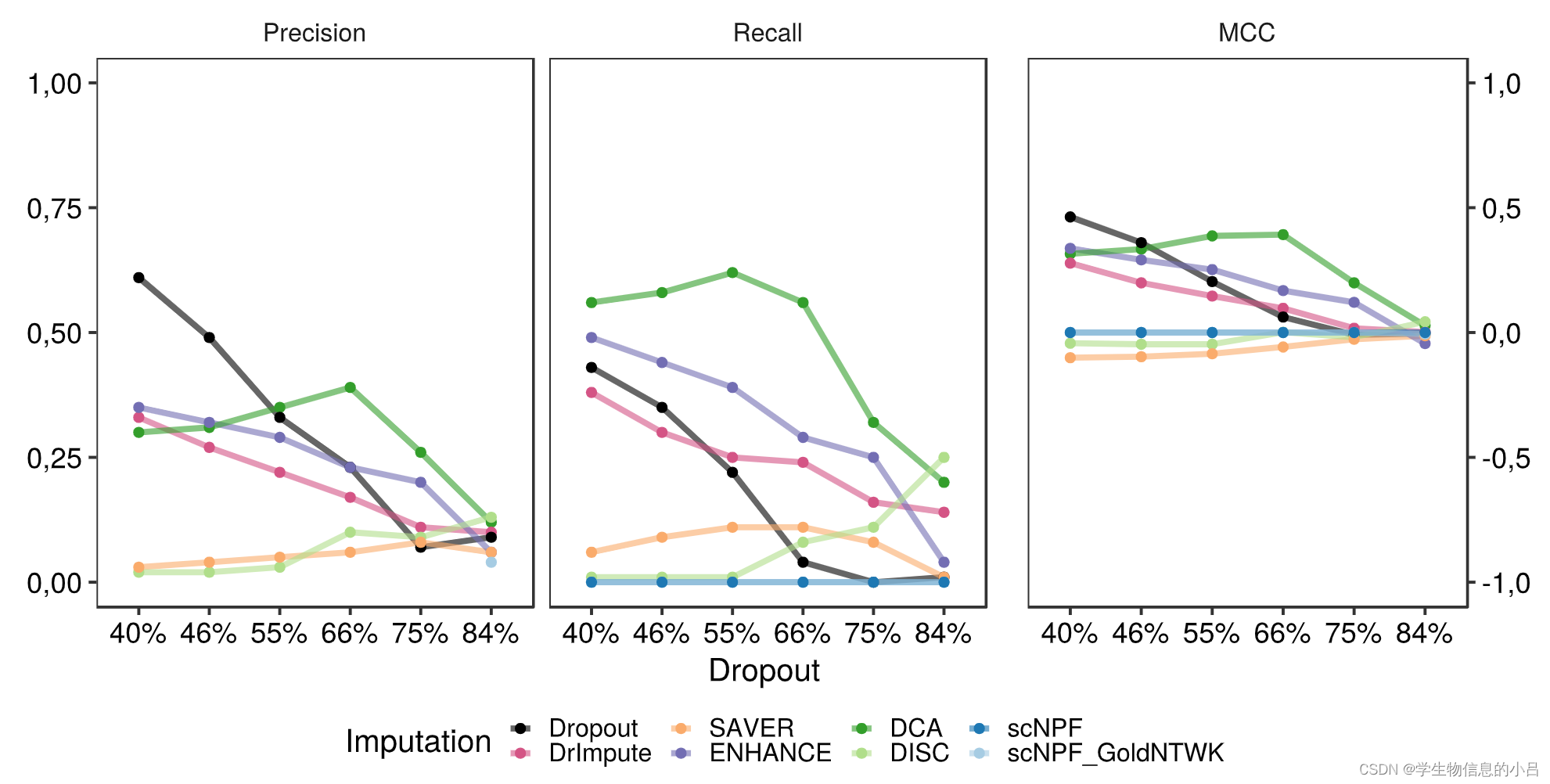
结论
具有低dropout水平的未插补数据,获得了总体良好的边缘恢复值。 DCA 显示在受中等dropout水平影响的数据集上表现适中
细胞间相关性
使用数据
人类视网膜数据集
基因表达谱中0占比为85%
评价指标
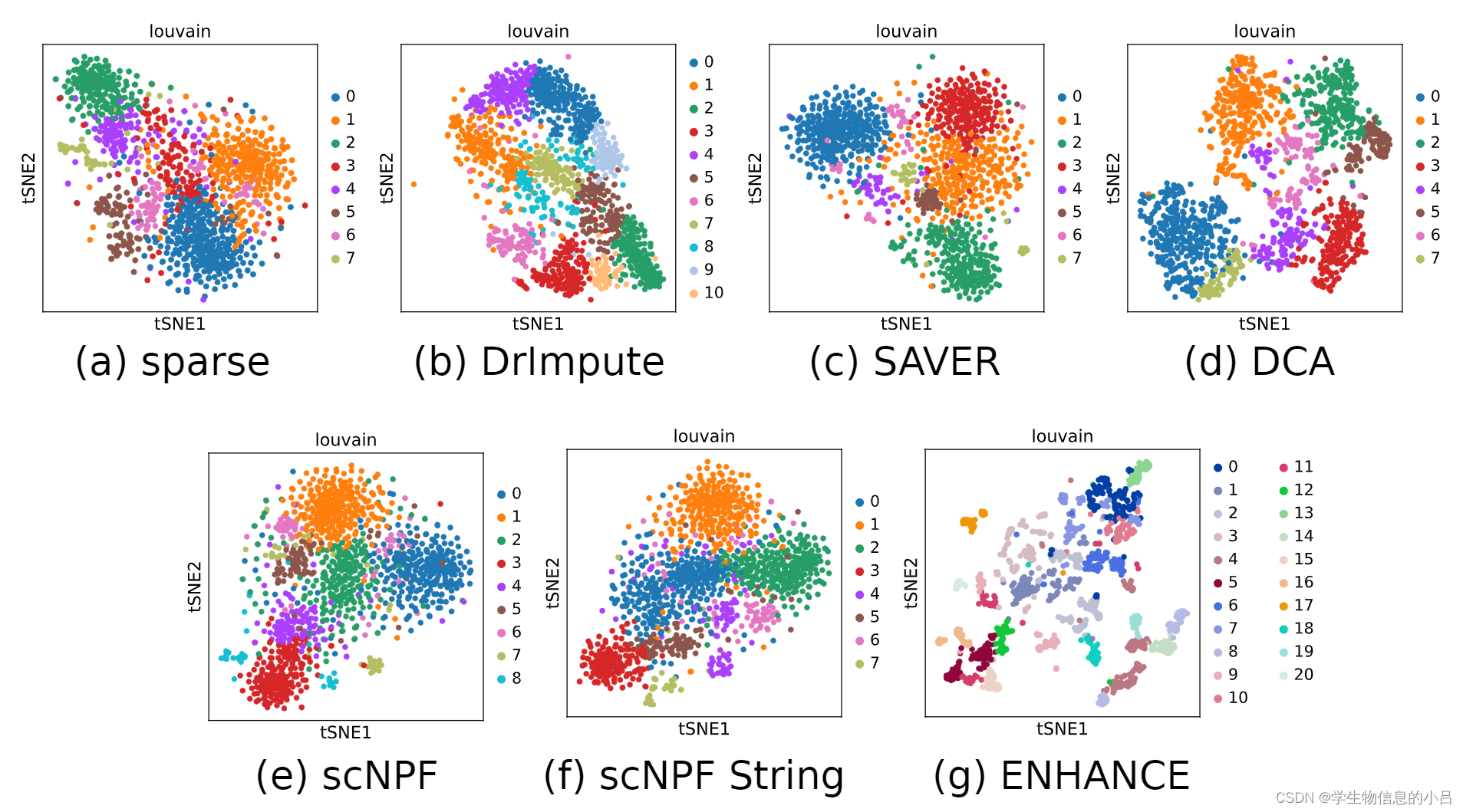
聚类cluster数目和轮廓系数:
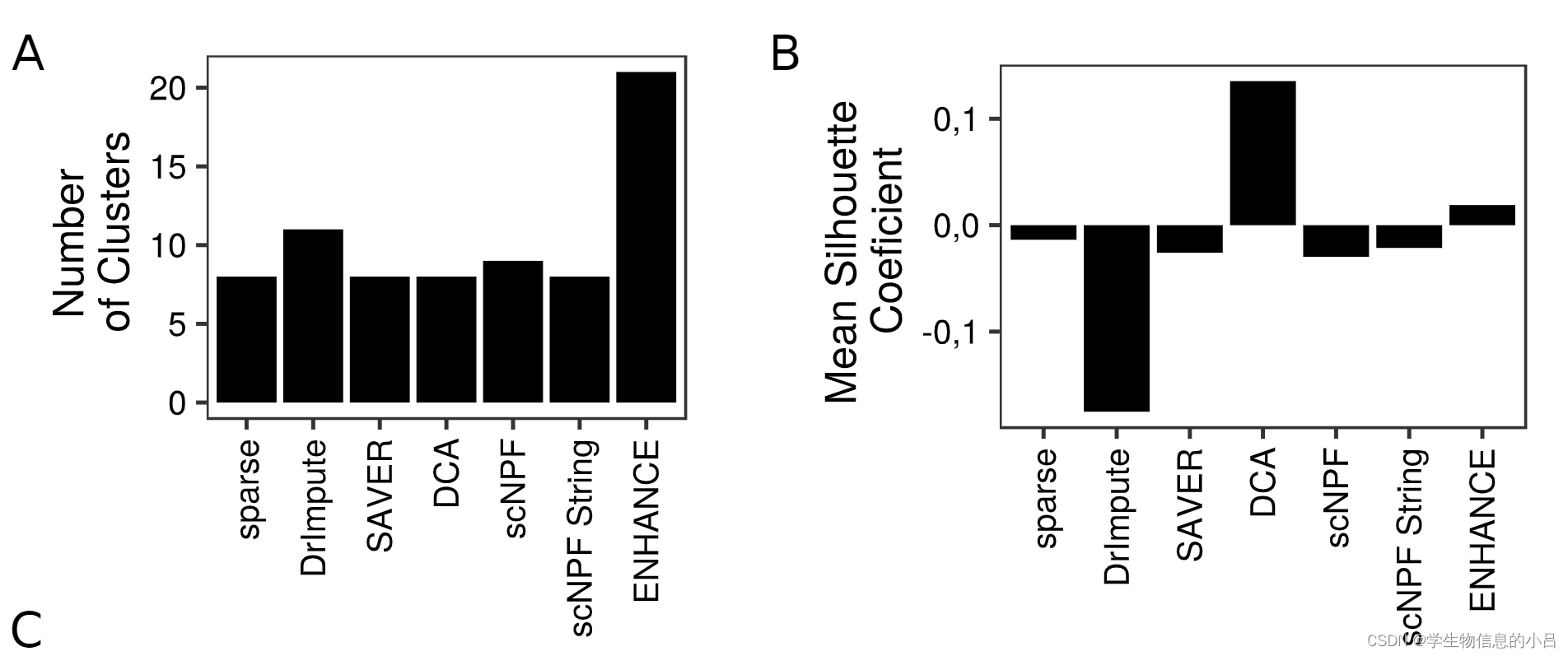
自建三种评价聚类的指标:
- 纯簇表示所有单元格具有完全相同注释的簇的百分比
- 混合注释总结具有至少两个不同注释的簇
- 不可分配包含不可能唯一注释的簇
结论
来自批量测序数据的相关结构可用于研究 dropout 对单细胞转录组数据中基因网络的影响
将下采样方法应用于批量 RNA-seq 数据集以生成非稀疏单细胞类Gold-data的思路是正确的,具有合理性。
我们能够证明,这种合成数据具有合适的基因-基因相关特性,可以推断近似无标度拓扑的网络。由于该数据集根据定义仅包含真零,因此我们能够随后生成具有定义丢失程度的数据集。与gold-data相比,这些reference数据集能够彻底调查数据丢失对揭示网络信息的影响,这些信息被测量为模块保存。
从低 dropout 率的 scRNA-seq 数据中检索可靠且有意义的生物基因网络仍然是可维护的(高达 55% 的 dropout)。 而dropout 水平超过 75% 甚至 84% 的数据集,类似于典型的最新 10X Genomics 数据输出,非常不适合网络推理分析。克服这种情况的一种选择可能是在网络推理之前操纵数据,例如通过数据插补。这可能有助于潜在地解除上述限制,以最终利用源数据的更高分辨率。
当前的插补方法不能改善来自高 dropout 数据集的网络推理
插补的效果高度依赖于所应用的方法,甚至更依赖于源数据集的 dropout 水平
大多数算法会改变完整的基因相关结构,而不是恢复以前隐藏的信息
| drop率 | 建议 |
|---|---|
| 对于低水平的dropout数据集 | 几乎不能从插补中受益,所以尽量使用原始数据,不使用插补数据。 |
| 对于中间级别的 dropout(高达 66%) | 与稀疏数据相比,估算数据集显示出更好的模块保存。 |
| 超过 75% 的丢失率 | 没有任何插补工具能够大致恢复真实的基因相关结构。 |
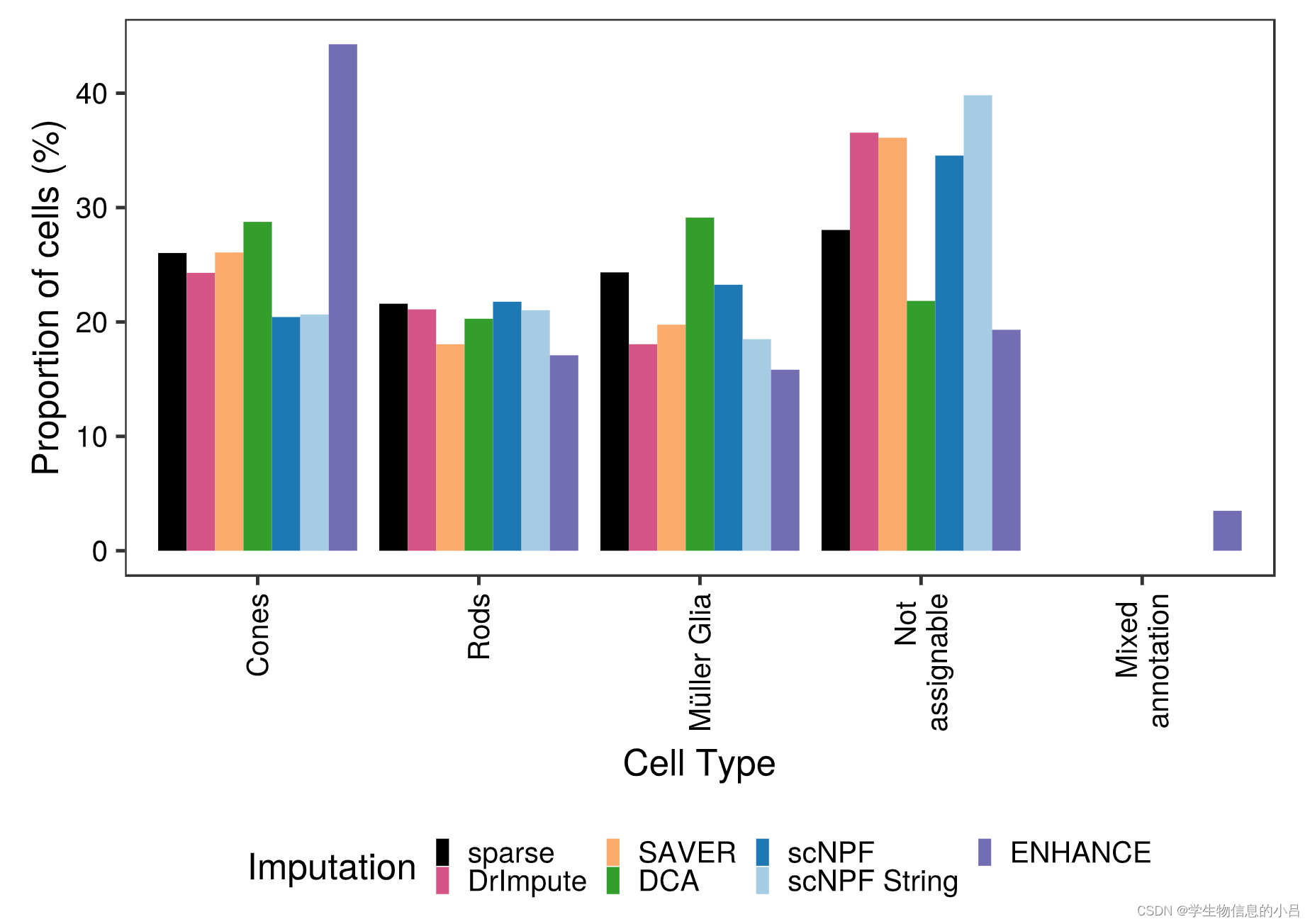
标记基因表达不受插补的影响
已知:插补方法倾向于将假阳性信号引入(高丢失)基因-基因相关数据
所以:插补方法对标记基因的整体表达谱没有负面影响是非常重要的。
上文中,纯视网膜簇的注释使用 DCA 或 ENHANCE 仅略有改善。与未估算的数据相比,所有剩余的工具导致视网膜特异性细胞类型减少,这是因为DCA 和 ENHANCE 允许注释更多的视网膜细胞,因此可以改善
一般来说,我们可以证明应用的插补工具保持标记基因的可用性,从而产生可比较数量的注释细胞簇
总结
根据在合成数据集中保留网络特征的发现,建议使用未插补数据来实现低水平的 dropout ,使用DCA 来对 具有中等dropout水平的数据集的插补。所研究的插补方法都不允许可靠地重建高级别辍学的相关网络。插补既没有明显阻碍也没有促进细胞簇注释。只有 DCA 略微改善了细胞的聚类。
如果 dropout 得到足够好的控制,从 scRNA-seq 数据中识别细胞类型特定网络特性在技术上似乎是可行的。这是否能够产生新的生物学见解仍有待证明。
























 1469
1469











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








