《DeepARG: a deep learning approach for predicting antibiotic resistance genes from metagenomic data》
文章地址:https://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-018-0401-z
DOI:10.1093/nar/gkae235
期刊:Nucleic Acids Research
2023影响因子/中科院分区:14.9/中科院2区
发布时间:2024年4月4日
PubTabor3.0: 一种强效AI文献资源解锁生物医药知识的工具
tools:https://www.ncbi.nlm.nih.gov/research/pubtator3/
摘要:
PubTator 3.0(https://www.ncbi.nlm.gov/research/pubtator3/)是一个生物医学文献资源,它使用最先进的人工智能技术提供语义和关系搜索,用于关键概念如蛋白质、遗传变异、疾病和化学物质。目前,它提供了超过十亿个实体和关系注释,涵盖了大约3600万篇PubMed摘要和600万篇来自PMC开放获取子集的全文文章,并每周更新。PubTator 3.0的在线界面和API利用这些预先计算的实体关系和同义词提供高级搜索功能,并支持大规模分析,简化了许多复杂信息需求。我们通过一系列实体对查询展示了PubTator 3.0的检索质量,证明PubTator 3.0检索到的文章数量多于PubMed或Google Scholar,并且在前20个结果中的精确度更高。我们进一步展示了将ChatGPT(GPT-4)与PubTator API集成,显著提高了其响应的事实性和可验证性。总之,PubTator 3.0提供了一套全面的功能和工具,使研究人员能够浏览不断增长的生物医学文献的财富,加快研究并为科学发现提供有价值的见解。
背景
生物医学文献是解决生物科学和临床科学信息需求的主要资源(1),然而对文献搜索的需求差异很大。例如,制定研究假设等活动需要一种探索性的方法,而解释遗传变异的临床意义等任务则更加集中。
传统的基于关键词的搜索方法长期以来一直是生物医学文献搜索的基础(2)。虽然这些方法通常对基本搜索效果不错,但也有显著的局限性,例如由于术语不同而错过相关文章,或因为表面层的术语匹配不能充分代表查询词所需的关联而包含不相关的文章。这些局限性耗费时间,并有风险使信息需求得不到满足。
自然语言处理(NLP)方法在创建生物信息学资源方面提供了极大的价值(3-5),并可以通过启用语义和关系搜索来改进文献搜索(6)。在语义搜索中,用户指出感兴趣的特定概念(实体),系统已经预先计算了与使用的术语无关的匹配项。关系搜索通过允许用户指定实体之间所需的关系类型(例如,化学物质是否增强或减少基因的表达)来提高精确度。在这方面,我们介绍了PubTator 3.0,这是一个新颖的资源,旨在支持生物医学文献中的语义和关系搜索。它的搜索能力允许用户探索六个关键生物医学实体的自动化实体注释:基因、疾病、化学物质、遗传变异、物种和细胞系。PubTator 3.0还识别并使12种常见实体间关系可搜索,增强了其对目标和探索性搜索的实用性。专注于生物医学科学中感兴趣的关系和实体类型使PubTator 3.0能够精确检索信息,同时提供广泛的实用性(详见补充表S1中的详细比较与其前身)。
总览
PubTator 3.0的在线界面,如图1和补充图S1所示,旨在支持交互式文献探索,支持语义、关系、关键词和布尔查询。自动完成功能提供语义搜索建议,帮助用户构建查询。例如,它会自动建议将“COVID-19”或“SARS-CoV-2感染”替换为语义术语“@DISEASE_COVID_19”。关系查询——在PubTator 3.0中是新功能——提供了更高的精确度,允许用户定位讨论实体间特定关系的文章。


PubTator 3.0提供统一的搜索结果,同时搜索大约3600万篇PubMed摘要和超过600万篇来自PMC开放获取子集(PMC-OA)的全文文章,提高了对文章全文中大量相关信息的访问能力(7)。搜索结果根据查询词之间关系的深度进行优先排序:包含语义术语之间可识别关系的全文文章获得最高优先级,而语义或关键词术语相邻出现的文章(例如,在同一句话中)获得次要优先级。搜索结果还根据匹配出现的章节进行优先排序(例如,标题中的匹配获得更高优先级)。用户可以通过使用过滤器进一步细化结果,将返回的文章缩小到特定的出版物类型、期刊或文章章节。
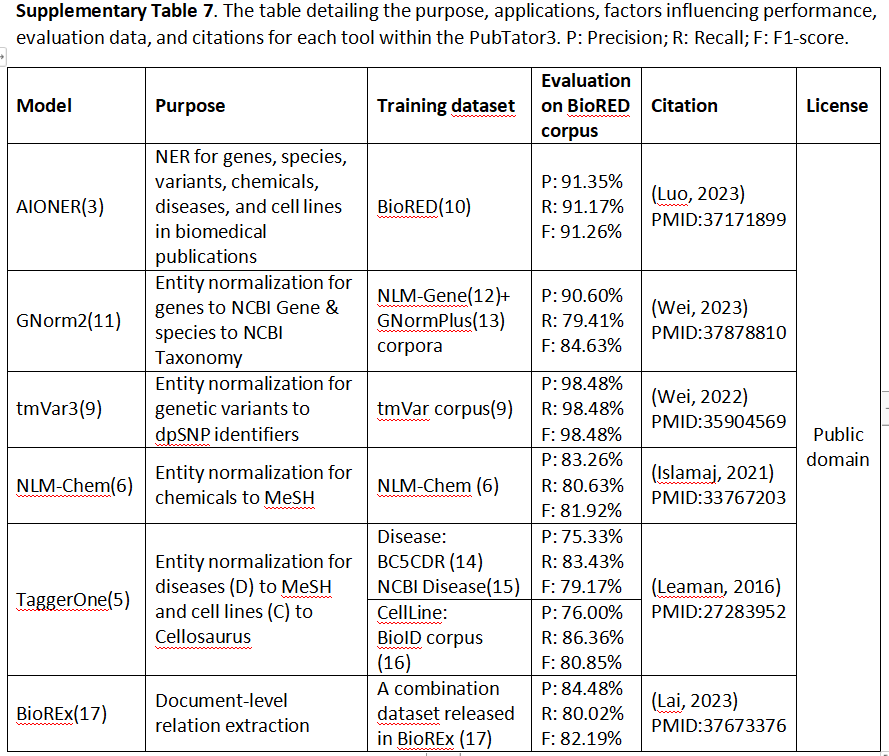
PubTator 3.0由自然语言处理(NLP)流程支持,如图2A所示。这个流程每周运行一次,首先识别新添加到PubMed和PMC-OA的文章。然后,文章通过三个主要步骤进行处理:(i)命名实体识别,由最近开发的深度学习变换器模型AIONER提供(8),(ii)标识映射和(iii)关系提取,由BioREx执行,用于12种常见类型的关系(在补充表S2中描述)。

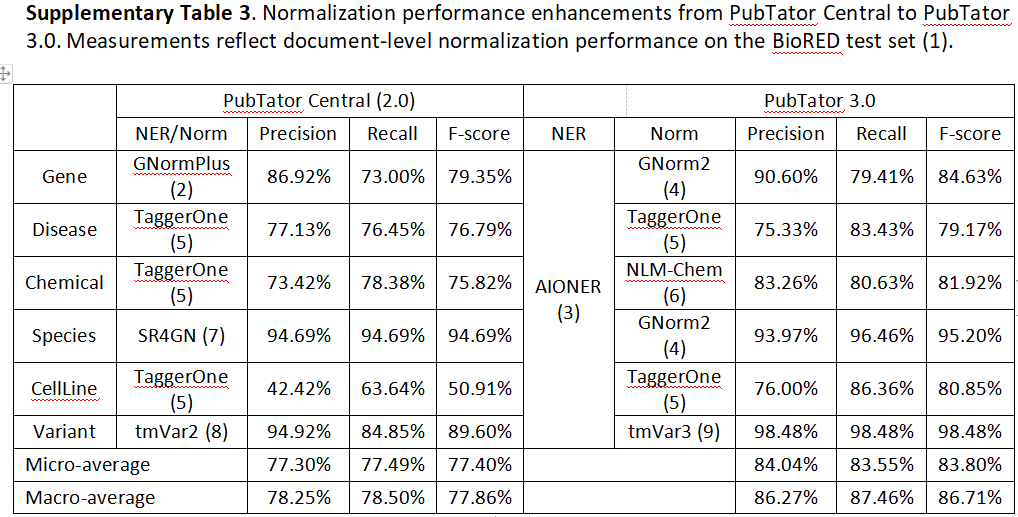
总体而言,PubTator 3.0包含了超过16亿个实体注释(460万个唯一标识符)和3300万个关系(880万个唯一对)。它在实体识别和标准化性能方面,相较于其前身版本PubTator 2(10),也即PubTator Central(见图2B和补充表S3),提供了增强的性能。我们在图2C中展示了PubTator 3.0的关系提取性能,并将其与**BioCreative V Chemical-Disease Relation(14)语料库上的先前最先进系统(11-13)**进行了比较,发现PubTator 3.0提供了显著更高的准确性。此外,当评估与PubMed和Google Scholar相比的随机样本实体对查询时,PubTator 3.0在前20个结果中一致地返回了更多文章,并具有更高的精确度(见图2D和补充表S4)。
Materials and methods
数据来源和文章处理
PubTator 3.0每周从BioC PubMed API(https://www.ncbi.nlm.nih.gov/research/bionlp/APIs/BioC-PubMed/)和BioC PMC API(https://www.ncbi.nlm.nih.gov/research/bionlp/APIs/BioC-PMC/)下载新文章,格式为BioC-XML(16)。使用Ab3P(17)识别本地缩写。文章文本和提取的数据使用MongoDB内部存储,并使用Solr进行索引以进行搜索,确保了强大且可扩展的可访问性,不受NCBI eUtils API等外部依赖的限制。
Entity recognition and normalization/linking
PubTator 3.0 使用了 AIONER(8),这是一个最近开发的命名实体识别(NER)模型,用于识别六种类型的实体:基因/蛋白质、化学物质、疾病、物种、遗传变异和细胞系。AIONER 利用一个灵活的标记方案,将分别创建的训练数据整合到单一资源中。这些训练数据集包括 NLM-Gene(18)、NLM-Chem(19)、NCBI-Disease(20)、BC5CDR(14)、tmVar3(21)、Species-800(22)、BioID(23)和 BioRED(15)。这种整合创建了一个更大的训练集,提高了模型对未见过数据的泛化能力。此外,它还支持同时识别多种实体类型,提高了效率,并简化了区分引用其他实体的实体之间的界限的挑战,例如疾病“α1-抗胰蛋白酶缺乏症”和蛋白质“α1-抗胰蛋白酶”。
我们在 14 个基准数据集(8)上评估了 AIONER 的性能,包括上述训练集的测试集。这次评估表明,AIONER 的性能超越或匹配了以前的最先进方法。
AIONER 找到的实体提及被标准化(链接)到适当实体数据库中的唯一标识符。标准化是由为每种实体类型设计(或适应)的模块执行的,使用最新版本。最近升级的 GNorm2 系统(24)将基因标准化为 NCBI Gene 标识符,并将物种提及标准化为 NCBI 分类法。tmVar3(21),也最近升级了,将遗传变异标准化;它使用 dbSNP 标识符为在 dbSNP 中列出的变异,否则使用 HGNV 格式。化学物质由 NLM-Chem 标记器(19)标准化为 MeSH 标识符(25)。TaggerOne(26)将疾病标准化为 MeSH,并将细胞系标准化为 Cellosaurus(27),使用一种新的仅标准化模式。这种模式只应用标准化模型,将提及和词汇表名称都转换为高维 TF-IDF 向量,并学习映射,如前所述。然而,它现在通过将每个词汇表名称映射到自身来增强训练数据,从而显著提高了在词汇表中但不在注释训练数据中的名称的性能。这些增强为实体标准化性能提供了显著的整体改进(补充表 S3)。
Relation extraction
PubTator 3.0的关系由统一的关系提取模型BioREx(9)提取,该模型旨在同时提取八种实体类型对之间的12种关系类型:化学物质-化学物质、化学物质-疾病、化学物质-基因、化学物质-变异、疾病-基因、疾病-变异、基因-基因和变异-变异。这些关系类型及其对应的实体对的详细定义在补充表S2中呈现。像BioREx这样的深度学习方法需要大量的训练数据。然而,关系提取的训练数据被分割成许多数据集,通常针对特定的实体对。BioREx通过数据为中心的方法克服了这一限制,协调不同训练数据集之间的差异,构建了一个全面、统一的数据集。
| PubTator 3.0 Relations | Description | Entity types |
|---|---|---|
| ASSOCIATE | Complex or unclear relationships | Chemical / Disease Chemical / Gene Chemical / Variant Disease / Gene Disease / Variant Variant / Variant |
| CAUSE | Triggering a disease by a specific agent | Chemical / Disease Variant / Disease |
| COMPARE | Comparing the effects of two chemicals or drugs | Chemical / Chemical |
| COTREAT | Simultaneous administration of multiple drugs | Chemical / Chemical |
| DRUG_INTERACT | Pharmacodynamic interactions between two chemicals | Chemical / Chemical |
| INHIBIT | Reduction in amount or degree of one entity by another | Chemical / Variant Gene / Disease |
| INTERACT | Physical interactions, such as protein-binding | Chemical / Gene Chemical / Variant Gene / Gene |
| NEGATIVE_CORRELATE | Increases in the amount or degree of one entity decreases the amount or degree of the other entity | Chemical / Gene Chemical / Variant Gene / Gene |
| POSITIVE_CORRELATE | The amount or degree of two entities increase or decrease together | Chemical / Chemical Chemical / Gene Gene / Gene |
| PREVENT | Prevention of a disease by a genetic variant | Variant / Disease |
| STIMULATE | Increase in amount or degree of one entity by another | Chemical / Variant Gene / Disease |
| TREAT | Treatment of a disease using a chemical or drug | Chemical / Disease |
我们通过使用手动注释的关系提取数据集的性能以及BioREx与值得注意的可比系统之间的比较分析来评估BioREx提取的关系。BioREx在BioRED语料库测试集(15)上建立了新的性能基准,将性能从74.4%(F分数)提高到79.6%,并展示了比替代模型如迁移学习(TL)、多任务学习(MTL)以及在孤立数据集上训练的最先进模型更高的性能(9)。对于PubTator 3.0,我们将其深度学习模块PubMedBERT(28)替换为LinkBERT(29),进一步将性能提高到82.0%。此外,我们使用BioCreative V Chemical Disease Relation语料库测试集(14)对BioREx与广泛使用的基于规则的提取多种关系的方法**SemRep(11)、CD-REST(**13)系统以及之前最先进系统(12)进行了比较分析。我们的评估表明,PubTator 3.0比之前的方法提供了显著更高的F分数。
Programmatic access and data formats
PubTator 3.0通过其API和批量下载提供程序化访问。API(https://www.ncbi.nlm.nih.gov/research/pubtator3/)支持关键词、实体和关系搜索,并且还支持以基于XML和JSON的BioC(16)格式以及制表符分隔的自由文本导出注释。PubTator 3.0的FTP站点(https://ftp.ncbi.nlm.nih.gov/pub/lu/PubTator3)提供注释文章和实体与关系的提取摘要的批量下载。程序化访问支持更灵活的查询选项;例如,信息需求“哪些化学物质能减少JAK1的表达?”可以通过API直接回答(例如 https://www.ncbi.nlm.nih.gov/research/pubtator3-api/relations?e1=@GENE_JAK1&type=negative_correlate&e2=Chemical)或通过过滤批量关系文件。此外,PubTator 3.0 API还支持用户定义的自由文本的注释。
Case study I: entity relation queries
实体关系搜索
我们通过准备一系列12个实体对作为案例研究,分析了PubTator 3.0的检索质量,并将其与PubMed和Google Scholar进行了比较。为了提供平等的比较,我们筛选了Google Scholar大约30%的结果,以排除那些PubMed中不存在的文章。为了确保结果数量足够低,以便筛选出PubMed中没有的文章,我们首先确定了2022年或之后首次在文献中一起讨论的实体对。然后我们随机选择了以下每种类型的两个实体对:疾病/基因、化学物质/疾病、化学物质/基因、化学物质/化学物质、基因/基因和疾病/变异。所选的任何关系对都没有出现在训练集中。比较是在2023年5月19日所有搜索引擎返回的搜索结果快照的基础上进行的。我们手动评估了每个系统和每个查询的前20个结果;如果文章提及了查询中的两个实体,并支持它们之间的关系,则被判断为相关。两位策展人独立地对每篇文章进行了判断,如有差异则进行讨论直至达成一致。策展人对检索方法没有盲审,但需要记录支持关系的文本(如果相关)。这个实验评估了每种检索方法前20个结果的相关性,无论文章是否出现在PubMed中。
我们的分析总结在图2D中,补充表S4提供了PubTator 3.0、PubMed和Google Scholar检索结果质量的详细比较。我们的结果表明,PubTator 3.0检索到的文章数量比比较系统多,并且其前20个结果的精确度更高。例如,对于查询“GLPG0634 + 溃疡性结肠炎”,PubTator 3.0返回了346篇文章,手动审查前20篇文章显示所有文章都包含了关于GLPG0634和溃疡性结肠炎之间关联的陈述。相比之下,PubMed只返回了总共18篇文章,其中只有12篇提到了关联。此外,在搜索“COVID19 + PON1”时,PubTator 3.0返回了PubMed中的212篇文章,超过了Google Scholar获得的43篇文章,其中只有29篇来自PubMed。这些差异可以归因于几个因素:(i) PubTator 3.0的搜索包括PMC-OA中可用的全文,从而显著扩大了文章的覆盖范围,(ii) 实体标准化提高了召回率,例如,将“paraoxonase 1”匹配到“PON1”,(iii) PubTator 3.0优先考虑包含查询实体之间关系的文章,(iv) PubTator 3.0优先考虑实体附近出现的文章,而不是远离的段落。在12个信息检索案例研究中,PubTator 3.0对前20篇文章的整体精确度为90.0%(216篇中的240篇),显著高于PubMed的精确度81.6%(103篇中的84篇)和Google Scholar的精确度48.5%(202篇中的98篇)
**注释:**使用数量评估是否不客观,从引用数,或者文章杂志影响因子来判定
Case study II: retrieval-augmented generation
在大型语言模型(LLMs)的时代,PubTator 3.0也可以通过检索增强生成来提高它们的事实准确性。尽管LLMs在语言能力上非常强大,但它们倾向于生成错误的断言,有时被称为幻觉(30,31)。例如,当被要求引用诸如“多柔比星能治疗哪些疾病”之类的问题时,GPT-4经常提供看似合理但实际上不存在的引用。通过使用PubTator 3.0 API增强GPT-4,可以通过提取的关系将模型的响应锚定到可验证的引用上,显著减少幻觉。
我们评估了三种GPT-4变体的回答引用准确性:PubTator增强的GPT-4、PubMed增强的GPT-4和标准GPT-4。我们进行了基于八个问题的定性评估,这些问题的选择如下:我们确定了PubMed查询日志中提到的实体,并从频繁搜索和偶尔搜索的实体中随机选择。然后,我们确定了每个实体的常见查询,这些查询请求关系信息,并将其适应为自然语言问题。因此,每个问题都基于真实PubMed用户的常见信息需求。例如,“Tocilizumab可能引起什么?”和“Doxorubicin能治疗什么?”这些问题分别改编自用户查询“Tocilizumab副作用”和“Doxorubicin治疗”。这类问题通常需要从多篇文章中提取信息,并理解生物医学实体和关系描述。补充表S5列出了所选的问题。
我们通过OpenAI ChatCompletion API的函数调用机制,使用PubTator 3.0增强了GPT-4大型语言模型(LLM)。这种集成涉及用三个PubTator API的描述提示GPT-4:(i) 查找实体ID,检索PubTator实体标识符;(ii) 查找相关实体,根据输入实体和指定的关系识别相关实体;(iii) 导出相关搜索结果,返回包含特定实体关系文本证据的PubMed文章标识符。我们的指令提示GPT-4将用户问题分解为这些API可以解决的子问题,执行函数调用,并将响应综合成一个连贯的最终答案。我们的提示通过指示GPT-4以“摘要:”开始其消息来促进总结性响应,并要求响应包括提供证据的文章引用。PubMed增强实验通过国家生物技术信息中心(NCBI)E-utils API为GPT-4提供了PubMed数据库搜索的访问权限(32)。我们使用了Azure OpenAI服务(版本2023-07-01-preview)和GPT-4(版本2023-06-13),并将解码温度设置为零以获得确定性输出。完整的提示提供在补充表S6中。
PubTator增强的GPT-4通常按三个步骤处理问题:(i) 查找标准实体标识符,(ii) 查找其相关实体标识符,(iii) 搜索PubMed文章。例如,为了回答“哪些药物可以治疗乳腺癌?”,GPT-4首先使用Find Entity ID API找到了乳腺癌的PubTator实体标识符(@DISEASE_Breast_Cancer)。然后,它使用Find Related Entities API通过“治疗”关系来识别与@DISEASE_Breast_Cancer相关的实体。为了演示,我们将输出实体的最大数量限制为五个。最后,GPT-4调用了Export Relevant Search Results API,以获取包含这些关系的证据的PubMed文章标识符。每种方法的每个提示的原始响应提供在补充表S6中。
我们通过审查每篇PubMed文章并验证每个引用的PubMed文章是否支持所述关系(例如,他莫昔芬治疗乳腺癌),手动评估了回答中引用的准确性。补充表S5报告了每种方法引用文章具有有效支持证据的比例。GPT-4经常生成伪造的引用,这被广泛称为幻觉问题。尽管PubMed增强的GPT-4显示了更高的准确引用比例,但一些引用的文章并不支持关系声明。这可能是因为PubMed基于关键词和布尔搜索,不支持特定关系的查询。由PubTator增强的GPT-4生成的响应展示了最高水平的引用准确性,强调了PubTator 3.0作为通过检索增强生成与LLMs(如GPT-4)一起解决生物医学信息需求的高质量知识源的潜力。在我们的实验中,使用Azure进行ChatGPT,使用GPT-4-Turbo的成本大约是1美元用于两个问题,或者当降级到GPT-3.5-Turbo时,可以问40个问题,包括输入/输出标记的成本。
Discussion
自2015年以来,PubTator的以前版本已经满足了超过十亿次API请求,支持了广泛的研究应用。许多研究利用PubTator注释进行特定疾病的基因研究,包括优先考虑候选基因(33)、确定基因-表型关联(34)以及识别疾病共病的遗传基础(35)。一些项目使用PubTator创建基因和遗传变异资源(36,37)或丰富疾病知识图谱(38,39)。此外,PubTator支持生物信息学工作(40,41)和创建自然语言处理基准(42)。具有提高的准确性,PubTator 3.0将更好地支持这些用例。
向PubTator 3.0引入关系注释为扩大使用场景开辟了新的途径。有了从文献中预先计算的关系,通常可以直接回答复杂的研究问题。例如,药物再利用可以被制定为识别针对特定基因的化学物质。相反,通过查询相同的化学/基因关系,可以确定化学物质的遗传目标。评估遗传变异的临床医生,例如针对罕见疾病或个性化医学,可能会探索特定遗传变异与疾病之间的关系。另一方面,生物学家可能会利用多个基因之间的相互作用来组装复杂的分子途径。
PubTator 3.0有几个值得注意的限制。尽管它能够从全文文章中提取关系,但由于计算限制,这个特性目前仅限于摘要。然而,该系统已经被设计为在未来的增强中支持全文关系提取。当前系统只提取12种关系类型,尽管这些代表常见的用途。最后,实体注释和关系提取是自动化的;尽管这些系统表现出高性能,它们的准确性仍然不完美。
Conclusion
PubTator 3.0 提供了一套全面的功能和工具,使研究人员能够浏览不断增长的生物医学文献的财富,加快研究并为科学发现提供有价值的见解。PubTator 3.0 的界面、API 和批量文件下载可在 https://www.ncbi.nlm.nih.gov/research/pubtator3/ 上获得。
Data availability
数据可以通过在线界面: https://www.ncbi.nlm.nih.gov/research/pubtator3/
通过 API: https://www.ncbi.nlm.nih.gov/research/pubtator3/api
批量 FTP 下载: https://ftp.ncbi.nlm.nih.gov/pub/lu/PubTator3/ 访问。
PubTator 3.0 每个组件的源代码都是公开可访问的。
AIONER 命名实体识别器可在: https://github.com/ncbi/AIONER 。
用于基因名称标准化的 GNorm2 可在: https://github.com/ncbi/GNorm2 。
用于变异名称标准化的 tmVar3 可在: https://github.com/ncbi/tmVar3 上获取。
用于化学物质名称标准化的 NLM-Chem Tagger 可在: https://ftp.ncbi.nlm.nih.gov/pub/lu/NLMChem 上获取。
用于疾病和细胞系标准化的 TaggerOne 系统可在: https://www.ncbi.nlm.nih.gov/research/bionlp/Tools/taggerone 上获取。
BioREx 关系提取系统可在: https://github.com/ncbi/BioREx 上获取。
定制 ChatGPT 以使用 PubTator 3.0 API 的代码可在: https://github.com/ncbi-nlp/pubtator-gpt 上获取。
每个工具的应用、性能、评估数据和引用详情都显示在补充表 S7 中。所有源代码也可在 https://doi.org/10.5281/zenodo.10839630 上获取。























 620
620

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








