Tips:权限申请:访问 ADNI官网 根据提示按部就班来,不麻烦,一般几天就会发邮件审核通过,网上也有很多写申请教程的博客,可以搜来看看。
快速索引
数据集下载
0 概览
登录状态下访问官网:
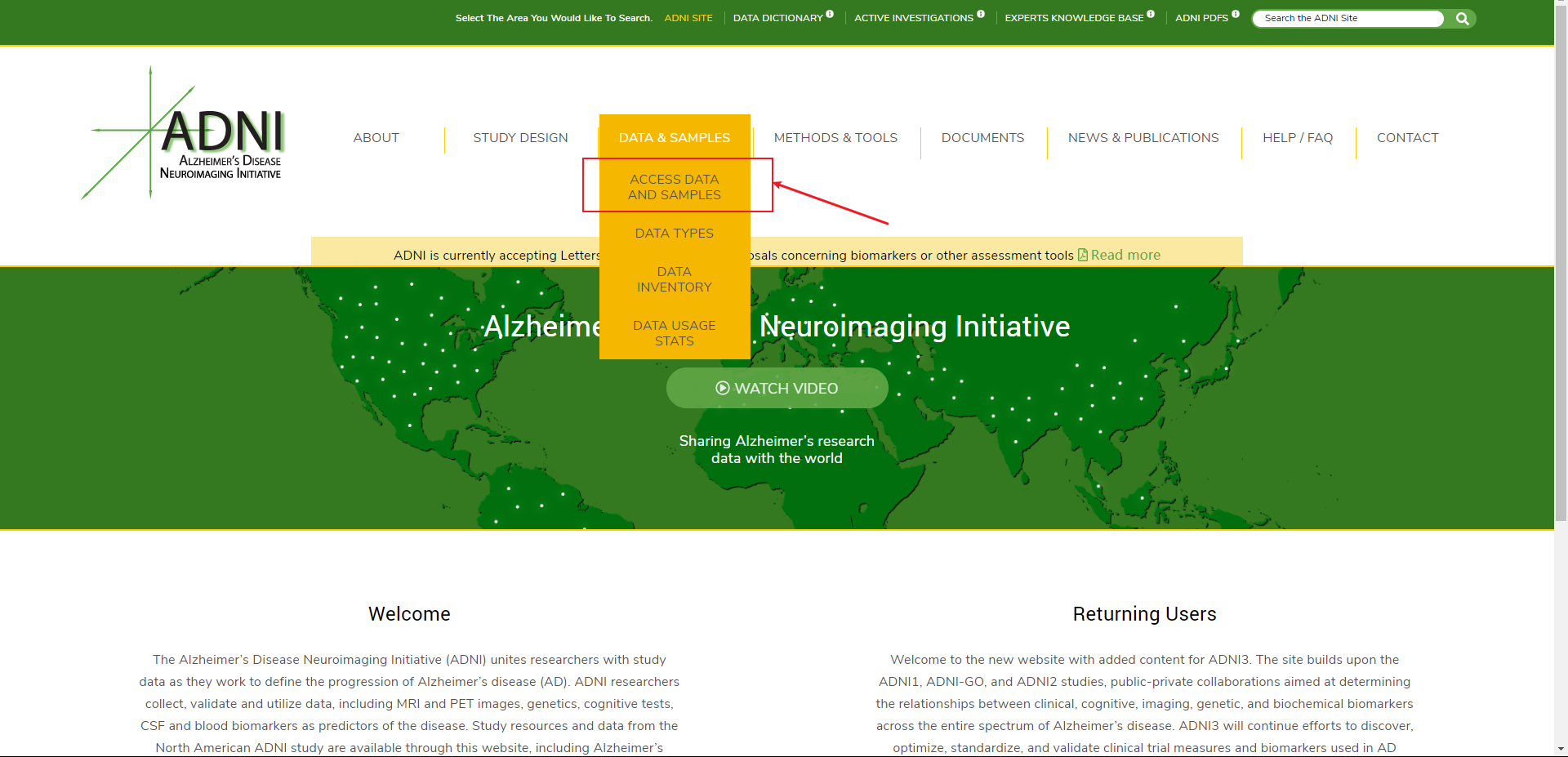
点击红框指示部分后,进入如下界面:

继续点击选中部分:

- search:初级搜索
- Advanced Search:高级搜索,提供更多筛选参数
- Data Collections:
- 数据收集处,可在筛选的时候将影像图片添加到指定文件夹中
- 筛选完后在此处统一打包下载( csv,zip )
- 可选择社区提供的Public Collections( 以便统一标准横向比较 )
1 筛选数据集
1.1 自定义筛选 - Advanced Search
选择Advanced Search (beta)进入自定义筛选页面

1.2 ADNI共享数据集 - Data Collections
在Data collections页面左侧,可以看到ADNI上一些已打包好的共享的数据集,也可以来选择下载

-
数据的采集时间:1Yr,2Yr…
-
扫描场强:1.5T,3T…
2 下载数据集
筛选完数据之后,就可进入Data Collection页面下载数据
可供下载的数据如下:
- csv:图像对应的标签,csv格式
- Metadata:图像对应的详细临床信息,xml格式,可用程序提取有用的信息加入到csv标签中
- Zip File:打包后的MRI扫描图像,nii格式
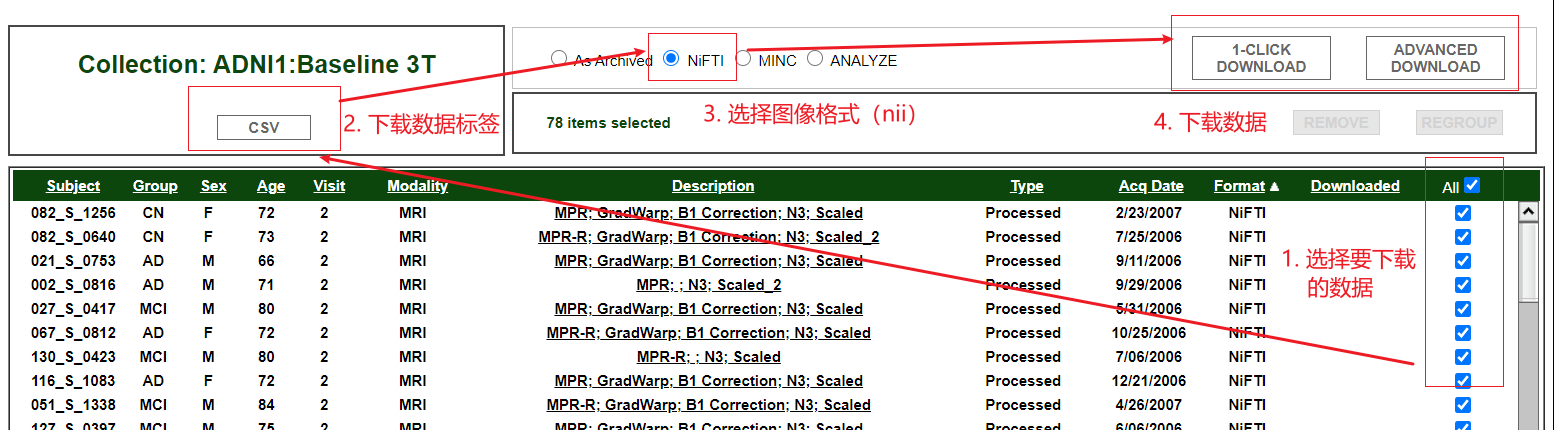
其中页面右上角两种下载数据方式区别如下:
- 1-CLICK:一键下载
- ADVANCED:分多个压缩包下载

数据初步处理
可视化处理
import nibabel as nib
import matplotlib.pyplot as plt
import numpy as np
img_path = r'D:\Dataset\ADNI_dataset\ADNI1_Annual_2_Yr_3T\ADNI\002_S_1261\MPR____N3__Scaled\2008-05-27_18_41_16.0\S60591\ADNI_002_S_1261_MR_MPR____N3__Scaled_Br_20081224110926692_S60591_I132220.nii'
image_data = nib.load(img_path).get_fdata()
x, y, z = image_data.shape # 获取三个维度上的长度
for _ in range(x): # 自定义横截面打印 (x or y or z, 相应的列表切片位置也要改变)
print(_)
plt.imshow(image_data[_, :, :], cmap='gray')
plt.show()
数据加载
import nibabel as nib
img_path = r'D:\Dataset\ADNI_dataset\ADNI1_Annual_2_Yr_3T\ADNI\002_S_1261\MPR____N3__Scaled\2008-05-27_18_41_16.0\S605 91\ADNI_002_S_1261_MR_MPR____N3__Scaled_Br_20081224110926692_S60591_I132220.nii'
image = nib.load(img_path)
print(image)
数据预处理
-
转换图像类型,图像分割(ROI区域),像素归一化处理,高斯滤波,平滑处理…
-
这里先不再作详细解释(因为我还不会😂,等之后学习到了再总结🐕),详见参考链接: ADNI数据预处理
参考链接























 2万+
2万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








