因为有对三维nii骨骼数据标注处理的需要,通过Arima软件分割出来的不同骨块需要灰度值处理才能在可视化软件中显现出肉眼可见的颜色区别方便进行校正、筛选或者其他的处理步骤。故重新捡起nibabel和SimpleITK来读取存储nii文件,在使用过程中发现了两个库对于nii文件的处理内部颇有些不同,在arima软件中的适配度也不一样,所以记录一下。
用Arima导出的单部分mask的背景均为0,骨骼均为1,所以需要读入后筛选值为1的元素进行修改。
nibabel:
import numpy as np
import nibabel as nib
nii_path = r'\10\L.nii'
src_img = nib.load(nii_path)
src_data = src_img.get_fdata()
modified_data = np.copy(src_data)
mask_indexes = np.where(src_data == 1)
modified_data[mask_indexes] += 1023
print(modified_data)
nii_img = nib.Nifti1Image(modified_data, src_img.affine, src_img.header)
nib.save(nii_img, r'\10\L_1.nii')SimpleITK
import numpy as np
import nibabel as nib
import os
import SimpleITK as sitk
img_name = r'57\S.nii'
ct = sitk.ReadImage(img_name, sitk.sitkInt16)
ct = sitk.GetArrayFromImage(ct)
modified_data = ct
mask_indexes = np.where(ct == 1)
modified_data[mask_indexes] += 1023
print(modified_data)
modified_data = sitk.GetImageFromArray(modified_data)
sitk.WriteImage(modified_data, r'57\S_1.nii')可以看出两端基础代码只是在读取和存入的时候不同,都希望更改为1024的灰度值,且用ITK-Snap查看图片信息的min-max值都为0-1024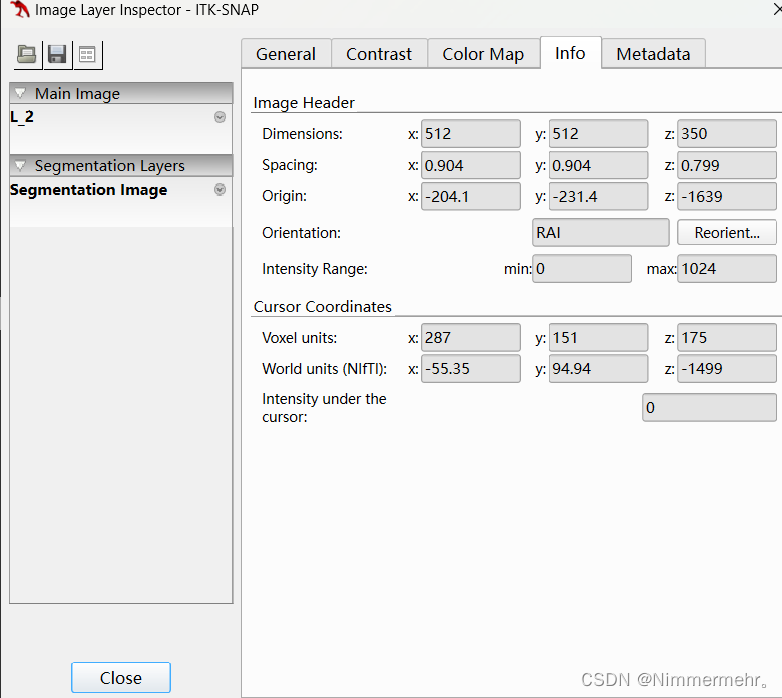
但是nibabel在Arima医疗软件中显示值固定为0-255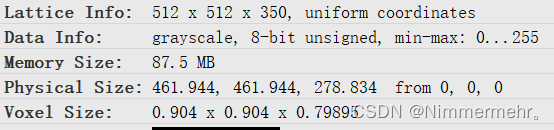
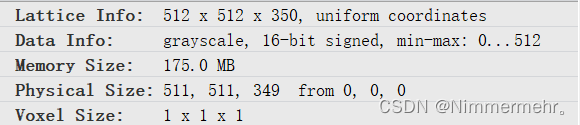
SimpleITK在Arima医疗软件中显示值则为正常的0-1024(512)等自定义的值
显示于融合mask中的结果就更明显
nibabel:
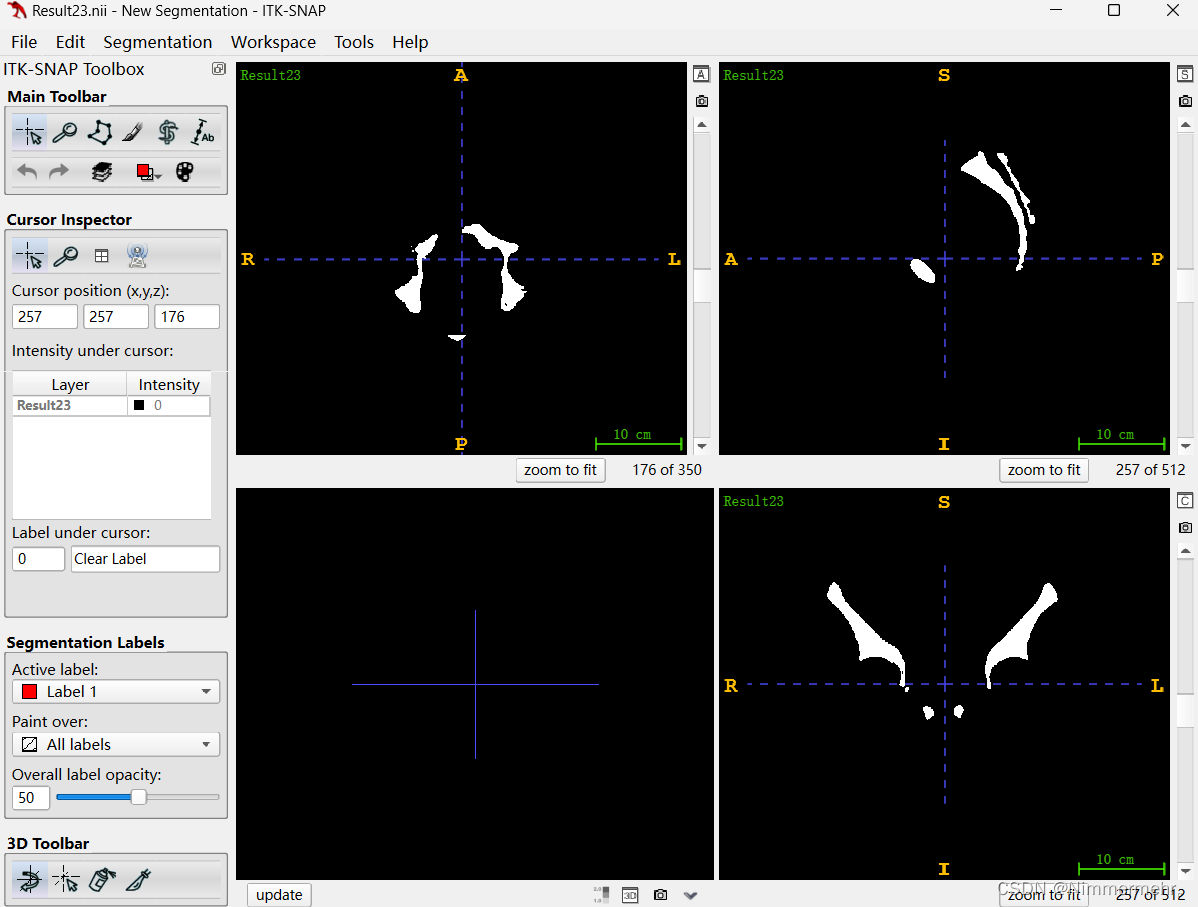
SimpleITK:

没有深入了解过这两个库的源码,不知道底层区别,但基于结果,建议在nii中使用SimpleITK库进行处理,效果更理想






















 773
773











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










