简报:在有着蛋白质结构预测领域奥林匹克竞赛之称的最新一届CASP比赛中(CASP15),密西根大学张阳教授和Peter Freddolino教授实验室的郑伟博士在多个比赛项目中获得冠军。其中D-I-TASSER算法(参赛名:“UM-TBM”)在单结构域蛋白质预测比赛中(按Z-score>-2)排名第一;D-I-TASSER算法(参赛名:“UM-TBM”)在多结构域蛋白质预测比赛中排名第一;DMF-Multimer算法(参赛名:“Zheng”)在在蛋白质-蛋白质复合体预测比赛中排名第一。其中,在后两项比赛中,郑伟博士开发的算法大幅领先于其他算法。
背景
蛋白质分子是生命活动的主要承担者。它们通过形成特定的三维空间结构,进而与细胞中其他分子形成特殊的相互作用来执行相应的生物学功能,即“结构决定功能”。了解蛋白质的三维结构也就成为理解生命活动的关键。一直以来,蛋白质的结构主要通过结构生物学实验手段获得,而这些实验手段需要花费大量的人力和物力。因此,通过实验手段获得的蛋白质结构数目仍然有限。多年来,安芬森关于“蛋白质的氨基酸序列应该完全决定其结构”的假定,一直激励计算生物学家们积极探索从序列出发直接预测高精度的蛋白质的三维结构的可能性。
这方面的探索近年来终于迎来了里程碑式的突破。2020年,DeepMind团队开发出新的端到端深度学习AlphaFold2算法,在第14届世界蛋白质结构预测比赛中(CASP14),成功在约2/3的目标蛋白上取得了可以与实验测定的结构相媲美的精度。CASP由马里兰大学John Moult教授等人在1994年创立,每两年举行一次,旨在对领域内的蛋白质结构预测技术作客观的测试和评价。该实验采用严格的双盲预测机制,被誉为评估蛋白质结构预测技术的金标准,也被业界视为“蛋白质结构预测领域的奥林匹克竞赛”。根据CASP赛事规则,所有参赛方法分为服务器组和人工组两类。服务器组参赛者只有72小时进行结构预测;人工组参赛者有三周时间进行结构预测,且人工组允许参赛队伍进行人工干预。这意味着服务器组纯粹依赖计算机预测,因此,服务器组的参赛难度往往比人工组高。
AlphaFold2的成功初步证明了“序列决定结构”这一设想,对蛋白质结构预测领域具有重要意义。然而,AlphaFold2也逐渐暴露出一些不足,比如对于一些“自由建模”,特别是没有同源结构的孤儿蛋白质,AlphaFold2的预测精度仍然有限。并且,AlphaFold2对于结构域之间的建模和蛋白质-蛋白质相互作用的构象预测的精度也偏低。如何解决上述问题成为领域内的热点。
2022年的CASP15是AlphaFold2问世之后的第一届CASP比赛,密西根大学张阳实验室的郑伟博士参加了该比赛并给出了他的答案。郑伟博士毕业于南开大学,先后师从胡刚教授,阮吉寿教授学习蛋白质结构生物信息学基础。其中博士研究的最后两年,在密西根大学张阳教授实验室进行访问交流。博士毕业后在密西根大学张阳教授实验室,继续从事AI辅助蛋白质结构预测等相关研究。
密西根大学张阳教授在该领域研究背景深远。其课题组开发的I-TASSER系列算法自从2006年CASP7开始,连续8届在CASP竞赛自动服务器组中雄踞第一【见I-TASSER连续八届在CASP自动组竞赛中蝉联冠军】!截至目前统计,I-TASSER已经拥有17万以上的用户,为160多个国家和地区提供了超过70万个蛋白质的结构预测服务。早在15年前,张阳教授就在PNAS上发文,前瞻性地提出,通过已知实验数据集解决蛋白质结构预测问题理论上可能性。这一理论也被随后历届CASP赛事所验证。
此外,张阳实验室开发的蛋白质模板探测服务器(LOMETS、CEthreader),蛋白质从头结构预测算法(QUARK、C-QUARK),蛋白质几何势能预测算法(DeepPotential、TripletRes、ResPRE),同源序列(MSA)探测(DeepMSA),蛋白质和肽链设计算法(EvoDesign),以及蛋白质功能预测服务器(COFACTOR、COACH、BioLiP)等也被业界广泛使用。张阳教授提出的TM-score和TM-align算法已经成为蛋白质结构预测领域衡量和比对蛋白质结构的一个通用标准。
CASP15比赛结果
2022年5月到8月,为期四个月的CASP15比赛在蛋白质结构预测方面进行了两个类别的实验:(1)蛋白质单体结构预测(REGULAR);(2)蛋白质复合体结构预测(MULTIMER)。其中第一项单体预测根据目标蛋白质为单结构域还是多结构域分为两类进行评测,即单结构域评测(domain prediction)和多结构域整体评测(inter-domain prediction)。
今年的参赛队伍多于往届,并且有较多的国内组织参与,根据CASP15会议官方统计,今年来自中国的参赛队伍数量基本与美国持平。在蛋白质结构预测比赛中,来自国内的队伍包括:华深智药的Omegafold、清华产业研究院AIRfold、百度飞桨的HelixFold、深势科技的Uni-Fold、西湖大学李子青团队的Manifold、北京大学高毅勤团队联合华为的MEGA-FOLD、上海天壤的TRFold、芝加哥大学丰田研究中心的许锦波教授团队、上海科技大学团队、清华大学的四支队伍、中科院计算所的卜东波教授团队、浙江工业大学的张贵军教授团队,山东大学的杨建益教授团队,中国人民大学龚新奇教授团队,北京智源人工智能研究院等一众来自于学界工业界的强队。另外,国外的一些老牌强队,如华盛顿大学的David Baker教授团队、密苏里大学的程建林教授等四支队伍、伦敦大学的David Jones教授团队、密西根州立大学的Michael Feig教授团队、普渡大学的木原大辅教授团队、哈佛大学Sergey Ovchinnikov的ColabFold、哥伦比亚大学Mohammed AlQuraishi教授的OpenFold、Meta(原Facebook)的ESMFold、日本Infinite Curation公司等也都如期而至,可以说是高手林立。
密西根大学张阳教授实验室郑伟博士同时参加了蛋白质单体结构和蛋白质复合体结构两项比赛。本次是郑伟博士第三次参加CASP蛋白质结构预测相关比赛。2018年CASP13及2020年CASP14,在张阳教授的带领下,郑伟博士主持,李阳博士、张成辛博士等人团队协作开发的算法服务器C-I-TASSER、D-I-TASSER(CASP14版)两次获得CASP服务器组冠军。本次CASP,郑伟博士主持开发的D-I-TASSER算法(CASP15版)和DMF-Multimer算法在不同的赛项中斩获多项冠军。
1. 蛋白质单体单结构域预测比赛项目(Protein single chain and single-domain prediction)
蛋白质单体结构预测是CASP中历史最为悠久的比赛。根据其演化和功能,一个蛋白质可拆解为多个基本的结构单元,一般被称之为蛋白质的结构域(domain,图1)。CASP评测者会将所有参赛组提交的结构(Model)按照实验结构的结构域划分,进行拆解评测,以评价参赛算法的基本单元结构预测能力。今年这个赛道有共计135个参赛组(包括47个服务器组,88个人工组)参赛。最终132个参赛组提交了结构。其中郑伟博士开发的UM-TBM服务器(基于D-I-TASSER算法)、杨建益教授课题组的Yang-Server服务器和日本Infinite Curation公司小田贤幸的人工组PEZYFoldings分列前三,并与后续算法梯队拉开距离。
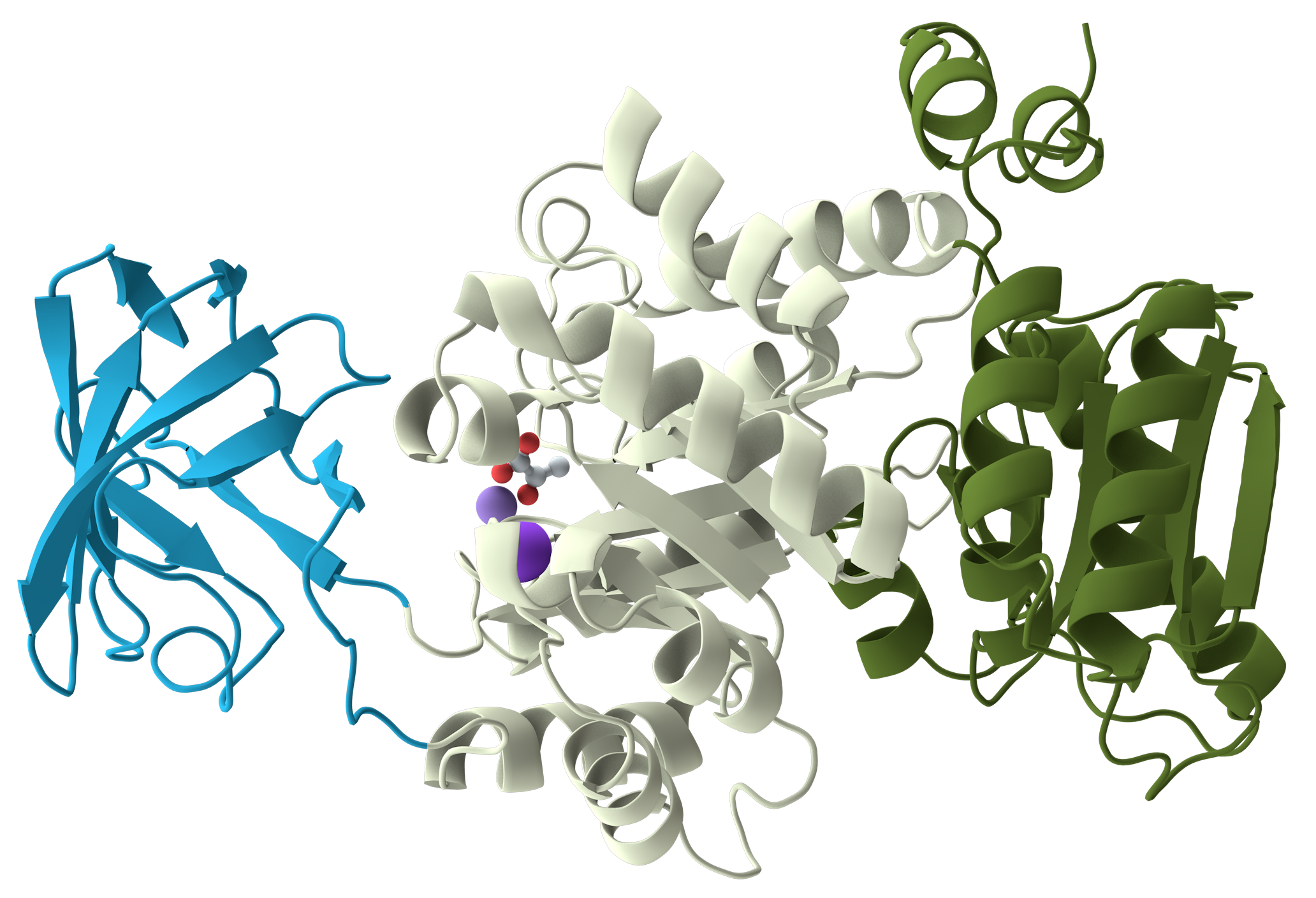
图1.多结构域蛋白质单体示意图。该蛋白质包含3个结构域(domain),不同结构域被标记为不同颜色。
前三名的打分基本持平,其中按照不同的打分评价函数及排序机制,三队各有千秋。如按照官方的GDT-TS打分计算并按照Z-score(>-2)去尾排序,郑伟博士开发的算法D-I-TASSER排名第一(图2A);按照官方的Z-score(>0)去尾排序,杨建益教授的Yang-Server排名第一(图2B);按照官方的GDT-HA等打分计算并按照Z-score(>-2)去尾排序,郑伟博士开发的算法D-I-TASSER再次排名第一(图2C);按照官方的Z-score(>0)去尾排序,则小田贤幸的人工组PEZYFoldings排名第一(图2D)。
由此可见,这个赛项竞争尤为激烈。其中GDT-TS打分主要用来评估蛋白质低分辨率结构,GDT-HA用于评测高分辨率结构。Z-score为一种统计指标,计算方式是(原始打分-参赛组均值)/参赛组标准差。Z-score用于衡量被评测算法与参赛队伍的平均值差异的大小。其中‘去尾’指的是当参赛队伍的Z-score小于某个阈值(-2或者0)时将被赋值为该阈值(这项去尾设计主要用于减少某些预测特别差的目标蛋白对一些创新团队整体排名的影响)。
值得一提的是,上次CASP14的来自于Google DeepMind开发的算法AlphaFold2的标准算法(NBIS-AF2-standard)也作为基线算法参与了比赛。但是无论何种评测排法,排名均在30名开外,显示出自CASP14以来,该领域算法的整体进展。
另外值得注意的是,历届CASP的整体排名,服务器组的精度是整体低于人工组的。本次CASP15的结果正好相反,最好的自动的服务器组精度要远好于大部分的人工组,甚至优于最好的人工组。这些进展显示,蛋白质结构预测领域的自动预测服务器已经成熟,能够提供完全独立的高精度结构预测。
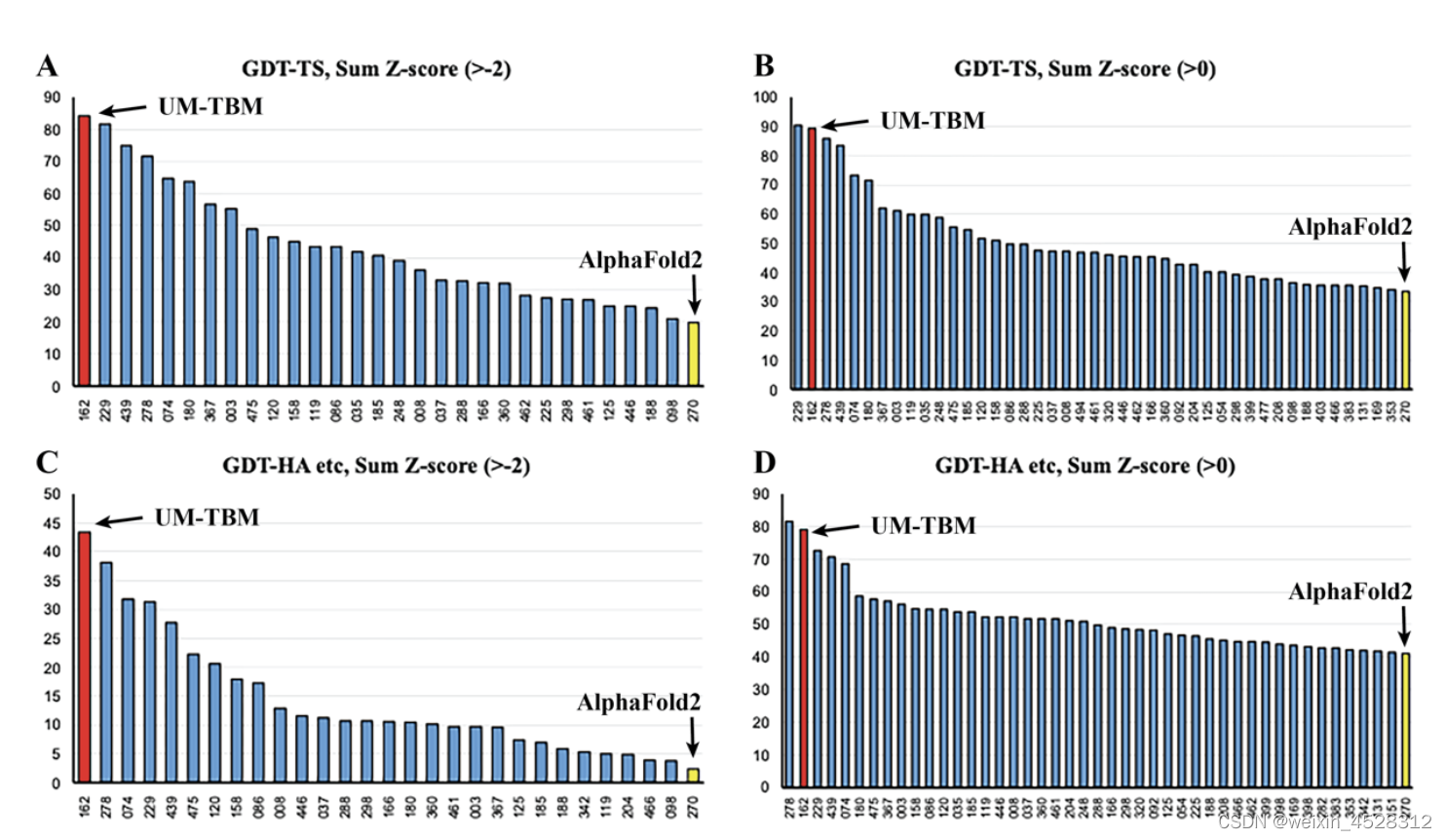
图2. CASP15在所有单体蛋白上预测结果基于单结构域的排名。红色为张阳实验室郑伟博士开发的D-I-TASSER算法,该服务器的参赛名字为‘UM-TBM’;黄色为标准版的AlphaFold2算法。(A) 基于GDT-TS指标的sum Z-score>-2.0进行的排名。(B) 基于GDT-TS指标的sum Z-score>0.0进行的排名。(C) 基于GDT-HA等指标的sum Z-score>-2.0进行的排名。(D) 基于GDT-HA等指标的sum Z-score>0.0进行的排名。数据来自CASP官方网站(https://predictioncenter.org/casp15/zscores_final.cgi)。
2. 蛋白质单体多结构域预测比赛项目(Protein single chain and multi-domain prediction)
由于历史原因,虽然CASP比赛对蛋白评测时会进行结构域区分,但是蛋白质在行使功能时往往以完整的单体结构(protein single chain)进行执行(图1)。所以对蛋白质整个单体结构,尤其是多结构域蛋白单体结构,进行评测往往更加能够反应蛋白质整体结构预测的能力。一直以来,因为额外自由度的引入,大体系多结构域蛋白质结构预测问题被认为要远远难于单结构域蛋白质结构预测。近年来,随着蛋白质结构预测的整体精度不断提升,CASP组委会也越来越重视蛋白质整体结构预测的精度。2020年CASP14比赛后,组委会增加了蛋白质单体多结构域评测项目。今年,在这个赛道中,共计98个参赛队伍参与了预测及评测。D-I-TASSER算法(UM-TBM服务器)再次以优异的成绩位列第一(图3)。其中D-I-TASSER算法打分(37.9)超出排名第二的算法打分(27.2)接近40%,并优于AlphaFold2标准算法打分(3.3)10倍有余。
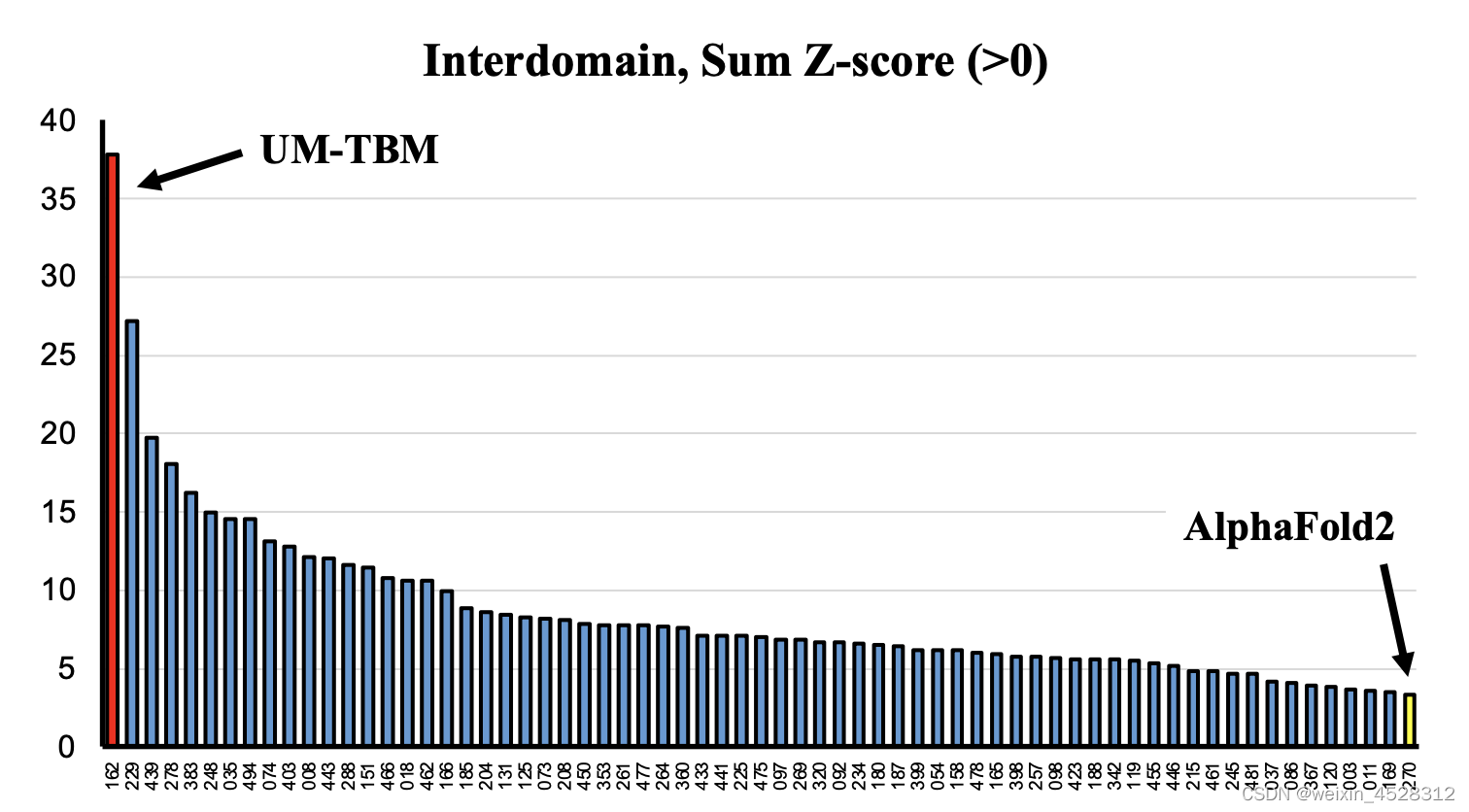
图3. CASP15参赛算法在所有多结构域蛋白上预测结果的排名(排名基于sum Z-score>0.0)。红色为张阳实验室郑伟博士开发的D-I-TASSER算法,该服务器的参赛名字为UM-TBM;黄色为标准版的AlphaFold2算法。数据取自CASP15官方网站(https://predictioncenter.org/casp15/zscores_interdomain.cgi)。
3. 蛋白质复合体结构预测比赛项目(Protein complex prediction)
蛋白质在生物体内正常行使功能实施,大部分是以复合物的形式进行的,比如,蛋白质-蛋白质(protein-protein),蛋白质-小分子(protein-ligand)相互作用的复合体。但是蛋白质-蛋白质复合体往往体系较大,其额外自由度的引入也让单体与单体之间的组合也多种多样(图4),因此,其预测难度被认为远远高于蛋白质单体结构预测。
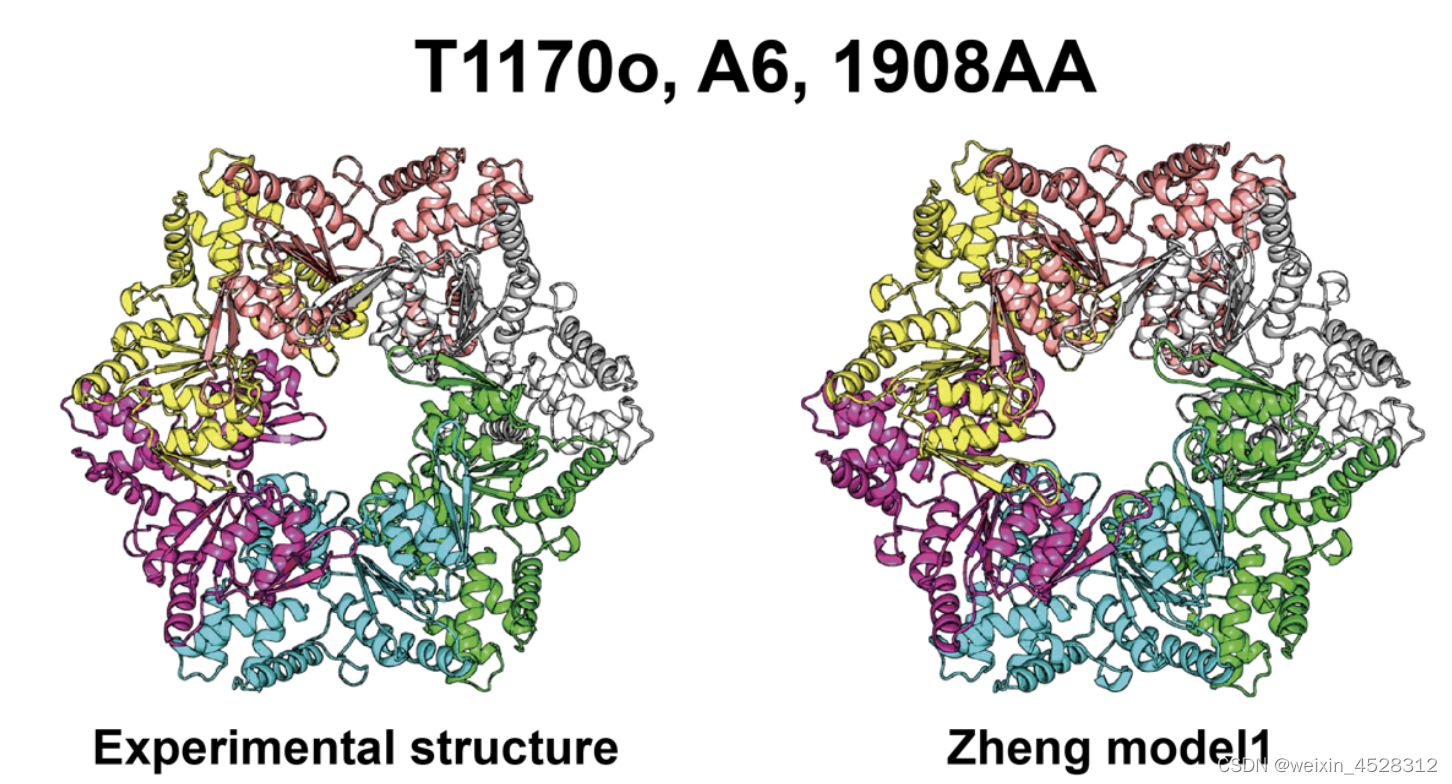
图4.蛋白质六聚体复合物示意图。每种颜色表示一个蛋白质单体,左侧为实验结构,右侧为密西根大学张阳实验室郑伟博士算法预测出的模型结构。
鉴于蛋白质-蛋白质复合体结构预测的重要性,2014年CASP11开始,组委会增加了该项比赛。今年CASP15蛋白质复合体预测,共计有86个参赛队伍(含25个服务器)参加了比赛。其中密西根大学张阳实验室郑伟博士开发的DMF-Multimer算法(参赛组名Zheng)在该项目比赛中拔得头筹(图5)。
其中值得注意的是,这是张阳教授实验室和郑伟博士首次参加该项目的比赛。组委会评测者对所有参赛组提交的蛋白质复合物的整体拓扑结构(Global Fold)及蛋白质-蛋白质接触面(Interface)预测精度进行了评估。DMF-Multimer在这两项指标上均大幅领先于其他参赛组。其中整体打分(35.4)高于第二名(29.9)、第三名(28.4)20%左右。其中第二名的Venclovas团队、第三名的Wallner均为该赛道的老牌强队,在历届CASP蛋白质复合体预测中均位于前列。值得注意的是,DMF-Multimer的打分(35.4)高于标准的AlphaFold2-Multimer打分(12.3)接近3倍。

图5. CASP15参赛算法在所有蛋白质复合体(Protein Complex/Multimer)上预测结果的排名(排名基于sum Z-score>0.0)。红色为张阳实验室郑伟博士开发的DMF-Multimer算法,该服务器的参赛名字为Zheng;黄色为标准版的AlphaFold2-Multimer算法。数据取自CASP15官方网站(https://predictioncenter.org/casp15/zscores_multimer.cgi)。
需要特别指出的是,在几个纳米抗体-抗原蛋白质复合体上,DMF-Multimer表现出了极高的预测精度(图6)。AlphaFold2-Multimer在这几个蛋白质复合体上预测结果的TM-score均小于0.7,而DMF-Multimer的预测结果的TM-score均大于0.9。TM-score表示预测结构与实验结构的差异,一般TM-score大于0.9说明预测的结果已经达到实验结构的误差精度。
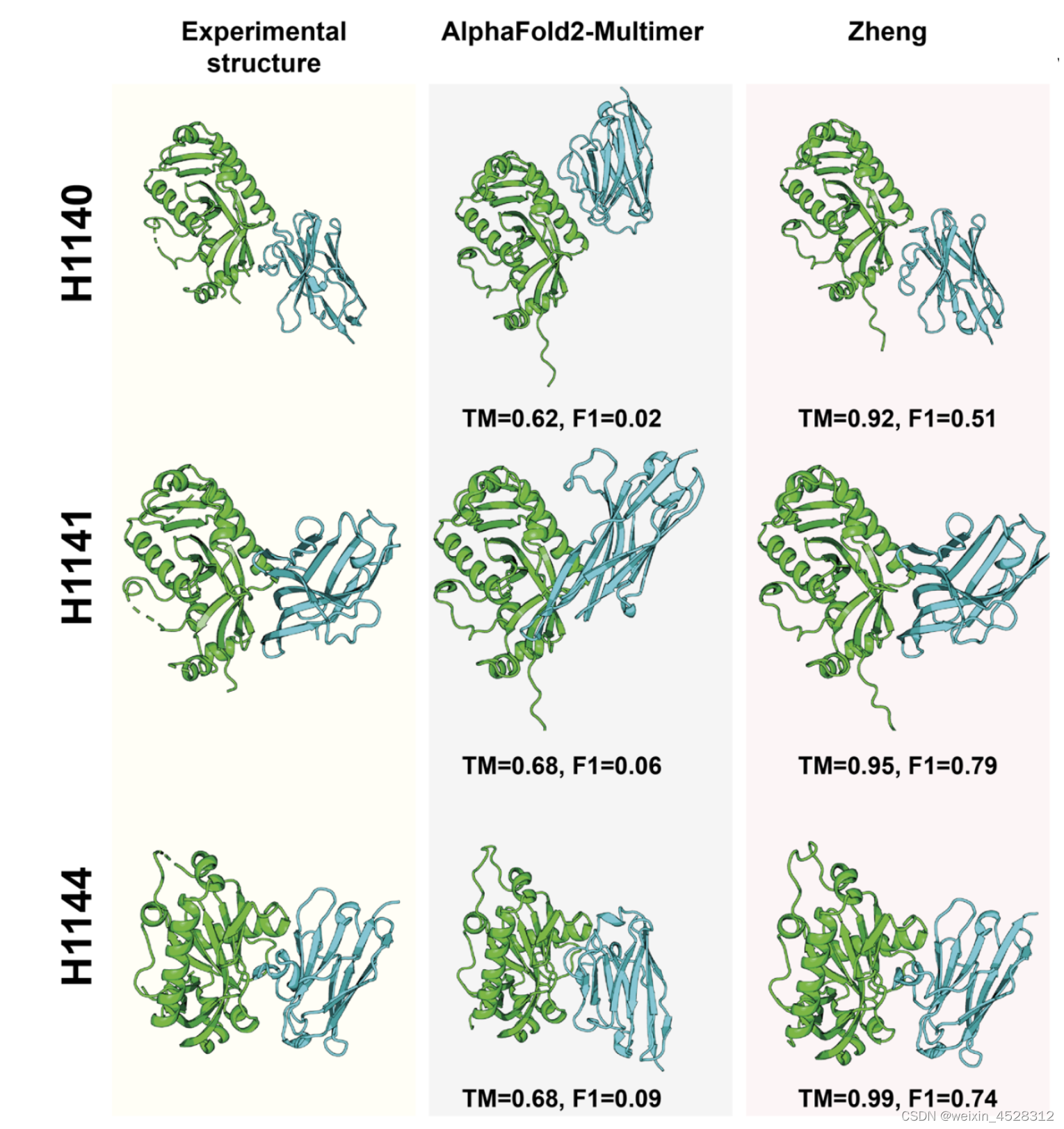
图6.CASP15中Zheng(DMF-Multimer)在纳米抗体与抗原复合物上的预测结果与AlphaFold2预测结果的对比。第一列为实验结构,第二列为AlphaFold2预测的结构,第三列为DMF-Multimer的预测结果。
总结及展望
对于今年CASP15张阳实验室郑伟博士的方法能够在单体、复合、以及多结构域蛋白上独占鳌头,郑伟博士在CASP15赛后,于土耳其举办的评议会议的邀请报告中给出了以下几方面的原因:
第一,利用D-I-TASSER平台将穿线模板和深度学习约束有机结合。张阳实验室李阳博士开发的AttentionPotential和DeepPotential等等深度学习算法精确预测了接触图,距离图,和氢键等信息,能够对AlphaFold2预测的距离进行很好的补充。为D-I-TASSER利用蒙特卡洛算法进行折叠起到了很大帮助。
第二,张阳实验室新开发的DeepMSA2,基于宏基因组数据库构建高质量的多序列比对(MSA),并且对多个MSA排序。这种高精度MSA的构建有助于机器学习更加精准的抽取蛋白质演化信息,从而对最后的蛋白质单体及复合体预测起到关键作用。
第三,张阳实验室郑伟博士和周晓根博士开发的结构域预测和组装算法对多结构域蛋白结构拆分组装。特别是,郑伟博士开发的基于深度学习接触图的结构域预测程序FUpred,可以有效的将序列进行结构域分割,以便在每个结构域上得到更精确的预测。
第四,对于复合蛋白的预测,DMF-Multimer提出了一种新的MSA配对连接方法,可以大大提高复合物MSA的质量,筛选有效的复合物演化信息,从而有效提高复合蛋白的结构预测。
尽管Google DeepMind的AlphaFold2在CASP14上表现出色。但今年的CASP15中,郑伟博士的D-I-TASSER以及DMF-Multimer等方法与标准版的AlphaFold2比均有显著提高,此外,很多其他组的参赛方法也优于AlphaFold2。由此看来,AlphaFold2并不是不能超越。另外,张阳实验室郑伟博士算法在三个不同赛道的领先结果也进一步显示,高精度蛋白质结构、多结构域蛋白质结构、蛋白质-蛋白质复合体结构,本质上遵从共同的原子作用关系,可能可以通过统一的计算方法得到。但是,要完全解决蛋白质结构预测的问题,特别是蛋白质四级结构预测、了解结构对功能的影响、了解变异对结构的影响、以及如何利用高精度结构进行药物分子设计等,仍然需要很多工作要做。
参考文献:
- CASP15蛋白质三级结构预测竞赛网站:https://predictioncenter.org/casp15/index.cgi
- D-I-TASSER在线服务器: https://zhanggroup.org/D-I-TASSER

























 122
122











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








