很多时候,在用中位数/均数、四分位数等特殊位置为截点时,生存分析往往没有差异。这个时候常常需要找到一个最佳截点使得生存分析有差异。常用的选取生存分析的最佳截断有很多种方法,如X-tile软件、R的CatPredi包、cutoff包。今天给大家介绍的是survminer 包的surv_cutpoint()函数。
载入示例数据
# 0. Load some data
library(survival)
library(survminer)
data(myeloma)
head(myeloma)> head(myeloma)
molecular_group chr1q21_status treatment event time CCND1
GSM50986 Cyclin D-1 3 copies TT2 0 69.24 9908.4
GSM50988 Cyclin D-2 2 copies TT2 0 66.43 16698.8
GSM50989 MMSET 2 copies TT2 0 66.50 294.5
GSM50990 MMSET 3 copies TT2 1 42.67 241.9
GSM50991 MAF <NA> TT2 0 65.00 472.6
GSM50992 Hyperdiploid 2 copies TT2 0 65.20 664.1
CRIM1 DEPDC1 IRF4 TP53 WHSC1
GSM50986 420.9 523.5 16156.5 10.0 261.9
GSM50988 52.0 21.1 16946.2 1056.9 363.8
GSM50989 617.9 192.9 8903.9 1762.8 10042.9
GSM50990 11.9 184.7 11894.7 946.8 4931.0
GSM50991 38.8 212.0 7563.1 361.4 165.0
GSM50992 16.9 341.6 16023.4 2096.3 569.2计算最佳截点
# 1. Determine the optimal cutpoint of variables
res.cut <- surv_cutpoint(myeloma, #数据集
time = "time", #生存状态
event = "event", #生存时间
variables = c("DEPDC1", "WHSC1", "CRIM1") #需要计算的数据列名
)
summary(res.cut) #查看数据最佳截断点及统计量> summary(res.cut)
cutpoint statistic
DEPDC1 279.8 4.275452
WHSC1 3205.6 3.361330
CRIM1 82.3 1.968317展示数据分布
# 2. Plot cutpoint for DEPDC1
# 以DEPDC1为例
plot(res.cut, "DEPDC1", palette = "npg")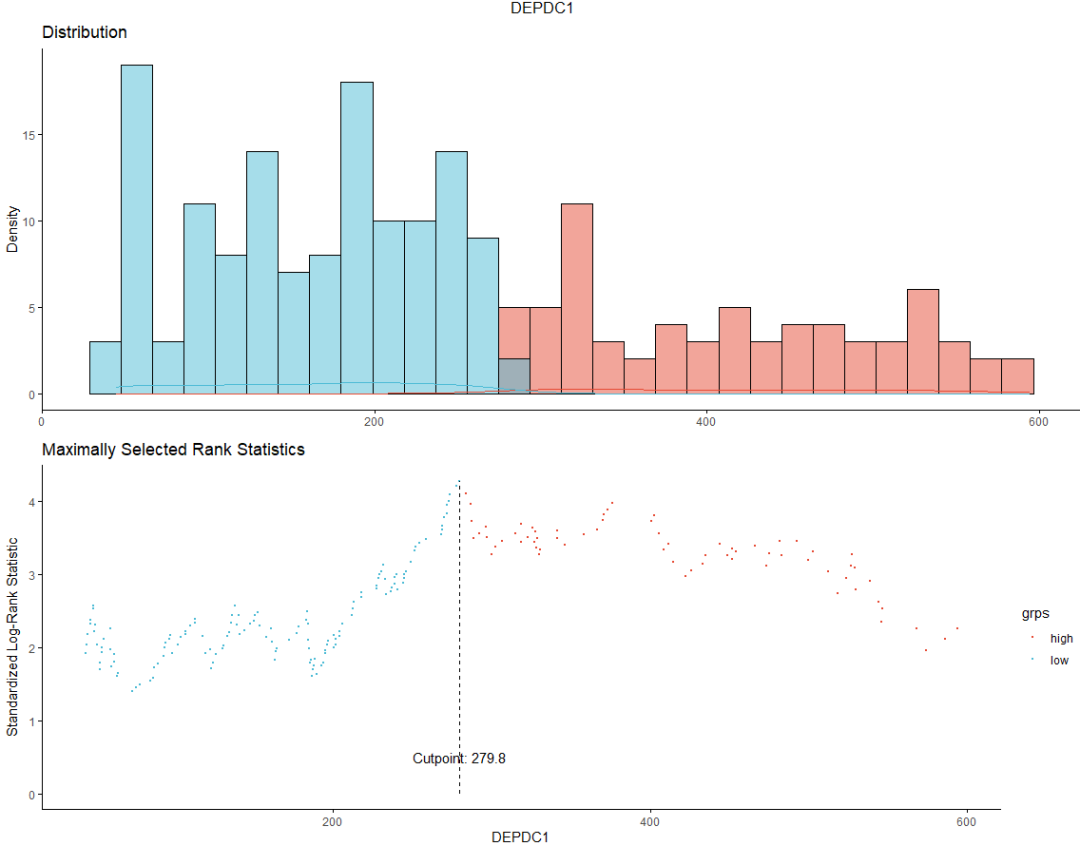
根据截点分类数据
# 3. Categorize variables
res.cat <- surv_categorize(res.cut)
head(res.cat)> head(res.cat)
time event DEPDC1 WHSC1 CRIM1
GSM50986 69.24 0 high low high
GSM50988 66.43 0 low low low
GSM50989 66.50 0 low high high
GSM50990 42.67 1 low high low
GSM50991 65.00 0 low low low
GSM50992 65.20 0 high low low绘制生存曲线
fit <- survfit(Surv(time, event) ~DEPDC1, data = res.cat)#拟合生存分析
#绘制生存曲线并显示P值
ggsurvplot(fit,
data = res.cat,
risk.table = TRUE,
pval = T)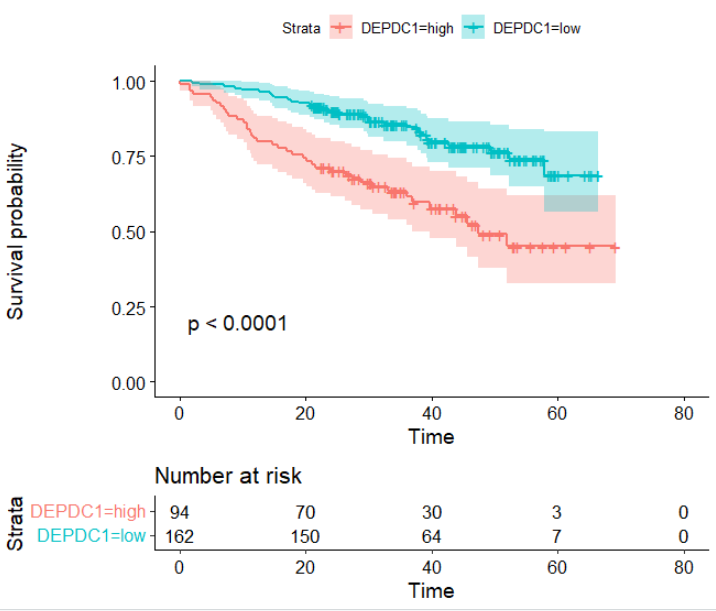
大功告成啦~
“Tips:这里只是用了简单的一个生存曲线图进行示范,如果大家需要更加复杂的生存曲线,只需要更改
ggsurvplot()函数的参数即可!关于生存分析本周末会在B站【木舟笔记】更新系列视频,需要的同学可以关注。
参考
surv_cutpoint: Determine the Optimal Cutpoint for Continuous Variables in survminer: Drawing Survival Curves using 'ggplot2' (rdrr.io)
“为爱发电不易~如果需要示例数据及代码(当然其实文中都已经写的很详细了)的同学,点赞&打赏任意金额,并在主页添加本人微信发送截图即可。






















 2138
2138











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








