Tips:对fMRI功能图像和结构图像预处理的可参考另外一篇博主的文章,很详细。点击跳转
但是对于我所要分析研究的fMRI数据没有结构像,所以那篇文章对于我来说也是一个学习和参考,也是摸索了好久才有点头绪,希望自己的一点经验能帮到大家。
一、准备工作
-
安装Dpabi。(注意在设置路径中选择【添加并包含子文件夹】,否则会出现无法正常启动Dpabi的情况)
-
在Matlab命令窗口输入【dpabi】启动,选择【DPARSF X.X】
-
然后选择【DPARSF Basic Edition】
这个能对功能图像进行处理,不需要结构图像。据说Advanced这个也能处理无结构图像的fMRI数据,但是我不会…
-
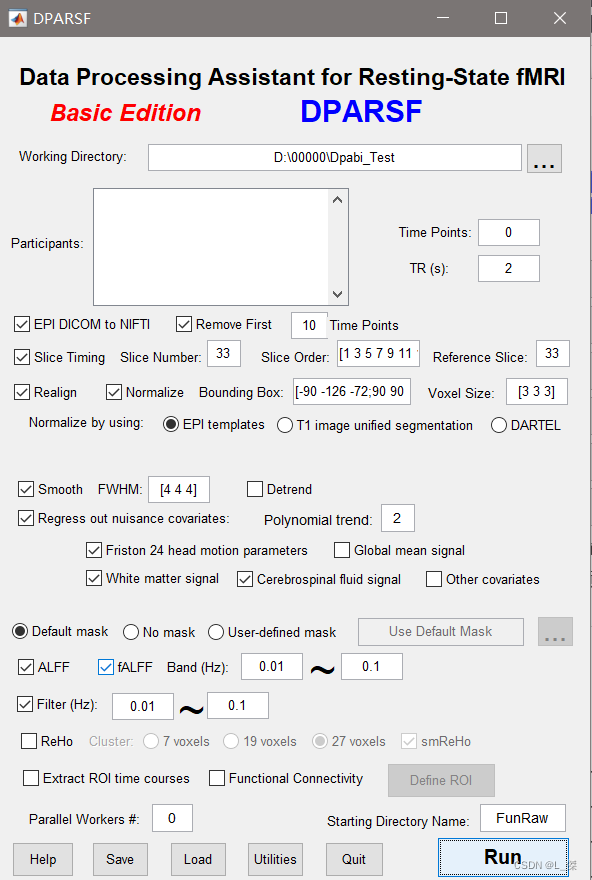
进入界面
-
数据集准备。可以从官网找数据练手,也能用自己的数据。(注意将每个被试放在FunImg文件夹中)

二、勾选预处理项目
- 选择FunImg上一级目录,也就是Test2

- 输入数据的Time Points 和 TR
Time Point可以用MRIcron软件来查看
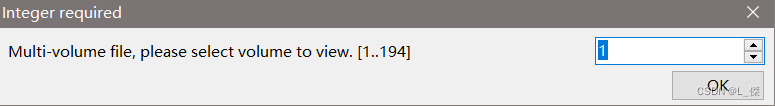
- 判断自己的fMRI数据是DICOM格式还是NII格式
DICOM格式则勾上,NII格式则不勾。

- 更改文件名。改成装被试文件的文件名,在本文的数据集中就是FunImg。
做完上述四步后【回车】,会自动读取出FunImg文件夹下的被试信息。没有出现的好好检查一下自己的数据集结构,对照上述步骤看看哪里出错了。

然后就是一般的预处理步骤了
-
去除前10个时间点

-
时间层矫正

这里要注意一下,每个fMRI数据的层数是不一样的。大家可以用MRIcron看一下。
例如本文的nii文件:
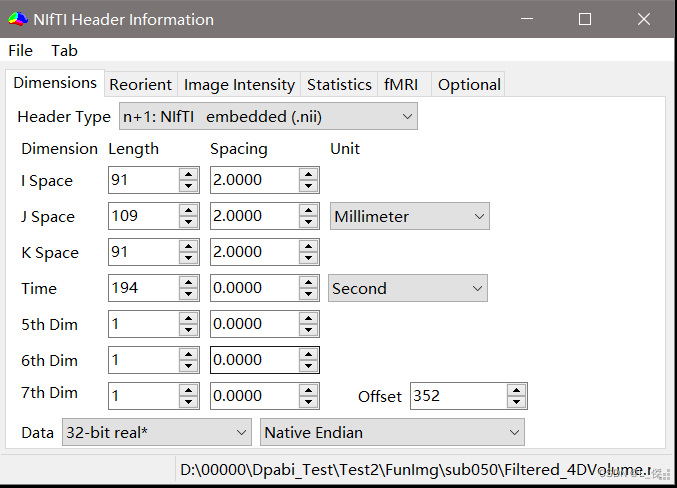
图中的【K Space】对应的数字就是层数
所以,本文的nii文件是91层。设置参数应该为:
Slice Numer:91
Slice Order:[1:2:91,2:2:90]
Reference Slice:91
【如果还不清楚,可以参考文章开头提到的那位博主的说明,更清晰一些。】 -
头动矫正、归一标准化、滤波等等。后面基本上不需要再点什么了。
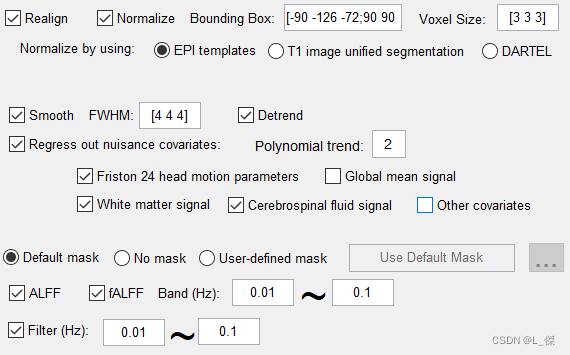
需要注意的是Normalize by using中,因为没有结构像,所以要选择EPI templates,不要选错了。 -
功能连接,提取ROI。需要点击【Define ROI】,选择需要的模板。
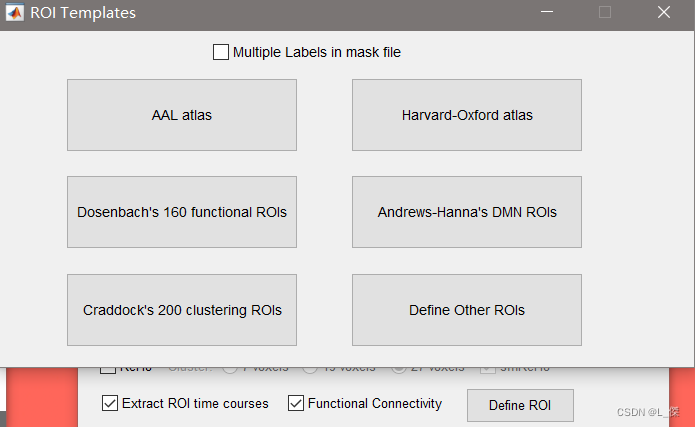
-
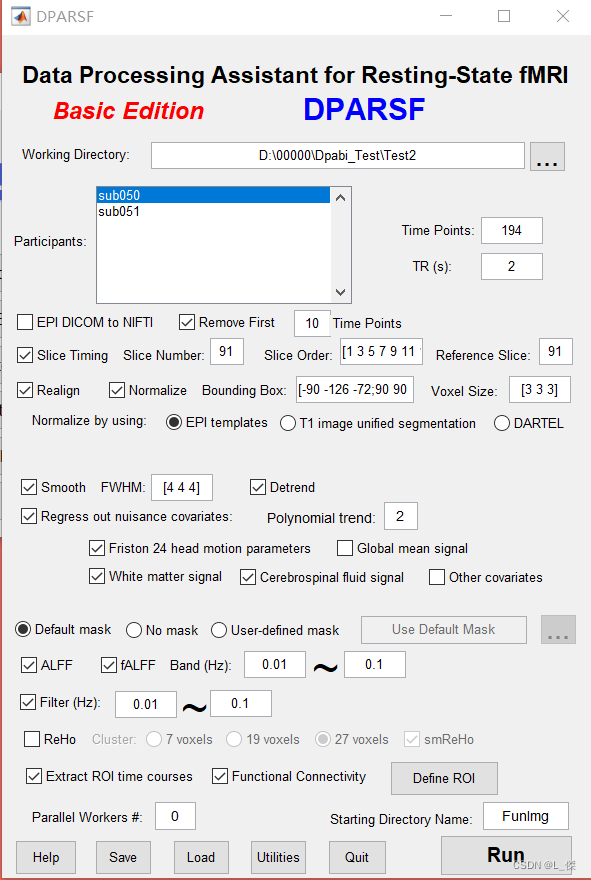
基本操作结束了,点击【RUN】。
三、查看结果
回到Matlab中命令行窗口查看运行结果。
如果一切顺利的话,文件目录应该会是这个样子:
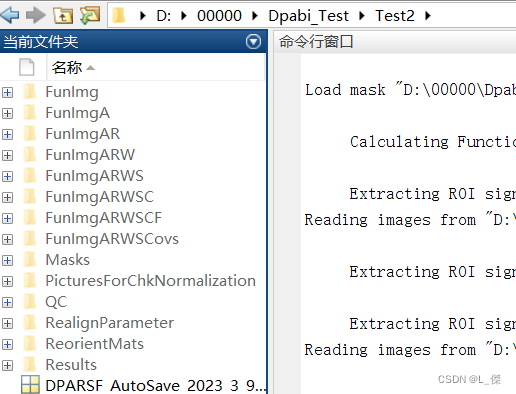
出现以下文件后缀的文件夹,说明你操作成功了。

最后,希望大家一次成功!






















 4万+
4万+











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








