分析rs-fMRI数据之前进行预处理是第一步,直接上验证过的非常详细的DPABI预处理步骤如下。
1.理论知识——通用的fMRI数据预处理
(1) 去除开始的几个时间点
扫描仪启动后,需要初始化,初始化时的磁场是不稳定的,为了去除不稳定的磁场对数据造成的影响,一般需要去除原始fMRI数据的前10秒的数据,如果TR是2秒,则是去除前5个时间点。就是为了去除初始数据点的影响。
(2) 对数据进行时间层矫正
时间层矫正,其实是相对参考层来说的,为了使得数据在每个时间点上与参考层做对齐处理。
采集数据时,机器多是一层一层间隔扫描的,不同层获取存在时间差,时间层校正就是把数据调整成看起来像是在同一个时刻完成了整个扫描。
如果隔层扫描,建议先做时间层校正,后做头动校正;如果连续扫描,建议先做头动校正,后做时间层校正;头动较小的被试先时间层校正后头动校正,头动明显的被试先头动校正后时间层校正。
(3) 头动矫正
在漫长的采集被试数据的过程中,头动是不可避免的,头动矫正就是尽量减小头动对数据造成的影响,剔除头动过大的数据。头动校正是为了对齐各个时间点的图像,一定程度上消除头动的影响,因为头动会造成体素的错位。
最常见的方法是选择一张图像作为参考,通常是平均图像,然后将所有体素进行刚体变换与参考图像对齐,从而去除平动和转动。一般定义的运动阈值:平移运动参数<2mm,旋转运动参数<2°。
(4) 空间标准化
空间标准化,有的叫归一化。其实就是图像配准处理,即将校正后的个体影像数据映射到标准的脑模板空间,并对图像进行重采样。
首先将T1像配准到功能像得到与功能像对齐的T1_1,然后将T1_1进行分割并配准到MNI而得到与MNI对齐的T1_2。将T1_1分割后配准到T1_2的过程参数应用到功能像的滤波后的时间点图像即可得到每个个体在MNI空间的功能像。
在fMRI领域有2大标准坐标系,一是基于单个被试的Talairach空间,一是基于大量被试的MNI空间, Talairach和MNI空间可以通过矩阵相互转换。
(5) 空间平滑
空间平滑,就是为了降低一些噪声,进行三维空间的滤波处理,如基于高斯核的平滑处理,高斯核的设计直接影响平滑效果和时效。FWHM:半高全宽,设置好高斯核的尺寸就可以就行高斯平滑处理去噪。
(6)去基线飘移
磁共振设备在连续扫描的过程中,随着扫描时长的加长而出现机器升温的情况,会使得信号产生线性飘移。
(7)带通滤波
低于静息态RS-fMRI数据研究发现,其低频(0.01~0.08Hz)具有生理意义,当计算脑自发活动的指标时一般需要进行带通滤波而提取出该频段的信息。
(8)处理中的几个方法词汇
ReHo:
利用KCC系数做局部一致性分析,用来测量局部神经元活动的一致性的方法,其指标越高,代表局部神经元活动一致性就越高,反之则表明活动的无序性或者紊乱。其容易受到高斯平滑的影响,因此计算该指标需要在做高斯平滑之前。
ALFF:
低频振幅振荡,通过计算低频段0.01~0.08HZ 内的BOLD信号的功率谱的均方根,利用低频信号的能量来表示不同脑区神经元活动强弱。
fALFF:
就是将低频信号的能量除以整个频段的功率,从而避免ALFF 指标群存在的一些缺陷,可以降低指标对生理噪声(大于0.1HZ)的敏感度。
ICA:
独立成分分析,是一种基于个体数据驱动的分析方法,分析功能连接时,不需要借助经验提取种子ROI,只需要基于数据分析空间独立成分从而 反馈出脑网络内脑区间的功能连接。可以采用GIFT(Group ICA fMRI Toolbox)工具箱进行完成,需要设置好成分数。配合使用ICASSO工具箱以增加稳定性。
2.基于DPABI的fMRI预处理步骤
基于DPABI进行数据处理是逐步进行的过程,逐步进行可以查看处理过程是否成功和每一步的输出文件,且处理步骤有先后顺序,处理时间受电脑配置差异而不同,每一个单一步骤处理正常完成就会在窗口打印Congratulations, the running of DPARSFA is done!!! 😃。
如果某个处理步骤没有打印该语句,有可能是该步骤处理中间报错了(需要仔细查看命令行窗口,并检查报错原因),也有可能系统仍然正在计算中,需要耐心等待。
基于DPABI的rs-fMRI预处理步骤有如下几步:
(1) 将DICOM转换为nii
(2) 去时间点和时间层矫正
(3) 头动矫正
(4) T1结构像分割
(5) 无关变量回归(相当于去噪)
(6) 空间标准化
(7) 空间平滑(如果后面要生成ReHo图的话,这一步就不做,需要生成ReHo图后再做平滑!!!)
(8) 去基线飘移
(9) 带通滤波(如果后面要做ALFF图和fALFF图,这一步不做!!!)
》》将DICOM转换为nii
为了处理过程搬运方便,如果原始数据是DICOM,需要将其转换为nii。

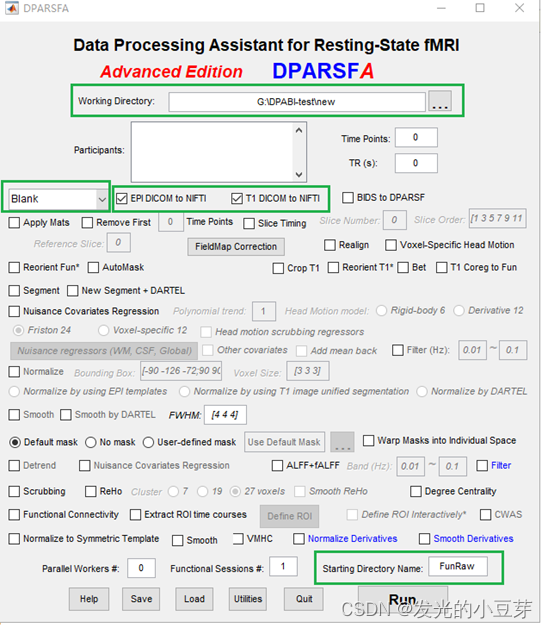


》》去时间点和时间层矫正
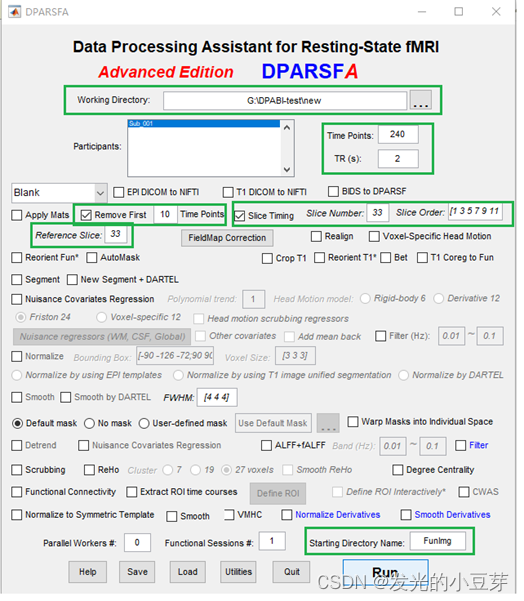


》》 头动矫正



》》T1结构像分割
这步处理时间有点长,需要足够的耐心~
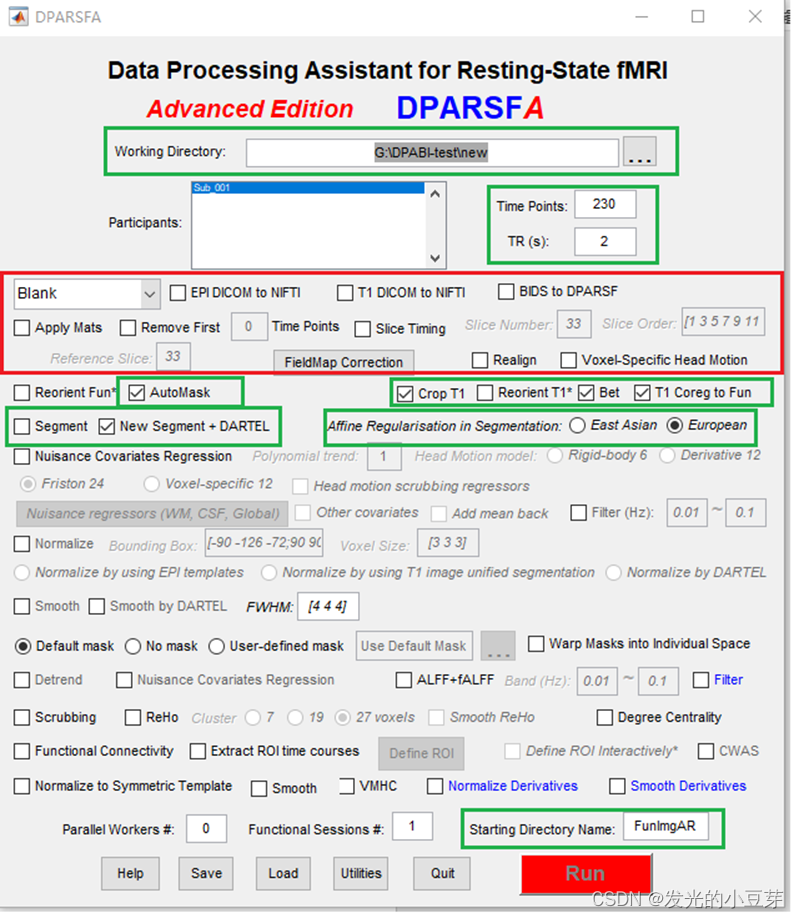


》》 无关变量回归(相当于去噪)
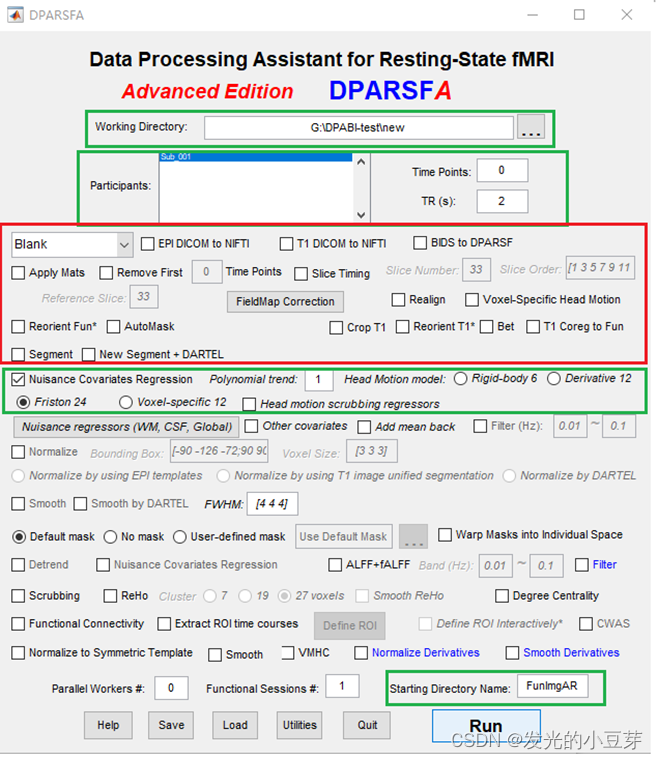


》》 空间标准化
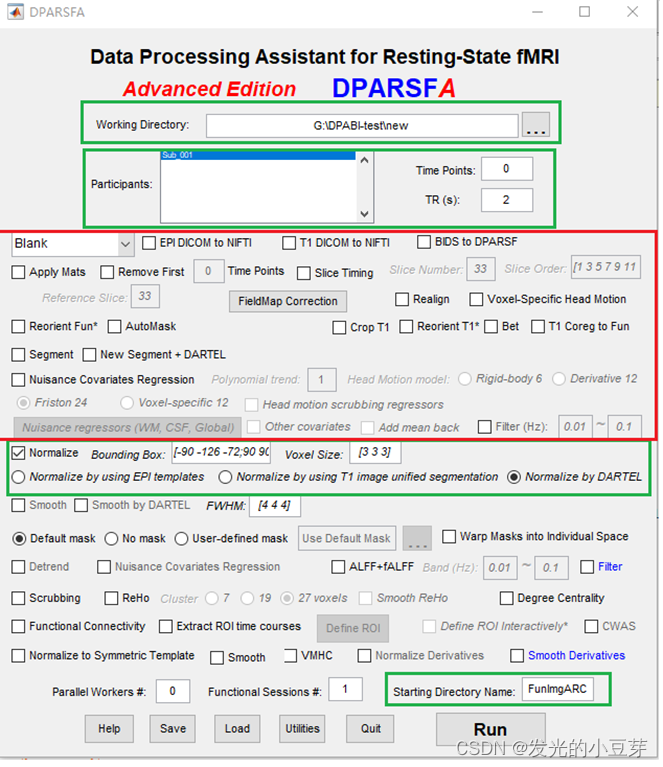


》》 空间平滑
如果后面要生成ReHo图的话,这一步就不做,需要生成ReHo图后再做平滑!!!


》》去基线漂移


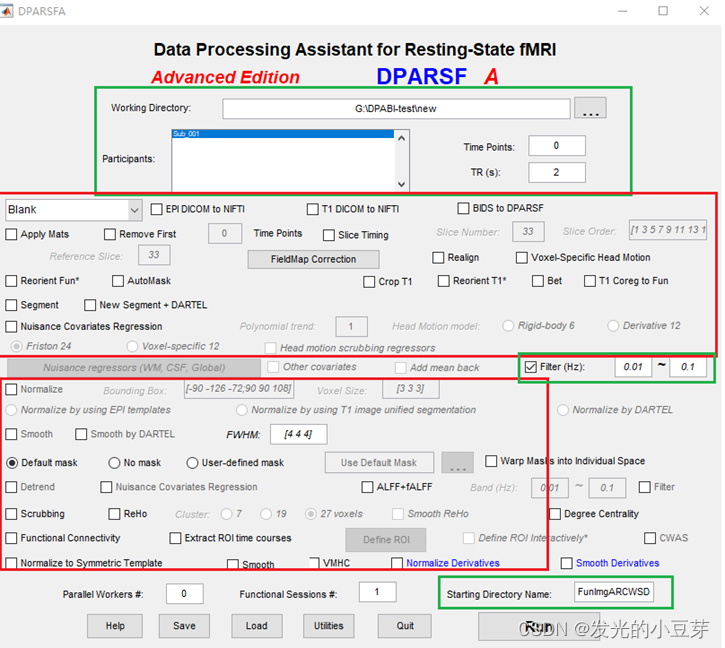
》》滤波
一般滤波范围0.01~0.1,但是设置范围为0.01-0.08能保留更多数据可使用。
如果后面要做ALFF图和fALFF图,这一步不做!!!


每一步都成功输出,还查看了相关输出文件的内部数据,rs-fMRI 预处理结束了,步骤之间能不能调换次序和省略,后面研究。预处理完成就可以着手计算ALFF、fALFF、ReHo等,还有FC分析,后面继续。。。

























 5万+
5万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








