生信碱移
单细胞lncRNAs
利用 ELATUS 通过 scRNA-seq 发现功能性 lncRNA。
来自西班牙纳瓦拉大学的研究者开发了一种名为 ELATUS 的专用工作流程,于2024年 11 月 9 日发表于 Nature Communications [IF: 14.7],旨在使用标准10x 单细胞RNA测序 (scRNA-seq) 检测到的更多的功能性lncRNA。
▲ DOI:10.1038/s41467-024-54005-7
-
标题: Uncovering functional lncRNAs by scRNA-seq with ELATUS
本质上来讲,这篇文章更多的是探索,而不是开发。对于想做方法开发的老铁,还是值得仔细一读的。当然,在应用层面,想在单细胞角度做更多的分析的老铁,比如添加一些 lncRNAs 的关联分析与挖掘,这个方法也是十分推荐。原理上讲,ELATUS 可以说在上游就更好的将read信息利用了起来,做到了一菜多吃。
单细胞与lncRNA
生物体的功能最终是由组成其组织的每个单个细胞的转录程序协调驱动的。深入了解细胞的转录配置可以揭示病理过程的内在机制。常规来说,基于二代测序的单细胞技术也能够对 lncRNA (长非编码RNA) 进行检测与定量。lncRNA 是 RNA 聚合酶 II 产生的转录本,长度超过 500 个核苷酸。但是,大多数单细胞水平的基因表达研究只关注蛋白编码基因, lncRNA 的研究很少。
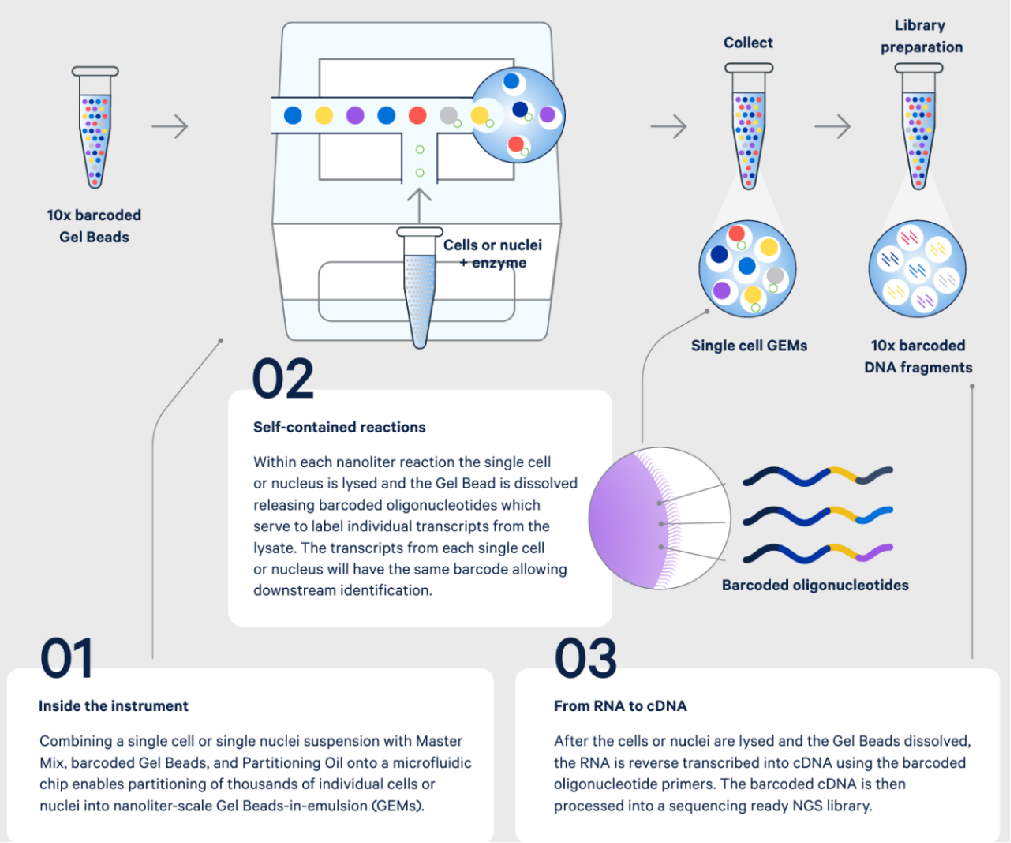
▲ 单细胞测序技术原理:结合微流控技术和二代测序,实现高通量单细胞转录组分析。通过将单细胞与条形码凝胶珠混合形成液滴(GEMs),在液滴内裂解细胞并用条形码标记mRNA,随后逆转录为cDNA并构建测序文库。
LncRNA的显著特点是其高度的组织和细胞类型特异性,其表达的改变还与多种病症有关。然而,它们的低表达和低注释准确性等局限性极大地阻碍了它们在单细胞研究中的应用。
比对方法影响lncRNA定量
scRNA-seq 分析流程始于对测序样本的预处理,这一关键步骤包括将含有测序 cDNA 的 reads 比对到参考基因组,同时校正细胞条形码和 UMI 条形码以识别单个 RNA 分子。
常规比对技术(如Cell Ranger)通过将测序数据逐一对比到参考基因组,找到精确位置并识别基因结构(如剪接点),因此精度高但计算时间长。伪比对技术(如Kallisto)则不进行这种逐一比对,而是将测序数据切成短片段(k-mer),快速与参考转录组中的片段匹配,通过匹配结果推测基因表达来源。

▲ 两种对齐方法的示意。
为此,本文作者首先综合比较了现有的scRNA-seq预处理管道,包括基于比对的方法(Cell Ranger和STARsolo)及伪比对方法(Kallisto-Bustools和Salmon-Alevin),评估它们对lncRNA检测和定量的影响。
作者发现,在蛋白质编码基因的检测以及细胞鉴定降维分布方面,几种方法并没有太大的差异:

▲ 几种方法在定量蛋白质基因以及细胞分群方面并没有太大差异。
但是,几种方法在 lncRNA 检测上表现出显著的差异。Kallisto 显著检测到更多的 lncRNA,包括高表达的 lncRNA,而其他方法未能量化这些基因。
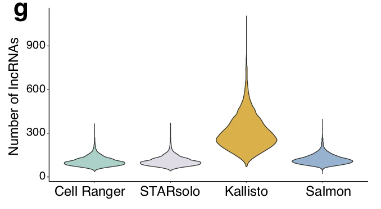
▲ Kallisto 显著检测到更多的 lncRNA。
scATAC-seq 验证 lncRNA 定量
作者认为,如果一个基因在 RNA-seq 中表达,那么 ATAC-seq 应该能在对应基因组位置检测到开放染色质信号。所以,通过对比 scATAC-seq 的开放染色质信号和 scRNA-seq 的基因表达数据,能够验证 Kallisto 和 Cell Ranger 各自定量的 lncRNA 是否有生物学依据。
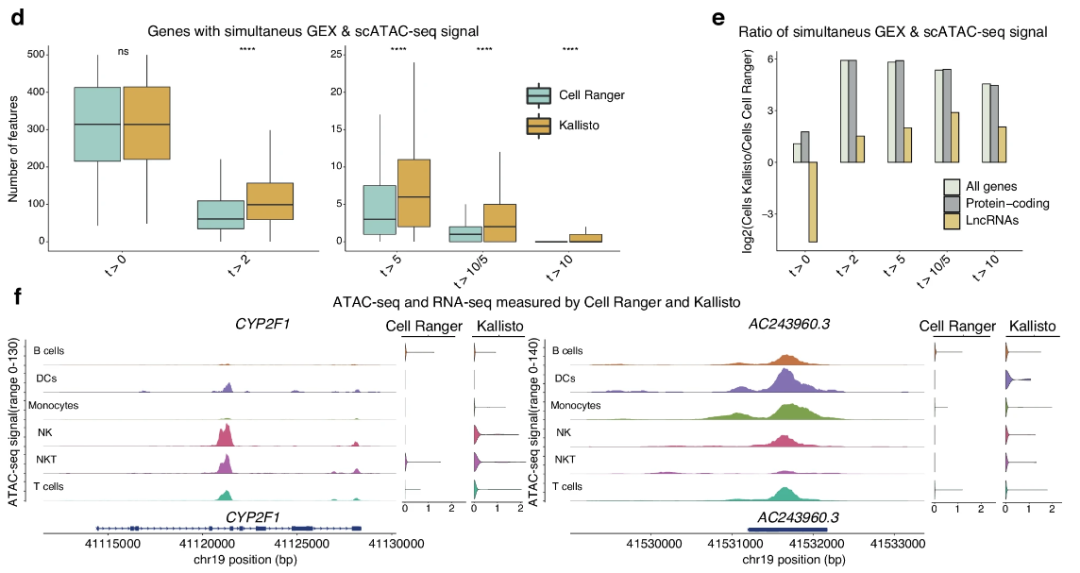
▲ 多数阈值下,Kallisto 定量的 lncRNA 表现出更高的同时激活比例。
作者发现,使用 Kallisto 处理的 scRNA-seq 数据,与 ATAC-seq 检测到的开放染色质信号更一致。多数阈值下,Kallisto 定量的 lncRNA 表现出更高的同时激活比例。比如,特定基因(蛋白质编码基因 CYP2F1 和 lncRNA AC242960.3)在不同细胞类型中,只有 Kallisto 的结果与 ATAC-seq 信号吻合。
ELATUS流程的开发
作者通过扩展测试验证了上面的结果,在多个公共数据集和不同注释标准中,Kallisto 始终检测到更多的 lncRNA。进一步分析表明,这种差异与lncRNA表达水平较低无关,而是源于预处理方法对注释不准确性的鲁棒性。
以上结果表明,Kallisto 对 lncRNAs 的额外检测,可以对数千个 lncRNAs 进行假设检验。在此基础上,作者开发了一个计算工作流程,用于阐明生物功能相关的 lncRNAs,也就是开头提到的 ELATUS 。简单来讲,他们认为生物功能相关的 lncRNAs 表达量应该超过一个阈值,而且应该具有高度特异性的细胞类型表达模式。应用这两种条件,则可以对检测到的 lncRNAs 进行过滤:
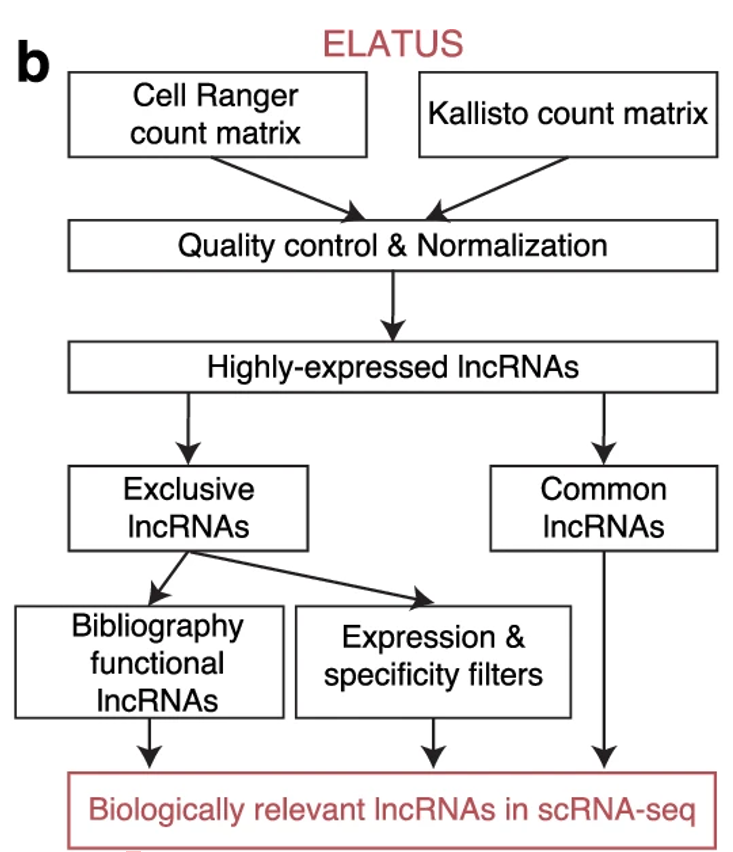
▲ ELATUS通过整合 Kallisto 和 Cell Ranger 的结果,结合严格的质量控制和筛选标准,筛选出高表达且具有细胞类型特异性的 lncRNA。它优先保留共同检测到的 lncRNA,同时根据 特异性指数(SI)和 表达倍数,从 Kallisto 独家检测的 lncRNA 中筛选功能性候选基因。
可能是作者根据经验想的点子
也可能只是想做个benchmark
反正这种方法不像是目的导向的
更像是结果导向
①发现方法对于lncRNA定量存在差异
②验证差异的可重复性与可行程度
③根据不足之处进行优化
小编是学习到了























 795
795

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








