https://www.sciencedirect.com/science/article/pii/S0006349523002369
Modeling software and model sharing platforms
Robust access to published models and their methods and data are crucial for reproducibility and reusability. Easy to use public platforms and community standards for documentation, testing, and minimal acceptable criteria can enhance this access. While some models may be initially generated using experimental code, if they gain more users in a public release, they should be required to meet reproducibility and usability standards. In many cases, experimental code has evolved into software that is intended for dissemination and use by the broader community.
SpringSaLaD
For example, SpringSaLaD is a software tool that explicitly models binding events and state changes while considering crowding effects (19).
https://vcell.org/webstart/SpringSaLaD/SpringSaLaDUsersGuideAndTutorial.pdf
https://www.cell.com/biophysj/fulltext/S0006-3495(15)04810-9
SpringSaLaD requires Java 8.0

Cellpack
CellPACK: Programs for Creating and Visualizing 3D Models of Cellular Environments
https://www.sciencedirect.com/science/article/pii/S002228362100588X?via%3DihubWelcome to cellPack’s documentation! — cellPack 1.0.8 documentationhttps://www.sciencedirect.com/science/article/pii/S002228362100588X?via%3Dihub
GALLERY – Center for Computational Structural Biology3D Whole Cell Model of a Mycoplasma Bacterium – Center for Computational Structural Biology
https://www.nature.com/articles/nmeth.3204
https://www.autopack.org/tutorials/nbcr-summer-training-2018
CellPACK
The CellPACK suite of programs are designed to create and visualize 3D models of cellular environments. This initiative builds on illustrative work from the Goodsell laboratory, and foundational methods were developed by Graham Johnson and Ludovic Autin during Johnson’s graduate work in the Olson laboratory.
CellPACK generates 3D models of cells based on a recipe of membranes, membrane-bound proteins, and soluble molecules. A tutorial for it is available at https://www.autopack.org/tutorials/nbcr-summer-training-2018. It is built on AutoPACK, a general tool for packing differently-shaped objects into a given space, which is available at http://autopack.org.
Setup
# create a virtual env: conda create -n autopack python=3.9 conda activate autopack pip install -e .[dev]Run pack code
# example pack v1 recipe : pack -r examples/recipes/v1/NM_Analysis_FigureB1.0.json -c examples/packing-configs/run.json # example pack v2 recipe : pack -r examples/recipes/v2/one_sphere.json -c examples/packing-configs/run.json # example pack from remote : pack -r github:recipes/NM_Analysis_FigureB1.0.json -c examples/packing-configs/run.jsonRun conversion code
To convert to simularium and view at Simularium
convert -r [FULL_PATH_TO_INPUT_RECIPE_FILE] -p [FULL_PATH_TO_PACKING_RESULT] -o [OUTPUT_PATH] ## Installation# Stable Release: pip install cellpack # Development Head: pip install git+https://github.com/mesoscope/cellpack.git
使用 simulariumio 可视化
simulariumio/examples/Tutorial_cellpack.ipynb at main · simularium/simulariumio · GitHub


CellVIEW
CellVIEW is a new tool that provides fast rendering of very large biological macromolecular scenes and is inspired by state-of-the-art computer graphics techniques. CellVIEW is implemented in a free-to-use game engine, unity3D. CellVIEW provides fast rendering by introducing new means to efficiently reduce the amount of processed geometries. CellVIEW is unique and has been specifically designed to match the ambitions of structural biologist to model and interactively visualize structures comprised of several billions atoms, such as this model of an entire HIV virion in blood plasma. CellVIEW was created in collaboration with Mathieu Le Muzic and Ivan Viola from the Vienna University of Technology. The software is free for download at http://www.autopack.org/home/cellview
MGLTools
Thew MGLTools software suite was developed in the Sanner lab at the Center for Computational Structural Biology (CCRB) formerly known as the Molecular Graphics Laboratory (MGL) of The Scripps Research Institute for visualization and analysis of molecular structures.
VISUALIZATION – Center for Computational Structural Biology
mgltools

Mesoscope
Mesoscope: A Web-based Tool for Mesoscale Data Integration and Curation
https://diglib.eg.org/items/3956a5e2-1fc3-46b6-9c8a-886a42f9a56c
Mesoscope@TSRI

unity3D
Unity是实时3D互动内容创作和运营平台。包括游戏开发、美术、建筑、汽车设计、影视在内的所有创作者,借助Unity将创意变成现实。Unity平台提供一整套完善的软件解决方案,可用于创作、运营和变现任何实时互动的2D和3D内容,支持平台包括手机、平板电脑、PC、游戏主机、增强现实和虚拟现实设备。
https://unity.cn/#%E6%A0%B8%E5%BF%83%E4%BA%A7%E5%93%81
Unity3d 教程 | Unity 中文课堂
https://zhuanlan.zhihu.com/p/540245002
https://zhuanlan.zhihu.com/p/128084962
CellPAINT
CellPAINT-2.0 Documentation – CellPAINT
https://drive.google.com/file/d/1oOkS2LAhEc6JSQ4nAjnca2SYqhoHVOAg/view
With CellPAINT, you can create dynamic illustrations of the inner molecular structure of viruses and living cells. CellPAINT pictures are based on experimental results from many different laboratories. The size and shape of each molecule is based on atomic structures determined by x-ray crystallography, NMR spectroscopy and electron microscopy, and key interactions, such as the embedding of proteins in membranes, are enforced as you create your pictures.

Mesoscale ExplorerBETA
YASARA - Pet World - sarscov2
Mol* Mesoscale Explorer — BETAMol* Mesoscale Explorer Documentation
Mol* Mesoscale Explorer
The advent of cryo-EM and cryo-ET, coupled with computational modeling, has enabled the creation of integrative 3D models of viruses, bacteria, and cellular organelles. Based on these models, the Mesoscale Explorer provides unprecedented access and insight into the molecular fabric of life, enhancing perception, streamlining exploration, and simplifying visualization of diverse data types, showcasing the intricate details with unparalleled clarity.



YASARA
YASARA - Yet Another Scientific Artificial Reality Application
YASARA is a molecular-graphics, -modeling and -simulation program for Windows, Linux, MacOS and Android developed since 1993, that finally makes it really easy to answer your questions. With an intuitive user interface, photorealistic graphics and support for virtual reality headsets, shutter glasses, autostereoscopic displays and input devices, YASARA creates a new level of interaction with the 'artificial reality', that allows you to focus on your goal and forget about the details of the program.
YASARA is powered by PVL (Portable Vector Language), a new development framework that provides performance way above traditional software. PVL allows you to visualize even the largest proteins and enables true interactive real-time simulations with highly accurate force fields on standard PCs, making use of GPUs if available (see benchmarks). You can push and pull molecules around and work with dynamic models instead of static pictures.
Being rooted in the academic world, all methods introduced by YASARA are described in peer-reviewed journals. You can obtain the initial stage YASARA View for free, while higher stages (YASARA Model, YASARA Dynamics, YASARA Structure) require a license fee that allows us to guarantee new developments, updates and support also in the future, independent of temporary grants. As a YASARA user, you are not limited to the role of a consumer. You can extend YASARA with macros, movies and plugins, or create gigastructures to share with the community.

whole-cell (WC)
DBS-pipes
DBS-pipes is database for raw high-throughput experimental data that facilitates and enhances the reproducibility of large-scale data analysis and modeling. DBS-pipes integrates metadata about samples, genome annotations, omics data, and data analysis pipelines.
Availability: In development
Author: Serrano Lab, CRG
Datanator
Datanator is a tool for discovering the data needed to build, calibrate, and validate whole-cell models. Datanator is composed of an integrated database of experimental data for whole-cell modeling and tools for identifying relevant data for modeling a specific organism and environmental condition. Datanator has a web-based graphical interface, a REST API, a Python API, and a command line interface.
Availability: Download
Author: Karr Lab, Mount Sinai
More info: Docs | License | Source | Test results , coverage
WC-KB-Gen
WC-KB-Gen is a Python framework for programmatically constructing knowledge bases for whole-cell models. WC-KB-Gen helps modelers retrieve data from external sources, organize this data, and record the provenance of this data. In turn, WC-KB-Gen helps modelers scalably, reproducibly, and comprehensibly discover and aggregate data for whole-cell models.
Author: Karr Lab, Mount Sinai
WC-KB
WC-KB is a data model for describing the experimental data needed to build, calibrate, and validate a whole-cell model. WC-KB includes a command line program and a Python API.
Availability: Download
Author: Karr Lab, Mount Sinai
More info: Docs | License | Source | Test results , coverage
Reference: Karr JR et al. WholeCellKB: model organism databases for whole-cell models. Nucleic Acid Res 2013.
WC-Model-Gen
WC-Model-Gen is a Python framework for programmatically constructing whole-cell models from large datasets described with WC-KB. WC-Model-Gen helps modelers retrieve data from a WC-KB and use the data to scalably design species and reactions. In turn, WC-Model-Gen helps modelers build models scalably, reproducibly, and comprehensibly.
Availability: Download
Author: Karr Lab, Mount Sinai
More info: Docs | License | Source | Test results , coverage
E-Cell
E-Cell P is a framework for automated bottom-up modeling and simulation of prokaryotes from their genomes. Starting from a genome sequence, the framework enables researchers to annotate genomes using knowledge collected from multiple databases, build agent-based models with single-nucleotide resolution, and generate genome-scale in silico omics data, all without detailed knowledge of mathematical modeling. E-Cell P also helps researchers evaluate the effect of mutations and synthetic genomes.
Author: Takahashi Lab, RIKEN
WC-Lang
WC-Lang is a data model and a file format for describing composite, multi-algorithmic whole-cell models. WC-Lang includes a command line interface and a Python API.
Availability: Download
Author: Karr Lab, Mount Sinai
More info: Docs | License | Source | Test results , coverage
WC-Rules
WC-Rules is a formalism for describing composite, mixed-grained, multi-algorithmic WC models. WC-Rules provides modelers a high-level, biologically-intuitive language for describing models in terms of patterns of metabolite, DNA, RNA, and protein species and rules for their interactions. Together, these species patterns and interaction rules enable modelers to describe the molecular complexity of any biochemical process including metabolism, replication, transcription, translation, and signaling. WC-Rules can also record the provenance of models including their data sources and assumptions.
Availability: Download
Author: Karr Lab, Mount Sinai
More info: Docs | License | Source | Test results , coverage
WC-Sim
WC-Sim is a simulator for composite, multi-algorithmic whole-cell models described with WC-Lang. WC-Sim is implemented using discrete event simulation. WC-Sim includes a command line interface and a Python API.
Availability: Download
Author: Karr Lab, Mount Sinai
More info: Docs | License | Source | Test results , coverage
Reference: Goldberg AP et al. Toward scalable whole-cell modeling of human cells. ACM Conf Prin Adv Discrete Simul 2016.
E-Cell
E-Cell, started in 1996, is an open-source software platform for modeling, simulation and analysis of complex, heterogeneous and multi-scale systems such as cells. The latest version, version 4, enables multi-algorithmic co-simulation of compartmental, mesoscopic, lattice, and particle submodels. E-cell supports high-performance computing, rule-based modeling, and integration with several bioinformatic resources.
Availability: Download
Author: Takahashi Lab, RIKEN
More info: Docs , License , Source
Scopyon
Scopyon is a bioimaging simulation software toolkit. Scopyon supports computational simulation of total internal reflection fluorescence microscopy (TIRF), laser-scanning confocal microscopy (LSCM), and super-resolution microscopy images. Combining optical microscopy with cell simulation, Scopyon generates digital images of cells that are quantitatively comparable with real experiments and that can be used to evaluate systematic effects in measurements.
Availability: Download
Author: Takahashi Lab, RIKEN
More info: License , Source
Reference: Watabe M et al, A Computational Framework for Bioimaging Simulation, PLoS One 2015
WC-Analysis
WC-Analysis is a Python framework for analyzing whole-cell knowledge bases, models, and simulation results. In particular, WC-Analysis helps models quickly and reproducibly conduct analyses for whole-cell modeling.
Availability: Download
Author: Karr Lab, Mount Sinai
More info: Docs | License | Source | Test results , coverage
WC-Viz
WC-Viz is an interactive web-based tool for visually analyzing the results of simulations of whole-cell models. WC-Viz enables modelers to design dashboards composed of time series plots and animated pathway diagrams of simulation results.
Use online: WholeCellViz.org
Author: Covert Lab, Stanford University
More info: License | Docs | Source
Reference: Lee R et al. WholeCellViz: data visualization for whole-cell models. BMC Bioinformatics 2013.
 WC-Test
WC-Test
WC-Test is a Python framework for programmatically verifying whole-cell models. WC-Test helps modelers verify that their models satisfy design constraints such as mass conservation, charge conservation, self replication and quickly find errors in their models. Furthermore, WC-Test can be integrated with continuous integration systems such as CircleCI. Together, WC-Test helps modelers collaborative develop high-quality models.
Author: Karr Lab, Mount Sinai
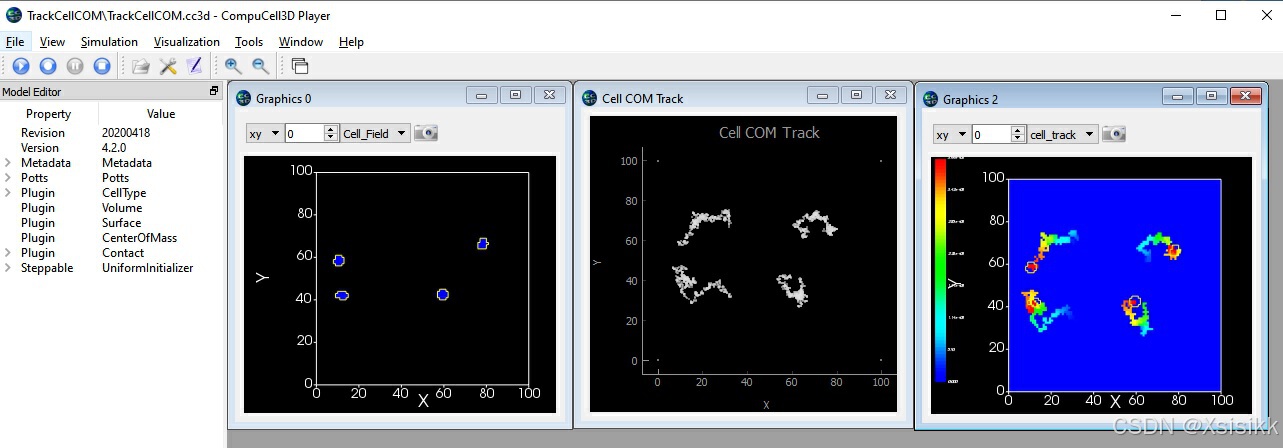
CompuCell3D
Cytosim
Cytosim is a cytoskeleton simulation suite designed to handle large systems of flexible filaments with associated proteins such as molecular motors (20), and MEDYAN software models cytoskeletons and their interactions with membranes in a multiscale/type manner, for example, by “iteratively switching between stochastic reaction-diffusion simulation and network mechanical equilibrations” (21,22).
Francois Nedelec / Cytosim · GitLab
https://github.com/simularium/Cytosimdoc/tutorials/tuto_introduction.md · master · Francois Nedelec / Cytosim · GitLabhttps://github.com/simularium/Cytosimhttps://e-learning.embl.de/installing-and-running-cytosim/2.%20Installing%20Cytosim.movhttps://github.com/simularium/Cytosim
Cytosim is an open-source cytoskeleton simulation suite designed to handle large systems of flexible filaments with associated proteins such as molecular motors. It is a versatile base that has been used to study actin and microtubule systems in 1D, 2D and 3D. It is built around a cross-platform C++ core engine running on UNIX, Mac OSX, GNU/Linux and Linux-environments within Windows. The code is modular and extensible, making Cytosim a convenient base that can be customized to meet particular tasks. Some of the most common tasks encountered during a simulation project are implemented in Python.
Cytosim has been used for about 100 research articles from the Nedelec and other groups.
Cytosim 是一个用于模拟细胞信号传导过程的计算工具或软件平台。它通常用于系统生物学和生物信息学领域,帮助研究人员理解和分析细胞内部复杂的信号网络和通路。以下是 Cytosim 可能具备的一些功能:
1. 建模:允许用户构建基于数学模型的细胞信号传导网络,这些模型可以包括各种生物分子(如蛋白质、酶、受体)和它们之间的相互作用。
2. 仿真:提供仿真引擎来模拟信号传导过程,包括信号的启动、传播、调节和终止。
3. 数据分析:分析和可视化仿真结果,帮助用户理解信号传导动态和网络行为。
4. 参数调整:允许用户调整模型参数,如反应速率、分子浓度等,以研究不同条件下的信号传导变化。
5. 实验验证:模型和仿真结果可以用于指导实验设计,或者与实验数据相结合,以验证模型的准确性。
6. 教育和研究:作为教学工具,帮助学生和研究人员学习细胞信号传导的基本原理和复杂性。
7. 药物发现:在药物开发过程中,用于预测药物对细胞信号传导网络的影响,从而加速药物靶点的发现和药物效果的评估。
请注意,Cytosim 可能是特定研究组或公司开发的专有软件,因此具体的功能和使用方式可能会有所不同。如果您需要更详细的信息,建议直接查看 Cytosim 的官方文档或联系开发者以获取准确的功能描述和使用指南。如果 Cytosim 是一个开源工具,您可能还可以在其官方网站或代码库(如 GitHub)中找到更多的资源和用户社区的支持。
To download the source code, enter these commands in a terminal window:
git clone https://gitlab.com/f-nedelec/cytosim cd cytosimTo compile using make, try:
makeIf this fails, parameters of
makefile.incneed to be updated manually. Altermatively, it is possible to use cmake to configuremakeautomatically:mkdir build cd build cmake .. make
Cytosim has a modular architecture and can be customized. The entities (fiber, motor, nucleator, etc.) are specified in derived C++ classes with usually few lines of code. These classes can be modified for custom applications.

The National Center for Multiscale Modeling of Biological Systems (MMBioS)
MCell
Cells are tightly packed with structures and molecules that carry out the day-to-day operations of living. Understanding how cellular design dictates function is essential to understanding life and disease, in the brain, heart, or elsewhere. MCell (Monte Carlo cell) is a program that uses spatially realistic 3-D cellular models and specialized Monte Carlo algorithms to simulate the movements and reactions of molecules within and between cells—cellular microphysiology.
CellBlender
GitHub - mcellteam/cellblender: Create, Simulate, Visualize, and Analyze Realistic 3D Cell Models
CellBlender
CellBlender is a Blender addon for creation, simulation, visualization, and analysis of realistic 3D Cell Models. CellBlender leverages the full-featured 3D content creation capabilities of Blender to support a rich environment for the creation of simulation-ready, biophysically realistic models of the microscopic structure and biochemical function of cells.
CellBlender is fully functional with MCell and partially functional with SBML (http://sbml.org). We invite the computational cell biology community to contribute to the project, adding features and support for their favorite simulation environments.
AlignTK
AlignTK is an image alignment toolkit. It is designed for batch-oriented alignment of a large number of 2-D images in either 2 or 3-dimensions. Although it has been applied most extensively to electron-microscopy (EM) images of neural tissue, the package can be used with arbitrary grayscale images. Multiple image formats are supported.
AlignTK currently only runs only on Linux platforms. If you would like to see a Windows-compatible release, please e-mail us to let us know of your interest in this.
In order to compile AlignTK, you will need to install the following packages (exact names may vary depending on particular Linux release):
- fftw3-devel
- libjpg-devel
- libtiff-devel
- libopenmpi-dev
- zlib-devel
Additionally, for the GUI-based programs, you will need X windows and:
- fltk1.1-devel
Once you have the prerequisites, the following installation commands should not produce errors:
- Download aligntk-1.0.tar.gz.
- $ tar zxf aligntk-1.0.tar.gz
- $ cd aligntk-1.0
- $ ./configure
- $ make
- [optional] $ make check
- $ sudo make install
This will install the tools into /usr/local/bin; to select a different target directory, use "./configure --prefix=/your_directory" and the binaries will be copied to /your_directory/bin.
If you don't wish to build the GUI tools (e.g., on the backend nodes of a cluster), then use "./configure --without-x".
You may specify a different MPI compiler by appending a "MPICC=your_mpi_compiler_name" to the end of the configure options. Similarly, the C and C++ compilers may be explicitly set with "CC=your_C_compiler_name" and "CXX=your_C++_compiler_name" options to configure. Compiler options may be also be set as configure options; for example, CFLAGS="-O3" or CXXFLAGS="-g".
If you would like to be notified of updates to the AlignTK package, please e-mail aligntk@psc.edu and ask to be placed on the aligntk-users mailing list. This list is low-volume and only used for AlignTK-related matters, and your e-mail address will not be distributed to other organizations.
ANM
ANM (Anisotropic network model) t is a simple tool for predicting the collective motions of molecular systems introduced using Elastic Network Models (ENM) and normal mode analysis. The biomolecular system is represented as a network, or graph, the nodes of which are the residues and the springs are their interactions. The model helps elucidate the intrinsic dynamics of proteins and their complexes and make inferences on functional mechanisms.

BioNetGen
Installation — BioNetGen documentation
Visual Studio Code - Code Editing. Redefined
GitHub - RuleWorld/BNGTutorial: Models that demonstrate the syntax and functionality of BioNetGen language and simulation tools.
BioNetGen is software for the specification and simulation of rule-based models of biochemical systems, including signal transduction, metabolic, and genetic regulatory networks.
BioNetGen has been used to generate models for reaction networks with hundreds of thousands of species and reactions. It is one of the few software tools available for generating physicochemical models of systems marked by combinatorial complexity - a hallmark of cellular signaling.


Software that Uses BioNetGen
This page links to other software that use BioNetGen code or BNGL-formatted models. Note: This list is a work in progress and may not be complete.
Software
| Software | Description | References |
|---|---|---|
| AMICI | Provides an interface to the CVODES and IDAS solvers for models specified in SBML or PySB, both of which are capable of representing BNGL models | |
| BioSimulators | Provides a registry of biosimulation tools supporting many different model formats, including BNGL | |
| BioUML | Java platform for -omics data analysis; a BNG plugin allows users to edit rule-based models as either BioUML diagrams or as BNGL text files | |
| Kappa | Another prominent rule-based modeling language. The syntax used to be very similiar to BNGL, but that is less true now. | Boutillier et al. |
| MCell | Uses 3D cellular models and Monte Carlo algorithms for simulating the movements and reactions of molecules within and between cells; the latest version, MCell4, uses BNGL syntax for rule-based modeling | |
| ML-Rules | Multi-level rule-based modeling formalism for modeling biochemical reaction networks | |
| MolClustPy | A Python package to characterize multivalent biomolecular clusters | Chattaraj et al. |
| NFsim | Biochemical reaction simulator designed to handle large numbers of possible molecular interactions or states; uses BNGL for model specification | |
| Parts & Pools | Employs BioNetGen to help design synthetic gene circuits | |
| PEtab | Data format for specifying systems biology parameter estimation problems using SBML | |
| Pleione | Provides methods for parameterizing models written in BNGL or kappa | |
| PyBioNetFit | Provides a suite of algorithms for parameterizing models specified in BNGL or SBML | |
| pyPESTO | Python toolbox for parameter estimation, including options to interface with AMICI and PEtab | |
| PySB | Allows for building models of biochemical systems as Python programs; internally translates Python code into BNGL or Kappa rules | |
| PyViPR | IPython widget for interactively visualizing systems biology models using PySB; supports BNGL and SBML models | |
| RuleDSD | Converts a DNA Strand Displacement (DSD) model into BNGL, then uses PySB to perform simulation and analysis | |
| Smoldyn | Performs cell-scale biochemical simulations; can perform rule-based modeling using either its native wildcards or by running BioNetGen on BNGL files | |
| SPATKIN | Uses an extension of BNGL to simluate biomolecular interactions on or at a 2D surface | |
| SRSim | Spatially simulates Brownian Dynamics of rule-based reaction systems (defined in BNGL) in 3D | |
| VDNA-Lab | Employs rule-based modeling (in BNGL format) to simulate DNA assembly systems | |
| VirtualCell | Cell biological systems modeling platform that uses a central database; BioNetGen@VCell can be used to perform rule-based modeling in the platform |
CellOrganizer
Cell Organizer: Building Models of Cell Structure from Microscope Images and Using them for High-Content Screening and Cell Simulations - MMBioS
https://mmbios.pitt.edu/images/tutorials/GLBIO2013/GLBIO-Tutorial-BNG-MCell-14May2013.pptx.pdf
3D Protoplast (Chloroplasts) - CellOrganizer
The CellOrganizer project provides tools for
- learning generative models of cell organization directly from images
- storing and retrieving those models in XML files
- synthesizing cell images (or other representations) from one or more models
Model learning captures variation among cells in a collection of images. Images used for model learning and instances synthesized from models can be two- or three-dimensional static images or movies.

GAMELAN
Generative Models of Conformational Dynamics - PMC GitHub - jiohyoo/gamelanpy: Python implementation of GAMELAN (GrAphical Models of Energy LANscapes) algorithm
GamelanPy is a Python implementation of the GAMELAN (GrAphical Models of Energy LANdscapes) algorithm. This algorithm is for learning a sparse Gaussian Mixture Model for structural fluctuations of a protein, but can also be used for other data where the original data can be assumed to follow a mixture of Gaussian distributions. GamelanPy supports options for sub-sampling methods for scalability and nonparanormal distributions for richer family of distributions than Gaussians. This package contains a pure python library and scripts for the command-line usages.
More information on GAMELAN can be found in this paper: Generative Models of Conformational Dynamics
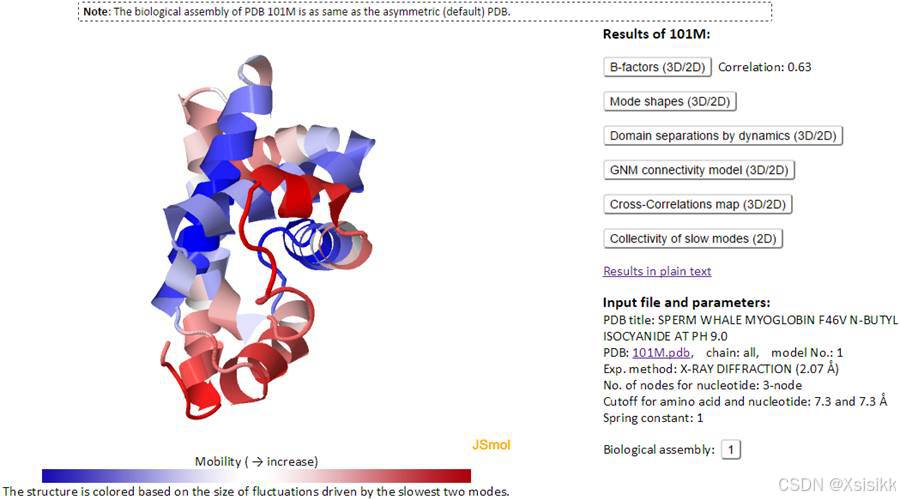
iGNM
Gaussian network model (GNM) is a simple yet powerful model for investigating the dynamics of proteins and their complexes. GNM analysis became a broadly used method for assessing the conformational dynamics of biomolecular structures with the development of a user-friendly interface and database, iGNM, in 2005. We present here an updated version, iGNM 2.0 iGNM - Online service for Gaussian Network Model, which covers more than 95% of the structures currently available in the Protein Data Bank (PDB). Advanced search and visualization capabilities, both 2D and 3D, permit users to retrieve information on inter-residue and inter-domain cross-correlations, cooperative modes of motion, the location of hinge sites and energy localization spots. The ability of iGNM 2.0 to provide structural dynamics data on the large majority of PDB structures and, in particular, on their biological assemblies makes it a useful resource for establishing the bridge between structure, dynamics and function.
GTKDynamo
https://sites.google.com/site/gtkdynamo/home
GTKDynamo is free/open source software which, together with pDynamo, transforms PyMOL into a powerful interface for molecular modeling. The interface has been designed to facilitate determining reaction pathways in biological systems, specially using hybrid QC/MM (or QM/MM) methods. Pymol has been chosen as a graphical interface to pDynamo because it has a python API with wide documentation available.
ProDy
ProDy — Protein Dynamics and Sequence Analysis
ProDy — Protein Dynamics and Sequence Analysis
http://www.bahargroup.org/prody/tutorials/membrane_anm/membrane_anm.pdf
ProDy is a free and open-source Python package for protein structural dynamics and sequence evolution analysis. It is designed as a flexible and responsive API suitable for interactive usage and application development. NMWiz, a VMD plugin GUI, also accompanies ProDy for streamlining protein dynamics analysis calculations and enabling comparative visual analysis of experimental and theoretical data.
With ProDy, you can perform principal component analysis of heterogeneous X-ray structures, NMR models, and MD snapshots. Protein dynamics can be modeled using normal mode analysis of anisotropic network model with optional distance and property dependent force constants. Powerful and customizable atom selections allow for contact identification and matching, superposing, and comparing multiple structures/chains. Newest additions to ProDy include fast and flexible features for analysis of sequence evolution and its comparison to protein functional dynamics.

Rhapsody
Rhapsody is a web tool for pathogenicity prediction of human missense variants based on sequence, structure and dynamics of proteins.

SWiFT-IR
SWiFT-IR uses Signal Whitening Fourier Transform Image Registration technique to achieve high precision image matching which is very robust to typical image distortions and defects. This high quality image matching in turn allows a hierarchical model based approach for the deformable registration of very deep image serial-section electron microscopy datasets. SWiFT-IR is also useful with other forms of grayscale and color imagery.
WE
The "weighted-ensemblizer" is an automated tool for setting up WESTPA-based weighted-ensemble (WE) simulations for existing MCell models. Because MCell models can be complex and expensive to simulate, WE can provide enhanced sampling for targeted observables of interest, such as the concentration of a certain species in a specified location, by using a strategy of replicating and pruning trajectories. The alpha-version webserver was developed by Rory Donovan, a student in the Zuckerman research group supported by the MMBioS resource.
For more information, see: Donovan RM, Tapia JJ, Sullivan DP, Faeder JR, Murphy RF, Dittrich M, Zuckerman DM. (2016) Unbiased Rare Event Sampling in Spatial Stochastic Systems Biology Models Using a Weighted Ensemble of Trajectories. PLoS Comput Biol 12(2): e1004611. doi: 10.1371/journal.pcbi.1004611
WESTPA
WESTPA (the Weighted Ensemble Simulation Toolkit with Parallelization and Analysis) is an open-source software package that provides a high-performance framework for carrying out extended-timescale simulations of rare events with rigorous kinetics using the weighted ensemble algorithm of Huber and Kim (1996). The software also includes options for further enhancing the sampling efficiency through reassignment of weights according to either equilibrium or nonequilibrium steady state, and a plugin for using a weighted ensemble-based string method. The software is designed to interface with any stochastic simulation engine, including but not limited to molecular dynamics (e.g. AMBER, GROMACS, and NAMD), Monte Carlo codes, BioNetGen, and MCell.
MEDYAN

MEDYAN(Microtubule and Actin Dynamics simulator for the cytoskeleton Network)是一个用于模拟细胞骨架动力学的计算工具。细胞骨架是由微管、肌动蛋白丝(微丝)、中间丝等蛋白质组成的复杂网络,它们在细胞形态、运动、分裂和信号传导中起着关键作用。MEDYAN 专注于模拟这些动态网络的行为。
MEDYAN 的主要特点包括:
1. 多尺度模拟:能够模拟从单个蛋白质到整个细胞骨架网络的多个尺度。
2. 动态模拟:可以模拟细胞骨架组件的动态组装和解聚过程。
3. 详细建模:提供了对细胞骨架组件和它们之间相互作用的详细描述。
4. 计算效率:优化的算法使得大规模模拟成为可能。
5. 灵活性:用户可以自定义模型参数,以适应不同的生物学系统和实验条件。
6. 可视化:提供了数据可视化工具,帮助用户理解模拟结果。
MEDYAN 通常用于研究细胞骨架的生物学功能,包括细胞形态的维持、细胞运动的机制、以及细胞如何响应外部信号。它也可以用于研究细胞骨架在疾病中的作用,例如癌症的转移和神经退行性疾病。
研究人员可以使用 MEDYAN 来构建模型,进行仿真实验,分析数据,并与实验结果进行比较,以验证模型的准确性和生物学意义。MEDYAN 的使用通常需要一定的计算生物学背景知识和对细胞骨架动力学的理解。
如果您对 MEDYAN 感兴趣,可以访问其官方网站或相关文献来获取更多信息,包括下载链接、用户手册和示例模型。
NERDSS
There are also software solutions for more general simulation needs; for example, NERDSS (23) and ReaDDY (24) provide relatively easy access to different reaction-diffusion systems.
NERDSS(Network Enhanced Regulatory DNA Sequence Simulator)是一个用于模拟基因调控网络和DNA序列的计算工具。它通常用于系统生物学和计算生物学领域,特别是在研究基因调控网络、转录因子结合位点、以及它们如何影响基因表达等方面。
NERDSS 的主要功能可包括:
1. 基因调控网络模拟:模拟基因、蛋白质、代谢物等生物分子之间的相互作用和调控关系。
2. DNA序列生成:生成包含特定调控元件(如转录因子结合位点)的DNA序列。
3. 动态模拟:模拟基因表达随时间的变化,以及基因调控网络对不同生物学条件的响应。
4. 参数化模型:允许用户定义和调整模型参数,如结合亲和力、反应速率等。
5. 数据集成:整合实验数据,如基因表达数据、蛋白质-DNA结合数据等,以构建和验证模型。
6. 可视化:提供网络和序列的可视化工具,帮助用户理解模型结构和动态行为。
7. 模型分析:分析基因调控网络的稳定性、鲁棒性和其他特性。
NERDSS 可以帮助研究人员理解基因调控的复杂性,揭示基因表达背后的调控机制,以及在疾病、发育和进化等生物学过程中的作用。此外,它还可以用于设计合成基因调控网络,为合成生物学和生物技术应用提供理论基础。
如果您需要更详细的信息或具体的使用指南,建议直接查看 NERDSS 的官方文档或联系开发者。如果 NERDSS 是一个开源工具,您可能还可以在其官方网站或代码库(如 GitHub)中找到更多的资源和用户社区的支持。
NERDSS(Network Enhanced Regulatory DNA Sequence Simulator)是一个合成生物学和系统生物学的工具,它专注于模拟基因调控网络和DNA序列。在合成生物学中,NERDSS 可以用于设计和预测基因回路的行为,这些基因回路是合成生物学中的关键组件。以下是一些具体的应用案例:
1. 基因回路设计:
使用 NERDSS 模拟不同的基因回路设计,如振荡器、逻辑门和传感器,以预测它们在实际生物系统中的行为。
2. 生物传感器开发:
设计能够响应特定环境信号(如化学物质、温度或pH变化)的生物传感器,并通过模拟验证其性能。
3. 细胞工厂构建:
在代谢工程中,NERDSS 可以帮助设计和优化基因表达调控,以提高目标化合物的生产效率。
4. 疾病模型构建:
构建合成基因网络来模拟疾病状态,用于研究疾病机理和测试潜在的治疗方法。
5. 合成生态学:
设计合成基因回路,以研究微生物群体之间的相互作用,这对于构建合成生态系统和生物修复等领域具有重要意义。
6. 基因治疗策略:
模拟基因调控网络的变化,以设计基因治疗策略,如通过合成基因回路来调控特定基因的表达,以治疗遗传性疾病。
7. 教育和培训:
在教学中使用 NERDSS 来帮助学生理解基因调控的复杂性,以及如何通过合成生物学方法来操纵这些网络。
8. 生物部件库构建:
利用 NERDSS 来模拟和验证合成生物学中使用的生物部件(如启动子、抑制子、RNA分子等)的功能。
9. 动态调控网络:
设计能够在不同条件下动态调控基因表达的网络,以响应复杂的生物学需求。
10. 生物安全和风险评估:
使用 NERDSS 来评估合成基因回路的潜在风险,确保合成生物学应用的安全性。
请注意,NERDSS 的具体应用案例可能会随着合成生物学领域的进展而不断发展。为了获取最新的应用案例和研究成果,建议关注合成生物学领域的最新文献和会议,或者直接查看 NERDSS 的官方文档和相关出版物。如果您对特定的应用案例感兴趣,可以提供更多的上下文信息,以便进行更详细的讨论。
ReaDDY
GitHub - readdy/readdy: Python / C++ based particle reaction-diffusion simulator
Welcome to the website of ReaDDy - a particle-based reaction-diffusion simulator, written in C++ with python bindings. ReaDDy is an open-source project, developed and maintained by Moritz Hoffmann, Christoph Fröhner and Frank Noé of the AI4Science (former CMB) group at the Freie Universität Berlin. This project continues the java software of the same name, by Johannes Schöneberg and Frank Noé.
## 安装
# optional: create environment for readdy, switch to that environment
conda create -n readdy python=3.9
conda activate readdy
# add conda-forge channel
conda config --env --add channels conda-forge
conda config --set channel_priority strict
# install readdy
conda install readdy
# Check if it worked, start a python interpreter and do
python3
>>> import readdyReaDDY(Reaction Diffusion in Diverse Dimensions)是一个用于模拟化学反应-扩散系统的计算工具,它特别适用于研究生物化学过程中的动态行为。这类系统在生物学、化学、物理和材料科学中非常常见,尤其是在细胞生物学和发育生物学中,许多重要过程都涉及到分子在空间中的扩散和反应。
ReaDDY 的主要特点包括:
1. 多尺度模拟:能够在不同的空间尺度上进行模拟,从单个分子到整个细胞或组织。
2. 多物理过程:模拟包括扩散、反应、绑定和解离等在内的多种物理和化学过程。
3. 空间分辨率:提供连续和离散的空间分辨率,以适应不同复杂性的系统。
4. 动态模拟:允许用户研究随时间变化的动态过程,如信号传导、基因表达和细胞迁移。
5. 参数化模型:用户可以自定义模型参数,如反应速率、分子浓度和空间结构。
6. 数据输出:提供多种数据输出选项,包括时间序列、空间分布图和动态模拟的可视化。
7. 灵活性:支持用户定义的化学反应网络,可以模拟复杂的生物化学过程。
8. 跨学科应用:适用于生物学、化学、物理学和工程学等多个学科领域。
ReaDDY 可以用于研究细胞内信号传导网络、细胞周期控制、胚胎发育中的形态发生、神经活动模型、生态学中的种群动态等。它是一个强大的工具,可以帮助科学家理解复杂系统中的动态行为,并预测系统对外部扰动的响应。
如果您需要更详细的信息或具体的使用指南,建议直接查看 ReaDDY 的官方文档或联系开发者。如果 ReaDDY 是一个开源工具,您可能还可以在其官方网站或代码库(如 GitHub)中找到更多的资源和用户社区的支持。
The Virtual Cell
VCell is an example of a more comprehensive web-based platform that enables users to build and share models of cell biological systems using the VCell database. It supports multiple simulation types, including deterministic, stochastic (SSA), and spatial stochastic (reaction-diffusion), and includes features for membrane flux, lateral membrane diffusion, and electrophysiology. VCell has a user-friendly interface for modelers, and geometries can be generated from analytical expressions or phenomenological inputs such as microscope images (25,26). In a more generalized manner, the challenge of model integration is also being addressed by tools such as Vivarium, allowing users to connect different types of models and interpreting between their data formats (27).
VCell- Modeling & Analysis Software – Virtual Cell Modeling & Analysis Software
https://vcell.org/webstart/VCell_Tutorials/VCell_Help/index.html
Virtual Cell (VCell) Modeling and Analysis Platform | SpringerLink
VCell支持从确定性(包括2D运动学支持的隔室常微分方程或反应-扩散-对流偏微分方程)到随机反应(SSA求解器)、空间随机(反应-扩散与Smoldyn)、混合确定性/随机以及无网络的基于代理的模拟。VCell还支持膜通量、侧膜扩散和电生理学,这些功能使其能够模拟细胞信号传导的多个方面。
The VCell BioModel consists of one Physiological Model that provides the conceptual representation of the model elements: structures, molecules, connectivity map, kinetics. It includes one or more Model Applications – analogous to “virtual experiments” that can be simulated: initial conditions, actual morphologies, electrical protocols, etc. An Application can be compartmental or it can use a Geometry. Each Application has a mathematical representation (Math Description) that is automatically generated and can be viewed in VCMDL format or through an equation browser. For each Application, a user can specify one or more Simulations (time length, resolution, solvers to use, parameter overrides, etc.) that will run and produce Simulation Results; these can be viewed in VCell, in a customized version of VisIt, or exported to the user’s computer in a variety of formats (raw data, spreadsheets, movies, images)
VCell(Virtual Cell)是一个综合性的计算平台,用于模拟细胞生物学系统中的物理化学和电生理过程。它由康涅狄格大学健康中心的细胞分析与建模国家资源开发,提供了自动化工具,用于在空间和时间上模拟各种细胞现象,既可以是确定性的也可以是随机性的。
VCell的功能包括:
1. 多尺度模拟:VCell能够模拟从分子间相互作用和运输到细胞膜电位动态的广泛细胞现象。这些机制可能是相互连接的,并且可以作为整体进行建模。
2. 空间和非空间模拟:VCell可以进行空间解析(确定性)系统模拟,包括膜上分子的侧向扩散与可逆解离到细胞质中的耦合,以及从实验图像生成几何结构的工具和将实验数据纳入VCell模拟的能力。
3. 图像基础几何结构:VCell允许用户使用来自实验图像的二维或三维几何结构,或者从理想化的解析表达式定义模型的几何形状。
4. 电生理学和反应动力学的耦合:VCell提供了将电生理学和反应动力学与运输机制(如扩散和定向运输)耦合的工具,并且可以将它们映射到各种形状的空间域上,包括从实验图像导出的不规则三维几何形状。
5. 确定性和随机性模拟:VCell提供了确定性和随机性(通过与Smoldyn软件包集成的最新添加到VCell的工具)模拟的能力。
6. 基于网络或图形表达的规则:VCell允许用户使用显式网络或图形表达的规则来建模,无论是对于没有建模经验的用户还是可以直接输入数学方程式的经验丰富的用户。
7. 远程服务器运行:VCell的服务器可以运行复杂的模拟,用户可以从任何低成本的笔记本电脑上进行操作。
8. 基于数据库的模型和模拟访问:VCell建立在中央数据库之上,并通过Web应用程序进行传播,用户可以从任何地方访问模型和模拟,并且可以与合作者共享模型或公开提供。
9. 新计算工具和建模能力:VCell最近开发并部署了新的强大的计算工具和建模能力,用于模拟空间解析(确定性)系统。
VCell是一个为从实验生物学家到理论生物物理学家等广泛科学家设计的建模和仿真工具,可以用于评估假设、解释实验数据或探究大型非线性系统的预测行为。VCell作为分布式应用程序部署,主要通过互联网使用,具有丰富的客户端Java界面和基于集群的服务器端,托管在康涅狄格大学细胞分析与建模中心。VCell的开发得到了国家医学科学研究所(NIGMS)的资助,并继续开发新技术以支持数学模型的细胞和系统生物学研究。
Example
https://vcell.org/vcell-news-eventsVCell News & Events – VCell- Modeling & Analysis Softwarehttps://vcell.org/vcell-news-events
A model of K+ and pH homeostasis in plant cells. – VCell- Modeling & Analysis Software


A VCell model of endocytic nanoclusters is published in Nature Comm – VCell- Modeling & Analysis SoftwareDynamin1 long- and short-tail isoforms exploit distinct recruitment and spatial patterns to form endocytic nanoclusters | Nature CommunicationsA VCell model of endocytic nanoclusters is published in Nature Comm – VCell- Modeling & Analysis Software2024年5月,澳大利亚和约翰霍普金斯大学的研究人员使用VCell设计和模拟了一个模型,解释了由Dynamin1长尾和短尾异构体的不同招募驱动的内吞纳米簇的形成。这表明VCell被用于研究细胞内吞作用的信号传导过程。
VCell 在模拟细胞信号传导时,确保模拟结果的准确性和可靠性主要通过以下几个方面:
1. 基于实验数据的模型构建:VCell 模型的构建通常基于实验数据,包括分子浓度、反应速率、蛋白质表达水平等。这些数据为模型提供了生物学上的真实性和可信度。
2. 详细的生物化学机制:VCell 允许用户详细地描述细胞内发生的生物化学反应和物理过程,包括酶促反应、信号转导途径、物质的运输和扩散等。这种详细的机制描述有助于提高模型的准确性。
3. 参数估计和敏感性分析:在模型构建过程中,需要对模型中的参数进行估计。VCell 提供了参数估计工具,可以通过实验数据来拟合模型参数。此外,敏感性分析可以帮助识别对模型输出影响最大的参数,从而确保模型的稳健性。
4. 模型验证:VCell 模型需要通过与实验数据的比较来进行验证。这包括模型预测的结果与实验观察的一致性检验,以及模型在不同条件下的预测能力。
5. 使用公认的数学和计算方法:VCell 使用公认的数学模型和计算方法来模拟细胞过程,如常微分方程(ODEs)、偏微分方程(PDEs)和随机模拟算法(如 Gillespie 算法)。这些方法在生物数学建模领域有着广泛的应用和验证。
6. 模型的可重复性和可共享性:VCell 支持模型的可重复性,用户可以共享和重新使用模型,这有助于其他研究者验证模型的准确性和可靠性。
7. 跨学科团队合作:VCell 模型的开发通常涉及生物学家、数学家、计算机科学家和工程师的合作,这种跨学科的合作有助于确保模型在生物学、数学和技术上都是准确和可靠的。
8. 持续的软件更新和技术支持:VCell 团队不断更新软件,修复已知的错误,改进算法,并提供技术支持,以确保模拟工具的准确性和可靠性。
9. 用户培训和文档:VCell 提供详细的用户手册、在线教程和培训课程,帮助用户正确使用软件,减少操作错误对模拟结果的影响。
通过上述措施,VCell 旨在提供一种准确和可靠的工具,以支持细胞信号传导的计算模拟。然而,任何模型都是对现实世界的简化,因此模型的准确性和可靠性也受到模型假设和简化程度的限制。因此,模型结果应与实验数据相结合,并在生物学上下文中进行解释。
BioSimulators
Vivarium is working on connecting to the Biosimulators database (28), which currently houses about 20 simulators that cover a broad range of simulations spanning multiple formats and algorithms. For example, these include BioNetGen, COBRA, COPASI, libRoadRunner, and Smoldyn.
BioSimulators is a free registry of biosimulation tools. The registry includes tools for a broad range of frameworks (e.g., logical, kinetic), simulation algorithms (e.g., FBA, SSA), and model formats (e.g., BNGL, CellML, NeuroML/LEMS, SBML, Smoldyn). Many of the simulation tools provide Python packages, command-line programs, and Docker images with consistent interfaces. Together, BioSimulators makes it easier to run simulations. BioSimulators is powered by several conventions including SED-ML
Vivarium 是一个开源的仿真平台,用于创建和运行复杂的仿真模型,特别是在社会科学、流行病学和生物统计学等领域。它被设计用来模拟个体(agents)在虚拟环境中的交互,这些个体可以代表人、动物、细胞或其他任何可以被建模为具有状态和行为的实体。
Vivarium 的主要特点包括:
1. 多主体仿真:能够模拟大量个体的复杂交互和行为。
2. 灵活的模型定义:用户可以自定义个体的行为规则、环境特性和仿真参数。
3. 数据驱动:可以整合真实世界的数据来驱动仿真模型,使其更加贴近实际情况。
4. 模块化架构:支持模块化设计,使得模型的不同部分可以独立开发和测试。
5. 可视化:提供工具来可视化仿真过程和结果,帮助用户理解模型行为。
6. 扩展性:设计用于处理大规模仿真,可以扩展到成千上万的个体和复杂的环境。
7. 跨学科应用:适用于多个学科领域,包括但不限于流行病学、经济学、社会学和生态学。
Vivarium 可以用于研究各种问题,如疾病传播、社会网络动态、经济系统行为、以及个体行为对群体动态的影响等。它提供了一个框架,允许研究人员构建和测试理论模型,以及进行政策干预的模拟实验。
如果您对 Vivarium 感兴趣,可以访问其官方网站或代码库(如 GitHub)来获取更多信息,包括下载链接、用户手册和示例模型。
BioNetGen
BioNetGen is software designed for modular, structure-based modeling of biochemical reaction networks. It can be applied to many other types of modeling as well. It provides a simple, graph-based syntax that lets users build reaction models out of structured objects that can bind and undergo modification.
COPASI
COPASI is a software application for simulation and analysis of biochemical networks and their dynamics.
COPASI is a stand-alone program that supports models in the SBML standard and can simulate their behavior using ODEs, SDEs, or Gillespie's stochastic simulation algorithm. Arbitrary discrete events can be included in such simulations. COPASI provides a set analysis methods and parameter estimation, a list of all features can be found here.
libRoadRunner
A high performance and portable simulation engine for systems and synthetic biology. It can run on many platforms including Windows, Mac OS, and Linux. libRoadRunner is major rewrite of the original C# roadRunner developed by Frank Bergmann and Herbert Sauro.
libRoadRunner comes with an extensive documentation including a significant C API, C++ API, Python bindings, ODE solver, structural analysis code, sensitivity, steady state analyses, event handling and stochastic simulation support. It includes significant improvements to performance, back-end design, by using LLVM to compile SBML models to machine code.
Smoldyn
Simulation projects are stored on the Biosimulations (29) site, which has online simulation deployment options.
Smoldyn is a computer program for cell-scale biochemical simulations. It simulates each molecule of interest individually to capture natural stochasticity and to yield nanometer-scale spatial resolution. It treats other molecules implicitly, enabling it to simulate hundreds of thousands of molecules over several minutes of real time. Simulated molecules diffuse, react, are confined by surfaces, and bind to membranes much as they would in a real biological system.
Smoldyn is easy to use and easy to install. It is more accurate and faster than other particle-based simulators. Smoldyn's features include: simulations in 1, 2, or 3 dimensions, a "virtual experimenter" who can manipulate or measure the simulated system, molecules with excluded volume, rule-based modeling, and Python bindings.

其他的
BioModels
BioModels Database是一个公开的、用于存储和交流数量型生物模型的数据库。它提供了一个平台,让研究人员可以分享他们的数学模型,并由更广泛的社区访问和重用这些模型。以下是BioModels的一些关键特点和功能:
1. 模型存储:BioModels存储了大量基于文献的机械模型,这些模型以标准格式(如SBML和CellML)编码,并在科学文献中发表过。这些模型涵盖了从分子到细胞、组织和整个生物体的不同生物学层次。
2. 模型验证:BioModels中的许多模型都经过了人工验证,以确保它们与原始出版物的一致性。这些模型的组件也与外部数据库资源和本体进行了交叉链接,以便于精确搜索。
3. 模型格式:BioModels支持多种模型格式,包括SBML、CellML、XPP、SciLab等,以及Python、Mathematica、Matlab SimBiology等建模软件生成的模型。
4. 搜索和检索:BioModels提供了强大的搜索系统,用户可以通过作者名、出版物、年份、模型名称或BioModel标识符等多种元数据参数进行搜索。
5. 协作管理:BioModels团队与模型作者合作,进行模型的管理和注释,以提高数据资源的可重复性和易访问性。
6. 数据链接:BioModels将其数据与其他公共数据资源链接,如Ensembl基因组浏览器和ChEMBL生物活性实体数据库,以及受控词汇表和本体,为每个模型提供了更广泛的生物学背景。
7. 模型共享和重用:BioModels鼓励研究人员共享和重用现有的模型,以构建新的模型或扩展现有模型,这对于科学研究和药物开发等领域非常有价值。
8. 编程访问:BioModels提供了Web服务,允许用户以编程方式访问和检索模型,这对于自动化工作流程和集成到其他生物信息学工具中非常有用。
9. 模型发布:在BioModels中,模型经过验证和注释后,会公开发布,供所有用户在Creative Commons CC0许可下自由使用、修改和分发。
BioModels Database由欧洲分子生物学实验室(EMBL-EBI)的BioModels.net团队维护,并且得到了包括EMBL和美国国家医学科学研究所在内的多个组织的资助。BioModels的建立是为了满足系统生物学社区的需求,它不仅是一个模型存储库,也是一个促进模型共享、重用和进一步研究的重要资源。
Geneformer
Transfer learning enables predictions in network biology | Nature
https://zhuanlan.zhihu.com/p/634446380
https://www.biorxiv.org/content/10.1101/2024.08.16.608180v1.full.pdf
Geneformer是一个基于Transformer模型的AI工具,它通过迁移学习从大规模单细胞转录组数据中学习基因网络的动态和相互作用。这个模型由来自麻省理工学院和哈佛大学博德研究所的研究人员及其合作者开发,它使用单细胞RNA表达数据中表达最高的基因来生成每个细胞的密集表示,这些表示可以用作各种下游预测任务的特征。
Geneformer的核心原理是利用自监督学习(self-supervised learning)技术,通过在大量数据上预训练来获得基因表达数据的丰富表示。在预训练阶段,Geneformer采用了一种蒙版语言建模技术,这种技术将部分基因表达数据蒙版,然后模型根据周围环境学习预测蒙版基因。这种方法不需要标记数据,而是使模型能够理解复杂的基因相互作用和调节机制。
Geneformer的架构支持在有限数据情况下的预测任务,其注意力机制使其能够专注于输入数据中最相关的部分,以便更好地捕捉关键信息。这种上下文感知方法允许模型通过考虑基因之间的关系和依赖性来进行预测。
在实际应用中,Geneformer已经被证明能够在数据有限的情况下准确预测基因行为和疾病机制,从而加速药物目标的发现,并提高对各种生物学背景下复杂基因网络的理解。例如,Geneformer能够使用仅5000个数据细胞重建心脏内皮细胞中的重要基因网络,这与之前使用超过30000个数据细胞进行训练时使用的先进方法一样准确。此外,它还可以在特定细胞类型分类任务中实现超过90%的准确度。
Geneformer模型和其训练数据集Genecorpus-30M都可以在Hugging Face上访问到。这使得研究人员可以利用这个强大的工具来解锁基因网络,即使在数据受限的情况下也能够进行有效的研究。
Escher
https://www.sciencedirect.com/science/article/pii/S0092867421014884?via%3Dihub
Escher 是一款用于可视化和分析代谢途径的软件,它允许用户构建、编辑和共享代谢网络。在研究最小细胞模型如 JCVI-syn3A 时,Escher 可以帮助研究者以直观的方式展示和分析细胞内的代谢过程。以下是使用 Escher 进行生物途径可视化和分析的一般步骤:
1. 构建代谢网络:
- 使用 Escher,研究者可以从头开始构建代谢网络,或者导入已有的代谢模型。
- 通过拖放反应和代谢物,构建出细胞内代谢途径的图示。
2. 导入数据:
- 将实验数据或模型数据导入 Escher,这可能包括基因表达数据、蛋白质丰度、代谢物浓度等。
3. 编辑和注释:
- 在 Escher 中,用户可以对代谢途径进行编辑,添加或删除反应,调整反应速率,或者注释特定的代谢物或酶。
- 可以为途径中的每个步骤添加详细的注释,包括 EC 号、基因名称、反应速率等。
4. 可视化调整:
- 根据需要调整可视化的布局、颜色和样式,以突出显示特定的代谢途径或反应。
- Escher 允许用户自定义图形和颜色编码,以表示不同的代谢状态或通量。
5. 分析和模拟:
- 使用 Escher 内置的分析工具来评估代谢网络的通量、瓶颈和调节点。
- 可以模拟不同的生物学条件,如基因敲除、环境变化等,以预测代谢网络的反应。
6. 共享和发布:
- Escher 允许用户将构建的代谢网络导出为多种格式,包括 JSON、PNG、PDF 等,方便在出版物中使用。
- 可以通过 Escher 的在线平台与合作者共享和讨论代谢网络。
7. 集成其他工具:
- Escher 可以与其他生物信息学工具和数据库集成,如 KEGG、MetaCyc、BRENDA 等,以获取更丰富的代谢信息。
在文章中提到的研究中,研究者可能使用 Escher 来可视化 JCVI-syn3A 的代谢网络,并分析其在不同条件下的行为,如在模拟最小细胞模型的代谢活动时。通过这种方式,Escher 帮助研究者理解细胞如何在基因组缩减的情况下维持基本的生命活动,并揭示了细胞生长和代谢之间的关键联系。

























 2454
2454

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








