基于XGBOOST预测肝硬化处于阶段
肝硬化是由多种肝脏疾病和病症引起的肝脏瘢痕化(纤维化)的晚期阶段,例如肝炎和慢性酗酒。每次肝脏受损时,它都会试图修复自身。在这个过程中,瘢痕组织形成。随着肝硬化的进展,越来越多的瘢痕组织形成,使肝脏难以正常工作(失代偿性肝硬化)。晚期肝硬化是危及生命的。
笔记本目标 | 摘要
我们创建了一些附加功能,并评估了不同方法和特征重要性在这个增强数据集上的性能。涵盖的方法有:
- ❌ LogisticRegression
- ❌ DecisionTreeClassifier
- ❌ RandomForestClassifier
- ❌ SVC
- ❌ KNN
- ✅ XGBClassifier
- ❓ CatBoostClassifier
- ✅ LGBMClassifier
- ❌ Keras Sequential NN
上述模型的参数化已包含在内,但尚未进行优化。
待办事项 📓
- 由于“我对
optuna不熟悉”,已删除了CatBoostClassifier,请检查优化后的模型是否能与其他方法竞争 - 当我在完整数据集上运行
XGBClassifier和LGBMClassifier时,我得到了与前20个最重要特征(Risk_Score、Diag_Month、Age_Years、Diagnosis_Date、Liver_Function_Index、SGOT、Albumin、Tryglicerides)相差8个特征的结果。在不同的数据特征上优化模型参数。函数已经能够处理不同的输入,但需要调整参数。 - 目前还没有实现降维方法
- 也没有指定最佳特征数
- 递归特征消除(RFE)算法可能很有意思,例如使用
sklearn.feature_selection.RFE
- 递归特征消除(RFE)算法可能很有意思,例如使用
数据集描述
数据集描述,取自原始数据源Cirrhosis Patient Survival Prediction,并添加了一些“常规见解”。
| 变量名 | 角色 | 类型 | 人口统计学 | 描述 | 单位 | 缺失值数量 |
|---|---|---|---|---|---|---|
ID | ID | 整数 | - | 唯一标识符 | - | 否 |
N_Days | 其他 | 整数 | - | 注册时间与1986年7月死亡、移植或研究分析时间之间的天数 | - | 否 |
Drug | 特征 | 分类 | - | 药物类型:D-青霉胺或安慰剂。药物类型可能会影响治疗的有效性,从而影响状态。 | - | 是 |
Age | 特征 | 整数 | 年龄 | 年龄。年龄可能与疾病进展有关;年龄较大的患者可能有不同的状态轨迹。 | 天 | 否 |
Sex | 特征 | 分类 | 性别 | 性别:M(男性)或F(女性)。生物性别可能会影响疾病模式和对治疗的反应,从而影响状态。 | - | 否 |
Ascites | 特征 | 分类 | - | 腹水的存在:N(否)或Y(是)。腹部积液是腹部积液的积聚,通常是晚期肝病的迹象,可能表明较差的状态。 | - | 是 |
Hepatomegaly | 特征 | 分类 | - | 肝肿大的存在:N(否)或Y(是)。肝脏肿大。如果存在,可能表明更严重的肝脏疾病和潜在的较差状态。 | - | 是 |
Spiders | 特征 | 分类 | - | 蜘蛛痣的存在:N(否)或Y(是)。蜘蛛痣是皮肤下可见的类似蜘蛛的小毛细血管,与肝病有关,可能表明更严重的疾病影响状态。 | - | 是 |
Edema | 特征 | 分类 | - | 水肿的存在:N(无水肿且无利尿剂治疗水肿)、S(水肿存在但无利尿剂治疗,或水肿通过利尿剂消退)或Y(尽管利尿剂治疗仍有水肿)。由于体内过多的液体而引起的肿胀,通常会加重预后并表明较差的状态。 | - | 否 |
Bilirubin | 特征 | 连续 | - | 血清胆红素。高水平可能表明肝功能障碍,并可能与更严重的疾病和较差的状态相关。 | mg/dL | 否 |
Cholesterol | 特征 | 整数 | - | 血清胆固醇。虽然与肝功能无直接关系,但异常水平可能与某些肝脏疾病和整体健康状况相关。 | mg/dL | 是 |
Albumin | 特征 | 连续 | - | 白蛋白。低水平可能是肝病的迹象,并可能表明肝脏合成蛋白质能力降低,从而导致较差的状态。 | g/dL | 否 |
Copper | 特征 | 整数 | - | 尿铜。在某些肝脏疾病(如威尔逊病)中升高,如果水平异常高可能会影响状态。 | µg/day | 是 |
Alk_Phos | 特征 | 连续 | - | 碱性磷酸酶。与胆管相关的一种酶;高水平可能表明阻塞或其他与肝脏相关的问题。 | U/Liter | 是 |
SGOT | 特征 | 连续 | - | 丙氨酸氨基转移酶。当升高时,可能表明肝损伤,并可能与恶化的状态相关。 | U/mL | 是 |
Triglycerides | 特征 | 整数 | - | 甘油三酯。虽然主要是心血管风险指标,但它们可能受肝功能和患者状态的影响。 | - | 是 |
Platelets | 特征 | 整数 | - | 每立方的血小板。低血小板计数可能是晚期肝病的结果,并可能表明较差的状态。 | mL/1000 | 是 |
Prothrombin | 特征 | 连续 | - | 凝血酶原时间。血液凝固所需的时间的测量。肝病可以导致时间延长,表明较差的状态。 | s | 是 |
Stage | 特征 | 分类 | - | 疾病的组织学阶段(1、2、3或4)。肝脏疾病的阶段与患者的状态直接相关-阶段越高,病情越严重。 | - | 是 |
Status | 目标 | 分类 | - | 患者的状态:C(被审查)、CL(因肝脏移植而被审查)或D(死亡) | - | 否 |
import sys
from catboost import CatBoostClassifier, CatBoostRegressor
from imblearn.over_sampling import RandomOverSampler
import lightgbm as lgb
import matplotlib.pyplot as plt
import numpy as np
import optuna
import pandas as pd
import plotly.express as px
import seaborn as sns
from sklearn.base import BaseEstimator, ClassifierMixin, TransformerMixin
from sklearn.decomposition import PCA
from sklearn.ensemble import RandomForestClassifier
from sklearn.ensemble import VotingClassifier
from sklearn.linear_model import LogisticRegression
from sklearn.manifold import TSNE
from sklearn.metrics import confusion_matrix, ConfusionMatrixDisplay
from sklearn.metrics import log_loss
from sklearn.metrics import make_scorer, mean_squared_error
from sklearn.model_selection import GridSearchCV
from sklearn.model_selection import cross_val_score, KFold, StratifiedKFold, GroupKFold, RepeatedStratifiedKFold
from sklearn.model_selection import train_test_split
from sklearn.naive_bayes import MultinomialNB, CategoricalNB, GaussianNB
from sklearn.neighbors import KNeighborsClassifier
from sklearn.pipeline import make_pipeline, Pipeline
from sklearn.preprocessing import LabelEncoder, OrdinalEncoder, OneHotEncoder
from sklearn.preprocessing import StandardScaler
from sklearn.svm import SVC
from sklearn.tree import DecisionTreeClassifier
from tqdm import tqdm
import xgboost as xgb
from xgboost import plot_importance
from ydata_profiling import ProfileReport
pd.set_option('display.max_columns', None)
# 设置显示所有列
sns.set_palette(sns.color_palette("Set2"))
# 设置颜色主题
print(f"Running on {sys.version}")
# 打印Python版本
KAGGLE_ENV = True
GENERATE_REPORTS = False
# 如果我们在Kaggle环境中运行,则需要使用Kaggle提供的数据路径。
if KAGGLE_ENV:
data_path = "../input/"
else:
data_path = "./data/"
/opt/conda/lib/python3.10/site-packages/numba/core/decorators.py:262: NumbaDeprecationWarning: [1mnumba.generated_jit is deprecated. Please see the documentation at: https://numba.readthedocs.io/en/stable/reference/deprecation.html#deprecation-of-generated-jit for more information and advice on a suitable replacement.[0m
warnings.warn(msg, NumbaDeprecationWarning)
/opt/conda/lib/python3.10/site-packages/visions/backends/shared/nan_handling.py:51: NumbaDeprecationWarning: [1mThe 'nopython' keyword argument was not supplied to the 'numba.jit' decorator. The implicit default value for this argument is currently False, but it will be changed to True in Numba 0.59.0. See https://numba.readthedocs.io/en/stable/reference/deprecation.html#deprecation-of-object-mode-fall-back-behaviour-when-using-jit for details.[0m
def hasna(x: np.ndarray) -> bool:
Running on 3.10.12 | packaged by conda-forge | (main, Jun 23 2023, 22:40:32) [GCC 12.3.0]
# 读取训练数据集
df_train = pd.read_csv(data_path + "playground-series-s3e26/train.csv").drop(
["id"], axis=1
)
# 读取测试数据集
df_test = pd.read_csv(data_path + "playground-series-s3e26/test.csv")
# 保存测试数据集的id列
test_IDs = df_test.id
# 在测试数据集中删除id列
df_test = df_test.drop("id", axis=1)
# 读取样本提交数据集
df_sample_sub = pd.read_csv(data_path + "playground-series-s3e26/sample_submission.csv")
# 读取辅助数据集
df_supp = pd.read_csv(
data_path + "cirrhosis-patient-survival-prediction/cirrhosis.csv"
)[df_train.columns]
# 合并辅助数据集和训练数据集
df_train = pd.concat(objs=[df_train, df_supp]).reset_index(drop=True)
# 设置标签列名
LABEL = "Status"
# 设置分类特征列名
CAT_FEATS = ["Drug", "Sex", "Ascites", "Hepatomegaly", "Spiders", "Edema", "Stage"]
# 设置数值特征列名
NUM_FEATS = [x for x in df_train.columns if x not in CAT_FEATS and x != LABEL]
# 保存原始特征列名
ORG_FEATS = df_train.drop(LABEL, axis=1).columns.tolist()
# 打印训练数据集的形状
print(f"Train shape: {df_train.shape}")
# 打印测试数据集的形状
print(f"Test shape: {df_test.shape}")
Train shape: (8323, 19)
Test shape: (5271, 18)
EDA | 描述性统计学
# 读取训练数据集
df_train
| N_Days | Drug | Age | Sex | Ascites | Hepatomegaly | Spiders | Edema | Bilirubin | Cholesterol | Albumin | Copper | Alk_Phos | SGOT | Tryglicerides | Platelets | Prothrombin | Stage | Status | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 999 | D-penicillamine | 21532 | M | N | N | N | N | 2.3 | 316.0 | 3.35 | 172.0 | 1601.0 | 179.80 | 63.0 | 394.0 | 9.7 | 3.0 | D |
| 1 | 2574 | Placebo | 19237 | F | N | N | N | N | 0.9 | 364.0 | 3.54 | 63.0 | 1440.0 | 134.85 | 88.0 | 361.0 | 11.0 | 3.0 | C |
| 2 | 3428 | Placebo | 13727 | F | N | Y | Y | Y | 3.3 | 299.0 | 3.55 | 131.0 | 1029.0 | 119.35 | 50.0 | 199.0 | 11.7 | 4.0 | D |
| 3 | 2576 | Placebo | 18460 | F | N | N | N | N | 0.6 | 256.0 | 3.50 | 58.0 | 1653.0 | 71.30 | 96.0 | 269.0 | 10.7 | 3.0 | C |
| 4 | 788 | Placebo | 16658 | F | N | Y | N | N | 1.1 | 346.0 | 3.65 | 63.0 | 1181.0 | 125.55 | 96.0 | 298.0 | 10.6 | 4.0 | C |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 8318 | 681 | NaN | 24472 | F | NaN | NaN | NaN | N | 1.2 | NaN | 2.96 | NaN | NaN | NaN | NaN | 174.0 | 10.9 | 3.0 | D |
| 8319 | 1103 | NaN | 14245 | F | NaN | NaN | NaN | N | 0.9 | NaN | 3.83 | NaN | NaN | NaN | NaN | 180.0 | 11.2 | 4.0 | C |
| 8320 | 1055 | NaN | 20819 | F | NaN | NaN | NaN | N | 1.6 | NaN | 3.42 | NaN | NaN | NaN | NaN | 143.0 | 9.9 | 3.0 | C |
| 8321 | 691 | NaN | 21185 | F | NaN | NaN | NaN | N | 0.8 | NaN | 3.75 | NaN | NaN | NaN | NaN | 269.0 | 10.4 | 3.0 | C |
| 8322 | 976 | NaN | 19358 | F | NaN | NaN | NaN | N | 0.7 | NaN | 3.29 | NaN | NaN | NaN | NaN | 350.0 | 10.6 | 4.0 | C |
8323 rows × 19 columns
%%time # 记录代码运行时间
if GENERATE_REPORTS: # 如果需要生成报告
# 生成数据分析报告
profile = ProfileReport(df_train, title="YData Profiling Report - Cirrhosis")
profile.to_notebook_iframe() # 将报告以iframe形式嵌入notebook中显示
CPU times: user 4 µs, sys: 0 ns, total: 4 µs
Wall time: 8.11 µs
# 将训练数据的描述统计信息存储在desc_df中
desc_df = df_train.describe(include="all")
# 转置desc_df,使得每一列对应一个特征
desc_df = desc_df.T
# 填充unique列中的缺失值,使用df_train中的唯一值数量进行填充
desc_df['unique'] = desc_df['unique'].fillna(df_train.nunique())
# 将count列的数据类型转换为int16
desc_df['count'] = desc_df['count'].astype('int16')
# 计算missing列的值,即数据集中的样本数量减去count列的值
desc_df['missing'] = df_train.shape[0] - desc_df['count']
# 返回添加注释后的desc_df数据框
desc_df
| count | unique | top | freq | mean | std | min | 25% | 50% | 75% | max | missing | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|
| N_Days | 8323 | 495 | NaN | NaN | 2024.528776 | 1094.968696 | 41.0 | 1220.0 | 1831.0 | 2689.0 | 4795.0 | 0 |
| Drug | 8217 | 2 | Placebo | 4164 | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 106 |
| Age | 8323 | 397 | NaN | NaN | 18381.192359 | 3686.832308 | 9598.0 | 15574.0 | 18713.0 | 20684.0 | 28650.0 | 0 |
| Sex | 8323 | 2 | F | 7710 | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 0 |
| Ascites | 8217 | 2 | N | 7813 | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 106 |
| Hepatomegaly | 8217 | 2 | Y | 4202 | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 106 |
| Spiders | 8217 | 2 | N | 6188 | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 106 |
| Edema | 8323 | 3 | N | 7515 | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 0 |
| Bilirubin | 8323 | 117 | NaN | NaN | 2.62594 | 3.847146 | 0.3 | 0.7 | 1.1 | 3.0 | 28.0 | 0 |
| Cholesterol | 8189 | 227 | NaN | NaN | 351.219074 | 196.775246 | 120.0 | 248.0 | 299.0 | 392.0 | 1775.0 | 134 |
| Albumin | 8323 | 167 | NaN | NaN | 3.545767 | 0.350697 | 1.96 | 3.35 | 3.58 | 3.77 | 4.64 | 0 |
| Copper | 8215 | 171 | NaN | NaN | 84.421546 | 76.32748 | 4.0 | 39.0 | 63.0 | 102.0 | 588.0 | 108 |
| Alk_Phos | 8217 | 364 | NaN | NaN | 1823.044883 | 1913.388685 | 289.0 | 834.0 | 1181.0 | 1857.0 | 13862.4 | 106 |
| SGOT | 8217 | 206 | NaN | NaN | 114.90653 | 49.134067 | 26.35 | 75.95 | 108.5 | 137.95 | 457.25 | 106 |
| Tryglicerides | 8187 | 154 | NaN | NaN | 115.662636 | 53.03766 | 33.0 | 84.0 | 104.0 | 139.0 | 598.0 | 136 |
| Platelets | 8312 | 252 | NaN | NaN | 264.827238 | 88.039809 | 62.0 | 210.0 | 264.0 | 316.0 | 721.0 | 11 |
| Prothrombin | 8321 | 50 | NaN | NaN | 10.634575 | 0.795707 | 9.0 | 10.0 | 10.6 | 11.0 | 18.0 | 2 |
| Stage | 8317 | 4 | NaN | NaN | 3.032103 | 0.867235 | 1.0 | 2.0 | 3.0 | 4.0 | 4.0 | 6 |
| Status | 8323 | 3 | C | 5197 | NaN | NaN | NaN | NaN | NaN | NaN | NaN | 0 |
我们可以看到,在合并的训练数据集中存在缺失值。然而,没有发现重复项。
请注意,Prothrombin仅显示50个不同的值,因此考虑将此特征视为分类变量可能是值得的(其他特征也可能如此)
让我们检查目标变量Status的分布情况。
# 统计每个类别的观测数量
status_counts = df_train[LABEL].value_counts()
labels = status_counts.index
sizes = status_counts.values
# 计算每个类别的百分比
percentages = 100.*sizes/sizes.sum()
# 创建带有百分比标签的饼图
plt.figure(figsize=(10, 6))
plt.pie(sizes, labels=[f"{l}, {s:.1f}%" for l, s in zip(labels, percentages)], startangle=90)
plt.gca().set_aspect("equal")
plt.legend(loc="upper right", bbox_to_anchor=(1.2, 1), labels=labels, title=LABEL)
plt.title(f"Distribution of {LABEL}")
plt.show()

我们可以看到目标变量是不平衡的。大多数患者都是被审查的(意味着患者在随访期间失去了联系,或者研究在患者死亡或接受肝移植之前结束)。这是生存分析中常见的问题,我们稍后会处理它。
还要注意,
StatusCL只有很少的观察结果。处理CL观察结果可能需要采取特殊的方式,例如从分类中删除它们。
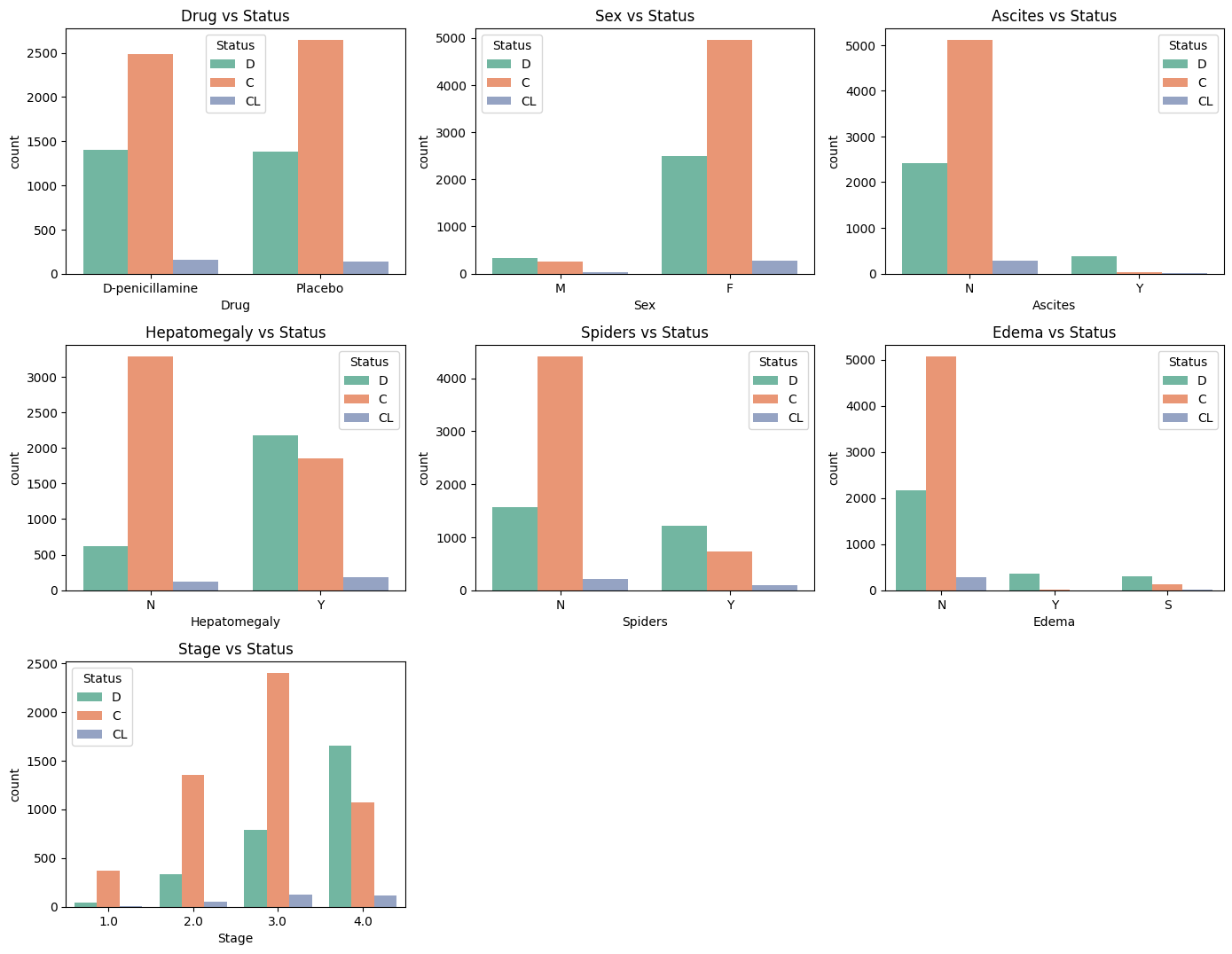
现在让我们检查特征的分布。我们将从分类特征开始。
# 设置图形大小
plt.figure(figsize=(14, len(CAT_FEATS) * 2))
# 遍历分类特征
for i, col in enumerate(CAT_FEATS):
# 在图形中创建子图
plt.subplot(len(CAT_FEATS) // 2 + 1, 3, i + 1)
# 绘制计数柱状图
sns.countplot(x=col, hue=LABEL, data=df_train)
# 设置子图标题
plt.title(f"{col} vs {LABEL}")
# 调整子图布局
plt.tight_layout()
让我们检查连续特征的分布。
# 创建一个2行3列的图像,大小为16x10
fig, axes = plt.subplots(2, 3, figsize=(16, 10))
# 使用enumerate函数遍历axes中的每个子图
for i, ax in enumerate(axes.flatten()):
# 在当前子图上绘制小提琴图,x轴为LABEL,y轴为NUM_FEATS[i],数据来源为df_train,绘制在ax上
sns.violinplot(x=LABEL, y=NUM_FEATS[i], data=df_train, ax=ax)
# 设置x轴刻度为原始标签(逆转换)
ax.set_title(f"{NUM_FEATS[i]} vs {LABEL}")
# 调整子图的布局
plt.tight_layout()
# 显示图像
plt.show()

我们得到了一些离群值。我们将在特征工程部分中删除它们。另外,我们最初对每个特征对“状态”的不同影响的初步假设似乎在第一眼看起来是成立的(仅考虑主要影响,没有考虑混淆影响)。
让我们检查特征之间的相关性。
# 设置图像大小
plt.figure(figsize=(12, 8))
# 创建一个布尔类型的矩阵,用于遮盖热力图中的下三角部分
mask = np.triu(np.ones_like(df_train[NUM_FEATS].corr(), dtype=bool))
# 绘制热力图,显示数值,并遮盖下三角部分
sns.heatmap(df_train[NUM_FEATS].corr(), annot=True, mask=mask)
# 显示图像
plt.show()
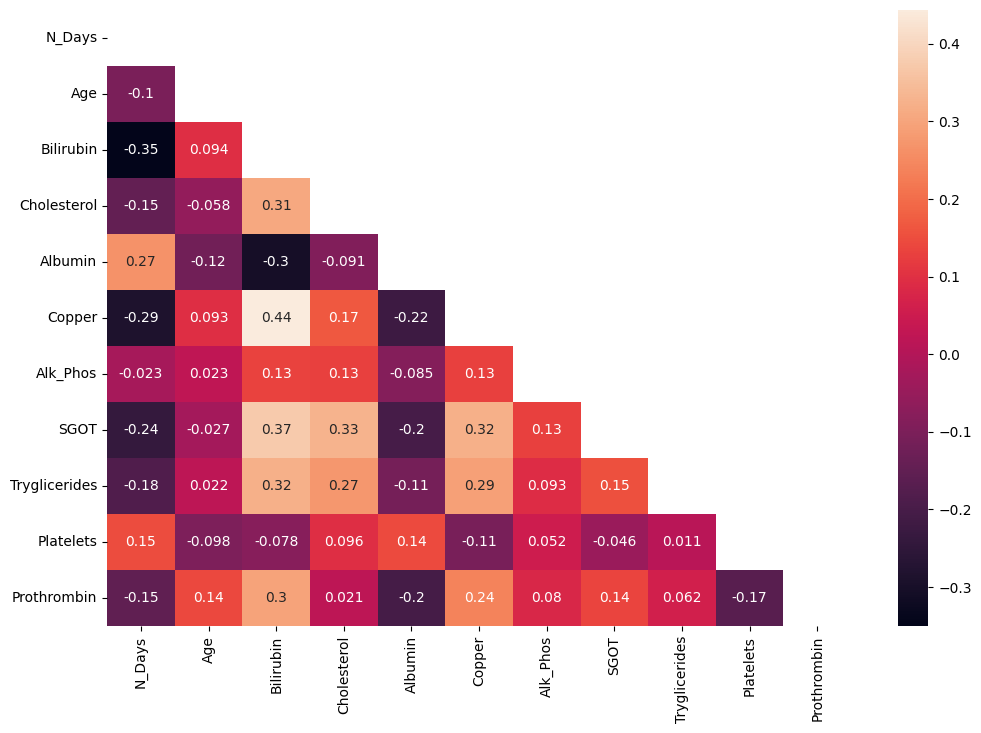
没有高度相关的特征。然而,我们可以看到Copper和Bilirubin之间存在中度正相关性,以及Albumin和Bilirubin之间存在中度负相关性。这是预期的,因为Copper和Bilirubin都与肝功能有关(特别是在肝功能障碍的情况下)。此外,Albumin和Bilirubin之间存在负相关性,因为Albumin是肝脏产生的蛋白质,而Bilirubin是红细胞分解的废物产物。
从Dee Dee的EDA笔记本中,我们还可以看到训练和测试数据之间的相关性几乎遵循相同的模式。
让我们在一对图中进一步分析相关性和Status聚类。
# 使用seaborn库中的pairplot函数绘制散点图矩阵
# df_train是数据集,包含了NUM_FEATS和LABEL这些特征
# sample函数用于随机抽样,frac参数表示抽样比例为0.01
# hue参数用于指定按照LABEL进行分类,并以不同颜色显示
# corner参数用于将图形的上三角和下三角设置为空白
pairplot = sns.pairplot(df_train[NUM_FEATS + [LABEL]].sample(frac=.01),
hue=LABEL,
corner=True)

在我看来,某些特征可以通过线性方法(仅检查2D空间)有效地分离。因此,经典的SVM方法可能表现得不错…
此外,特征分布(直方图)显示出一些漂亮的“钟形曲线”…
特征工程
TODO 我应该在这个笔记本中包含一个特征和转换的“概述”表格…
# 设置数据版本以存储数据
DATA_VERSION = 24
# 类别特征
CAT_FEATS = ["Drug", "Sex", "Ascites", "Hepatomegaly", "Spiders", "Edema", "Stage"]
# 数值特征
NUM_FEATS = [x for x in df_train.columns if x not in CAT_FEATS and x != LABEL]
# 复制数据框并进行修改
df_train_mod = df_train.copy()
df_test_mod = df_test.copy()
# 打印训练数据框的形状
print(f"Train shape: {df_train_mod.shape}")
# 打印测试数据框的形状
print(f"Test shape: {df_test_mod.shape}")
# 断言训练数据框的列数减1等于测试数据框的列数
assert df_train_mod.shape[1]-1 == df_test_mod.shape[1]
Train shape: (8323, 19)
Test shape: (5271, 18)
填补缺失值
受到这个notebook的启发。如果你喜欢这个工作,请点赞。
然而,在处理填补值时,我需要使用optuna(其他notebook)来调整参数优化… 目前还不令人满意 - 因此,我只是删除了带有缺失值的观测数据。
# 找出缺失的分类特征
missing_cat=[f for f in df_train_mod.columns if df_train_mod[f].dtype=="O" if df_train_mod[f].isna().sum()>0]
# 计算训练集和测试集中缺失值的百分比
train_missing_pct = df_train_mod[missing_cat].isnull().mean() * 100
test_missing_pct = df_train_mod[missing_cat].isnull().mean() * 100
# 将训练集和测试集中缺失值的百分比合并为一个DataFrame
missing_pct_df = pd.concat([train_missing_pct, test_missing_pct], axis=1, keys=['Train %', 'Test%'])
print(missing_pct_df)
# 定义分类特征填充的参数
cat_params={
'depth': 6,
'learning_rate': 0.1,
'l2_leaf_reg': 0.7,
'random_strength': 0.2,
'max_bin': 200,
'od_wait': 65,
'one_hot_max_size': 70,
'grow_policy': 'Depthwise',
'bootstrap_type': 'Bayesian',
'od_type': 'Iter',
'eval_metric': 'MultiClass',
'loss_function': 'MultiClass',
}
# 定义函数:存储每个特征中缺失值的行
def store_missing_rows(df, features):
missing_rows = {}
for feature in features:
missing_rows[feature] = df[df[feature].isnull()]
return missing_rows
# 定义函数:填充分类特征中的缺失值
def fill_missing_categorical(train, test, target, features, max_iterations=10):
df = pd.concat([train.drop(columns=target), test], axis="rows")
df = df.reset_index(drop=True)
# Step 1: 存储每个特征中缺失值的行
missing_rows = store_missing_rows(df, features)
# Step 2: 初始时将所有缺失值填充为"Missing"
for f in features:
df[f] = df[f].fillna("Missing_" + f)
for iteration in tqdm(range(max_iterations), desc="Iterations"):
for feature in features:
# 跳过没有缺失值的特征
rows_miss = missing_rows[feature].index
missing_temp = df.loc[rows_miss].copy()
non_missing_temp = df.drop(index=rows_miss).copy()
missing_temp = missing_temp.drop(columns=[feature])
other_features = [x for x in df.columns if x != feature and df[x].dtype == "O"]
X_train = non_missing_temp.drop(columns=[feature])
y_train = non_missing_temp[[feature]]
# 使用CatBoostClassifier模型进行训练
catboost_classifier = CatBoostClassifier(**cat_params)
catboost_classifier.fit(X_train, y_train, cat_features=other_features, verbose=False)
# Step 4: 预测特征的缺失值并更新所有N个特征
y_pred = catboost_classifier.predict(missing_temp)
# 如果需要,将y_pred转换为字符串
if y_pred.dtype != "O":
y_pred = y_pred.astype(str)
df.loc[rows_miss, feature] = y_pred
train[features] = np.array(df.iloc[:train.shape[0]][features])
test[features] = np.array(df.iloc[train.shape[0]:][features])
return train, test
Train % Test%
Drug 1.273579 1.273579
Ascites 1.273579 1.273579
Hepatomegaly 1.273579 1.273579
Spiders 1.273579 1.273579
# 找出缺失值的特征
missing_num=[f for f in df_train_mod.columns if df_train_mod[f].dtype!="O" and df_train_mod[f].isna().sum()>0]
# 计算训练集和测试集中缺失值的百分比
train_missing_pct = df_train_mod[missing_num].isnull().mean() * 100
test_missing_pct = df_test_mod[missing_num].isnull().mean() * 100
# 将训练集和测试集中缺失值的百分比合并到一个DataFrame中
missing_pct_df = pd.concat([train_missing_pct, test_missing_pct], axis=1, keys=['Train %', 'Test%'])
print(missing_pct_df)
# 定义CatBoost和LightGBM的参数
cb_params = {
'iterations': 500,
'depth': 6,
'learning_rate': 0.02,
'l2_leaf_reg': 0.5,
'random_strength': 0.2,
'max_bin': 150,
'od_wait': 80,
'one_hot_max_size': 70,
'grow_policy': 'Depthwise',
'bootstrap_type': 'Bayesian',
'od_type': 'IncToDec',
'eval_metric': 'RMSE',
'loss_function': 'RMSE',
'random_state': 42,
}
lgb_params = {
'n_estimators': 50,
'max_depth': 8,
'learning_rate': 0.02,
'subsample': 0.20,
'colsample_bytree': 0.56,
'reg_alpha': 0.25,
'reg_lambda': 5e-08,
'objective': 'multiclass',
'metric': 'multi_logloss',
'boosting_type': 'gbdt',
'random_state': 42,
}
# 定义计算RMSE的函数
def rmse(y1,y2):
return(np.sqrt(mean_squared_error(y1,y2)))
# 定义填充缺失值的函数
def fill_missing_numerical(train,test,target, features, max_iterations=10):
train_temp=train.copy()
if target in train_temp.columns:
train_temp=train_temp.drop(columns=target)
# 将训练集和测试集合并到一个DataFrame中
df=pd.concat([train_temp,test],axis="rows")
df=df.reset_index(drop=True)
# Step 1: 存储每个特征中缺失值的实例
missing_rows = store_missing_rows(df, features)
# Step 2: 初始时将所有缺失值填充为"Missing"
for f in features:
df[f]=df[f].fillna(df[f].mean())
# 找出非数值型特征
cat_features=[f for f in df.columns if not pd.api.types.is_numeric_dtype(df[f])]
dictionary = {feature: [] for feature in features}
# 迭代填充缺失值
for iteration in tqdm(range(max_iterations), desc="Iterations"):
for feature in features:
# 跳过没有缺失值的特征
rows_miss = missing_rows[feature].index
missing_temp = df.loc[rows_miss].copy()
non_missing_temp = df.drop(index=rows_miss).copy()
y_pred_prev=missing_temp[feature]
missing_temp = missing_temp.drop(columns=[feature])
# Step 3: 使用其余特征使用随机森林预测缺失值
X_train = non_missing_temp.drop(columns=[feature])
y_train = non_missing_temp[[feature]]
model = CatBoostRegressor(**cb_params)
# if iteration>3:
# model = lgb.LGBMRegressor()
model.fit(X_train, y_train,cat_features=cat_features, verbose=False)
# Step 4: 预测缺失值并更新所有N个特征
y_pred = model.predict(missing_temp)
df.loc[rows_miss, feature] = y_pred
error_minimize=rmse(y_pred,y_pred_prev)
dictionary[feature].append(error_minimize) # 将error_minimize值添加到字典中
for feature, values in dictionary.items():
iterations = range(1, len(values) + 1) # x轴的值(迭代次数)
plt.plot(iterations, values, label=feature) # 绘制图像
plt.xlabel('Iterations')
plt.ylabel('RMSE')
plt.title('Minimization of RMSE with iterations')
plt.legend(bbox_to_anchor=(1.05, 1), loc='upper left')
plt.show()
train[features] = np.array(df.iloc[:train.shape[0]][features])
test[features] = np.array(df.iloc[train.shape[0]:][features])
return train,test
Train % Test%
Cholesterol 1.609996 0.0
Copper 1.297609 0.0
Alk_Phos 1.273579 0.0
SGOT 1.273579 0.0
Tryglicerides 1.634026 0.0
Platelets 0.132164 0.0
Prothrombin 0.024030 0.0
Stage 0.072089 0.0
# 如果DROP_MISSING为True,则删除缺失值
if DROP_MISSING:
# TODO: 这只是初步的处理方法...不太优雅
df_train_mod = df_train_mod.dropna()
df_test_mod = df_test_mod.dropna()
else:
# 否则,使用fill_missing_categorical函数填充分类变量的缺失值
df_train_mod, df_test_mod = fill_missing_categorical(df_train_mod, df_test_mod, LABEL, missing_cat, 5)
# 使用fill_missing_numerical函数填充数值变量的缺失值
df_train_mod, df_test_mod = fill_missing_numerical(df_train_mod, df_test_mod, LABEL, missing_num, 5)
标签和特征编码
由于一些特征是分类的,我们将对它们进行转换。
| 编码技术 | 变量类型 | 支持高基数 | 处理未知变量 | 缺点 |
|---|---|---|---|---|
| 标签编码 | 名义 | 是 | 否 | 未知变量 |
| 有序编码 | 有序 | 是 | 是 | 类别被解释为数值 |
| One-Hot / Dummy 编码 | 名义 | 否 | 是 | 虚拟变量陷阱 大数据集 |
| 目标编码 | 名义 | 是 | 是 | 目标泄漏 类别分布不均 |
| 频率 / 计数编码 | 名义 | 是 | 是 | 类似编码 |
| 二进制编码 | 名义 | 是 | 是 | 不可逆 |
| 哈希编码 | 名义 | 是 | 是 | 信息丢失或冲突 |
(感谢这个notebook)
- 对于特征
Edema,使用One-Hot-Encoding编码 - 对于特征
Stage,使用Ordinal Encoding编码 - 对于特征
Drug、Sex、Ascites、Hepatomegaly、Spiders,使用Binary-Encoding编码 - 对于标签
Status,使用LabelEncoding编码
使用One-Hot-Encoding而不是OrdinalEncoding(N: 0, S: 1, Y: 2)对Edema进行编码稍微提高了分数。
标签编码
# 实例化一个标签编码器
label_encoder = LabelEncoder()
# 对训练数据集中的标签进行编码
df_train_mod[LABEL] = label_encoder.fit_transform(df_train_mod[LABEL])
特征编码
# encoders是一个字典,包含了各种特征的编码器
encoders = {
# 'Drug'特征的编码器,使用了OrdinalEncoder方法,未知值设为-1,已知值为['Placebo', 'D-penicillamine']
'Drug': OrdinalEncoder(handle_unknown='use_encoded_value', unknown_value=-1, categories=[['Placebo', 'D-penicillamine']]),
# 'Sex'特征的编码器,使用了OrdinalEncoder方法,未知值设为-1
'Sex': OrdinalEncoder(handle_unknown='use_encoded_value', unknown_value=-1),
# 'Ascites'特征的编码器,使用了OrdinalEncoder方法,未知值设为-1
'Ascites': OrdinalEncoder(handle_unknown='use_encoded_value', unknown_value=-1),
# 'Hepatomegaly'特征的编码器,使用了OrdinalEncoder方法,未知值设为-1
'Hepatomegaly': OrdinalEncoder(handle_unknown='use_encoded_value', unknown_value=-1),
# 'Spiders'特征的编码器,使用了OrdinalEncoder方法,未知值设为-1
'Spiders': OrdinalEncoder(handle_unknown='use_encoded_value', unknown_value=-1),
# 'Edema'特征的编码器,使用了OneHotEncoder方法
'Edema': OneHotEncoder(),
# 'Stage'特征的编码器,使用了OrdinalEncoder方法,未知值设为-1
'Stage': OrdinalEncoder(handle_unknown='use_encoded_value', unknown_value=-1)
}
for feat, enc in encoders.items():
if isinstance(enc, OrdinalEncoder):
# 对训练集和测试集中的特征进行编码,并将结果转换为整数类型
df_train_mod[feat] = enc.fit_transform(df_train_mod[[feat]]).astype('int32')
df_test_mod[feat] = enc.transform(df_test_mod[[feat]]).astype('int32')
if isinstance(enc, OneHotEncoder):
# 对训练集中的特征进行编码,并获取新的列名
new_cols = enc.fit_transform(df_train_mod[[feat]]).toarray().astype('int8')
# col_names = [f"{feat}_{cat}" for cat in enc.categories_[0]]
col_names = enc.get_feature_names_out()
# 将新的列添加到数据框中
df_train_mod[col_names] = new_cols
df_train_mod.drop(feat, axis=1, inplace=True) # 删除原始列
# 对测试集进行相同的操作
new_cols_test = enc.transform(df_test_mod[[feat]]).toarray().astype('int8')
df_test_mod[col_names] = new_cols_test
df_test_mod.drop(feat, axis=1, inplace=True)
额外功能
我们将创建一些额外的功能。下面提供了解释:
| 转换器类 | 类型 | 描述 |
|---|---|---|
DiagnosisDateTransformer | num | 通过从“年龄”中减去“N_Days”来计算“诊断日期”。这可能提供了一个更直接的时间自诊断,有助于分析。 |
AgeBinsTransformer | cat | 将“年龄”分为区间(19、29、49、64、99),将连续变量转换为分类变量,以简化分析。 |
BilirubinAlbuminTransformer | num | 通过将“Bilirubin”和“Albumin”相乘创建一个新特征“Bilirubin_Albumin”,可能突出这两个变量之间的相互作用。 |
NormalizeLabValuesTransformer | num | 将实验室值(如“Bilirubin”、“胆固醇”等)标准化为它们的z分数,为建模目的标准化这些特征。 |
DrugEffectivenessTransformer | num | 通过结合“Drug”和“Bilirubin”水平生成一个新特征“Drug_Effectiveness”。这假设“Bilirubin”的变化反映了药物的有效性。 |
SymptomScore(Cat)Transformer | num | 将症状(如“腹水”、“肝肿大”等)的存在总结为单个“Symptom_Score”,简化患者症状的表示。 |
LiverFunctionTransformer | num | 将关键肝功能测试的平均值计算为“Liver_Function_Index”,提供全面的肝健康度量。 |
RiskScoreTransformer | num | 使用“Bilirubin”、“Albumin”和“Alk_Phos”的组合计算“Risk_Score”,可能为患者提供综合风险评估。 |
TimeFeaturesTransformer | num | 从“N_Days”中提取“Year”和“Month”,将连续时间测量转换为更易于解释的分类时间单位。 |
class DiagnosisDateTransformer(BaseEstimator, TransformerMixin):
def fit(self, X, y=None):
return self
def transform(self, X):
X['Diagnosis_Date'] = X['Age'] - X['N_Days'] # 计算诊断日期
return X
class AgeYearsTransformer(BaseEstimator, TransformerMixin):
def fit(self, X, y=None):
return self
def transform(self, X):
X['Age_Years'] = round(X['Age'] / 365.25).astype("int16") # 计算年龄(以年为单位)
return X
class AgeGroupsTransformer(BaseEstimator, TransformerMixin):
"""老年人可能更容易受到健康问题的影响(相互作用)。也可以涵盖生活方式的影响,例如饮酒等。"""
def fit(self, X, y=None):
return self
def transform(self, X):
# 使用上面的年份,最小值为26,最大值为78
X['Age_Group'] = pd.cut(X['Age_Years'], bins=[19, 29, 49, 64, 99], labels = [0, 1, 2, 3]).astype('int16') # 将年龄分组
return X
class BilirubinAlbuminTransformer(BaseEstimator, TransformerMixin):
def fit(self, X, y=None):
return self
def transform(self, X):
X['Bilirubin_Albumin'] = X['Bilirubin'] * X['Albumin'] # 计算胆红素和白蛋白的乘积
return X
class DrugEffectivenessTransformer(BaseEstimator, TransformerMixin):
# 占位符概念,假设“胆红素”改善是有效性的一种衡量指标
def fit(self, X, y=None):
return self
def transform(self, X):
X['Drug_Effectiveness'] = X['Drug'] * X['Bilirubin'] # 计算药物和胆红素的乘积
return X
class SymptomScoreTransformer(BaseEstimator, TransformerMixin):
# 根据数据集的解释,我们添加所有“不良”症状
def fit(self, X, y=None):
return self
def transform(self, X):
# symptom_columns = ['Ascites', 'Hepatomegaly', 'Spiders', 'Edema']
symptom_columns = ['Ascites', 'Hepatomegaly', 'Spiders', 'Edema_N', 'Edema_S', 'Edema_Y']
X['Symptom_Score'] = X[symptom_columns].sum(axis=1) # 计算症状得分
return X
class SymptomCatTransformer(BaseEstimator, TransformerMixin):
def __init__(self):
self.symptom_columns = ['Ascites', 'Hepatomegaly', 'Spiders', 'Edema_N', 'Edema_S', 'Edema_Y']
self.encoder = OneHotEncoder(handle_unknown='ignore')
def fit(self, X, y=None):
X_copy = X.copy()
symptom_scores = X_copy[self.symptom_columns].apply(lambda row: ''.join(row.values.astype(str)), axis=1)
self.encoder.fit(symptom_scores.values.reshape(-1, 1)) # 对症状得分进行独热编码
return self
def transform(self, X):
X_transformed = X.copy()
symptom_scores = X_transformed[self.symptom_columns].apply(lambda row: ''.join(row.values.astype(str)), axis=1)
encoded_features = self.encoder.transform(symptom_scores.values.reshape(-1, 1)).toarray().astype("int8") # 对症状得分进行编码
encoded_feature_names = self.encoder.get_feature_names_out(input_features=['Symptom_Score'])
# 删除原始的症状列,并添加新的编码特征
# X_transformed.drop(columns=self.symptom_columns, inplace=True)
X_transformed[encoded_feature_names] = pd.DataFrame(encoded_features, index=X_transformed.index)
return X_transformed
class LiverFunctionTransformer(BaseEstimator, TransformerMixin):
def fit(self, X, y=None):
return self
def transform(self, X):
liver_columns = ['Bilirubin', 'Albumin', 'Alk_Phos', 'SGOT']
X['Liver_Function_Index'] = X[liver_columns].mean(axis=1) # 计算肝功能指数
return X
class RiskScoreTransformer(BaseEstimator, TransformerMixin):
def fit(self, X, y=None):
return self
def transform(self, X):
X['Risk_Score'] = X['Bilirubin'] + X['Albumin'] - X['Alk_Phos'] # 计算风险得分
return X
class TimeFeaturesTransformer(BaseEstimator, TransformerMixin):
def fit(self, X, y=None):
return self
def transform(self, X):
X['Diag_Year'] = (X['N_Days'] / 365).astype(int) # 计算诊断年份
X['Diag_Month'] = ((X['N_Days'] % 365) / 30).astype(int) # 计算诊断月份
return X
class ScalingTransformer(BaseEstimator, TransformerMixin):
def __init__(self):
self.scaler = StandardScaler()
self.num_feats = NUM_FEATS + ['Diagnosis_Date', 'Age_Years', 'Bilirubin_Albumin', 'Drug_Effectiveness',
'Symptom_Score', 'Liver_Function_Index', 'Risk_Score', 'Diag_Year', 'Diag_Month']
def fit(self, X, y=None):
self.scaler.fit(X[self.num_feats])
return self
def transform(self, X):
X_scaled = X.copy()
X_scaled[self.num_feats] = self.scaler.transform(X_scaled[self.num_feats]) # 对数值特征进行标准化
return X_scaled
# 定义流水线
pipeline = Pipeline([
('diagnosis_date', DiagnosisDateTransformer()),
('age_years', AgeYearsTransformer()),
('age_groups', AgeGroupsTransformer()),
('bilirubin_albumin', BilirubinAlbuminTransformer()),
('drug_effectiveness', DrugEffectivenessTransformer()),
('symptom_score', SymptomScoreTransformer()),
('symptom_cat_score', SymptomCatTransformer()),
('liver_function', LiverFunctionTransformer()),
('risk_score', RiskScoreTransformer()),
('time_features', TimeFeaturesTransformer()),
#('scaling', ScalingTransformer()),
# ... ?
])
# 将流水线应用于数据框
df_train_mod = pipeline.fit_transform(df_train_mod)
df_test_mod = pipeline.transform(df_test_mod)
# 更新CAT_FEATS
CAT_FEATS = ['Drug', 'Sex', 'Ascites', 'Hepatomegaly', 'Spiders', 'Edema', 'Stage', #old
'Age_Group', 'Symptom_Score'] # new
# 更新NUM_FEATS ????
异常值检测和移除
如前所述,数值特征看起来“有点”正态分布。因此,特征值超过平均值6个标准差的观测值被视为异常值,我们希望将它们移除。
# 复制训练数据集
tmp_df = df_train_mod.copy()
# 计算每一列的均值和标准差
means = tmp_df[NUM_FEATS].mean()
std_devs = tmp_df[NUM_FEATS].std()
# 定义一个阈值,通常为距离平均值3个标准差
n_stds = 6
thresholds = n_stds * std_devs
# 检测异常值
outliers = (np.abs(tmp_df[NUM_FEATS] - means) > thresholds).any(axis=1)
# 输出检测到的异常值数量和阈值
print(f"检测到{sum(outliers)}个异常值,距离平均值超过{n_stds}个标准差...")
Detected 147 that are more than 6 SDs away from mean...
# 通过这行代码生成的布尔型序列可以用来过滤掉异常值
outliers_df = tmp_df[outliers]
# 用过滤掉异常值后的数据覆盖原始训练数据
df_train_mod = tmp_df[~outliers].reset_index(drop=True)
print(f"过滤掉异常值后的训练数据形状: {df_train_mod.shape}")
Train data shape after outlier removal: (8034, 55)
降维
根据Vilius Pėstininkas的好评,我们将尝试降低数据的维度。
待定!!!
# 导入必要的库
from sklearn.preprocessing import StandardScaler
from sklearn.ensemble import RandomForestClassifier
from sklearn.decomposition import PCA
from sklearn.manifold import TSNE
import plotly.express as px
import numpy as np
import pandas as pd
def tsne_with_feature_selection_and_pca(data, num_feats, target_column, n_components=2, top_n_features=10, pca_components=None):
"""
根据特征重要性选择前n个特征,可选应用PCA,然后使用t-SNE进行可视化。
参数:
data (DataFrame): 输入数据。
num_feats (list): 数值特征列名列表。
target_column (str): 目标列的名称。
n_components (int): t-SNE的维数(2或3)。默认为2。
top_n_features (int): 基于重要性选择的前n个特征数。默认为10。
pca_components (int or None): 应用t-SNE之前要保留的PCA组件数。如果为None,则不应用PCA。
"""
global label_encoder
# 标准化数值特征
scaler = StandardScaler()
numerical_data_scaled = scaler.fit_transform(data[num_feats])
# 随机森林获取特征重要性
rf = RandomForestClassifier(random_state=42)
rf.fit(numerical_data_scaled, data[target_column])
importances = rf.feature_importances_
# 选择前n个特征
indices = np.argsort(importances)[-top_n_features:]
selected_features = [num_feats[i] for i in indices]
# t-SNE所需的数据
tsne_data = numerical_data_scaled[:, indices]
# 可选应用PCA
if pca_components is not None and pca_components < len(selected_features):
pca = PCA(n_components=pca_components)
tsne_data = pca.fit_transform(tsne_data)
# 应用t-SNE
tsne = TSNE(n_components=n_components, learning_rate='auto', init='random', perplexity=30, random_state=42)
tsne_results = tsne.fit_transform(tsne_data)
# 创建t-SNE结果的DataFrame
tsne_df = pd.DataFrame(tsne_results, columns=[f'Component {i+1}' for i in range(n_components)])
tsne_df[target_column] = label_encoder.inverse_transform(data[target_column].values)
# 使用Plotly进行可视化
if n_components == 3:
fig = px.scatter_3d(tsne_df, x='Component 1', y='Component 2', z='Component 3', color=target_column)
else:
fig = px.scatter(tsne_df, x='Component 1', y='Component 2', color=target_column)
fig.update_layout(width=800, height=600)
fig.show()
df_train_red = df_train_mod
# tsne_with_feature_selection_and_pca(df_train_red, NUM_FEATS, LABEL, n_components=3, top_n_features=10, pca_components=None)
# 特征组合
# 所有特征
# df_train_pca = df_train_mod.drop([LABEL], axis=1)
# df_test_pca = df_test_mod
# 数值特征
df_train_pca = df_train_mod[NUM_FEATS]
df_test_pca = df_test_mod[NUM_FEATS]
# 一些特征,这里的特征是从之前的运行中迭代地获取的
# FEATS = ['Platelets', 'Copper', 'Alk_Phos', 'Diagnosis_Date', 'SGOT', 'Age', 'N_Days']
# PCA_FEATS = [c for c in df_train_mod.drop(LABEL, axis=1).columns.values if c not in FEATS]
# df_train_pca = df_train_mod[PCA_FEATS]
# df_test_pca = df_test_mod[PCA_FEATS]
# PCA降维
pca = PCA(n_components=10)
df_train_pca = pca.fit_transform(df_train_pca)
df_test_pca = pca.transform(df_test_pca)
# 输出每个主成分的解释方差
print(f"Explained variance per component: {np.round(pca.explained_variance_ratio_, 1)}")
Explained variance per component: [0.7 0.2 0.1 0. 0. 0. 0. 0. 0. 0. ]
eps_expl_var_treshold = 0 # 设置解释方差阈值为0
n_pcas = np.sum(np.round(pca.explained_variance_ratio_, 3) > eps_expl_var_treshold) # 计算满足解释方差阈值的主成分个数
pca_c_names = [f"PCA_{i}" for i in range(n_pcas)] # 创建主成分列的名称列表
print(f"PCA column names: {pca_c_names}") # 打印主成分列的名称
df_train_mod[pca_c_names] = df_train_pca[:,0:n_pcas] # 将训练数据集的主成分列添加到df_train_mod中
df_test_mod[pca_c_names] = df_test_pca[:,0:n_pcas] # 将测试数据集的主成分列添加到df_test_mod中
PCA column names: ['PCA_0', 'PCA_1', 'PCA_2', 'PCA_3']
存储特征工程结果
为了进行参数优化,我们将修改后的数据框存储起来,并在另一个笔记本中找到最佳参数。
# 将修改后的训练数据保存为CSV文件,文件名为"train_mod_v{DATA_VERSION}.csv"
df_train_mod.to_csv(f"train_mod_v{DATA_VERSION}.csv")
# 将修改后的测试数据保存为CSV文件,文件名为"test_mod_v{DATA_VERSION}.csv"
df_test_mod.to_csv(f"test_mod_v{DATA_VERSION}.csv")
模型选择
我们有几个潜在的模型可以用来解决这个分类问题
| 方法 | 描述 | 优点 | 缺点 | Python 库 |
|---|---|---|---|---|
| 逻辑回归 | 一种使用逻辑函数来建模二元依赖变量的统计模型。 | - 简单易懂 - 训练和预测速度快 - 适用于线性可分数据 | - 不适用于数据中的复杂关系 - 不能捕捉非线性模式 | scikit-learn, LogisticRegression |
| 决策树 | 一种非参数的监督学习方法,用于分类和回归。 | - 易于解释和可视化 - 可处理数字和分类数据 - 不需要特征缩放 | - 容易过拟合 - 数据微小变化时可能不稳定 - 偏向于主导类 | scikit-learn, DecisionTreeClassifier |
| 随机森林 | 一种集成学习方法(装袋),在训练期间构建多个决策树。 | - 比决策树更好地处理过拟合 - 适用于分类和回归 - 可处理具有更高维度的大数据集 | - 比决策树更复杂且不易解释 - 训练时间更长 - 可能需要更多内存 | scikit-learn, RandomForestClassifier |
| 提升 | (不是真正的模型,而是一种集成方法)依次应用弱模型,专注于先前模型的错误。包括AdaBoost,Gradient Boosting等。 | - 通常提供高准确性 - 处理不同类型的数据 - 提供特征重要性分数 | - 如果没有适当的调整,容易过拟合 - 训练时间长 - 与简单模型相比,解释性较差 | scikit-learn, XGBoost, LightGBM, CatBoost |
| 支持向量机(SVM) | 用于分类和回归的监督学习模型及其相关学习算法。使用“分离超平面”将特征空间分割。 | - 在高维空间中有效 - 在具有清晰(线性)分离边界的情况下工作良好 - 可为决策函数指定不同的核函数 | - 不适用于大型数据集 - 对噪声数据敏感 - 需要仔细选择核和正则化项 | scikit-learn, SVC |
| 朴素贝叶斯 | 一组基于应用贝叶斯定理的监督学习算法,假设每对特征之间具有条件独立性。 | - 简单快速 - 在分类输入变量方面表现良好 - 适用于大型数据集 | - 基于独立预测变量的假设,这很少是真实的 - 不适用于回归任务 | scikit-learn, GaussianNB, MultinomialNB |
| K最近邻(KNN) | 用于分类和回归的非参数方法。 | - 简单易实现 - 无需训练期 - 自然处理多类情况 | - 在大型数据集上速度较慢 - 对不相关特征和数据规模敏感 - 需要所有训练数据的内存 | scikit-learn, KNeighborsClassifier |
| 神经网络/深度学习 | 一组算法,松散地模仿人脑,旨在识别模式。 | - 非常强大和灵活 - 可以模拟复杂的非线性关系 - 适用于大型数据集和高维数据 | - 需要大量数据 - 计算密集 - 容易过拟合,需要调整 | tensorflow, keras, pytorch |
我们将运行上述模型的选择,并检查它们在训练和验证集上的性能。最优模型参数化尚未执行!
特征选择和参数调整
参数调整在另一个笔记本中完成。
我们不希望在我们的模型中使用所有的工程特征。因此,我们只选择潜在有趣的特征。
# 导入所需的库
from sklearn.feature_selection import RFECV
# 将训练数据集赋值给df_feat_sel_train
df_feat_sel_train = df_train_mod
# 创建特征选择模型
feat_sel_model = xgb.XGBClassifier()
# 创建递归特征消除对象
rfe = RFECV(estimator=feat_sel_model, min_features_to_select=1, step=1, n_jobs=-1, verbose=1)
# 在训练数据集上进行特征选择
rfe.fit(df_feat_sel_train.drop(LABEL, axis=1), df_feat_sel_train[LABEL])
# 打印特征排名
print("Feature Ranking: ", rfe.ranking_)
# 对训练数据集进行特征选择
rfe.transform(df_feat_sel_train.drop(LABEL, axis=1))
# 获取被选择的特征列
sel_feats = df_feat_sel_train.drop(LABEL, axis=1).columns[rfe.support_].values
# 打印被选择的特征列数量
print(len(sel_feats))
# 打印被选择的特征列列表
print(sel_feats.tolist())
# 导入statsmodels库
import statsmodels.api as sm
# 创建一个多项逻辑回归模型
feat_check_model = sm.MNLogit(df_train_mod[LABEL], df_train_mod[['Platelets', 'Copper', 'Alk_Phos', 'Diagnosis_Date', 'SGOT', 'Age'] + pca_c_names])
# 使用训练数据拟合模型
res = feat_check_model.fit()
# 打印模型的摘要信息
print(res.summary())
# 特征列表,包含了用于训练的特征
# All
# FEATS = df_train_mod.drop(LABEL, axis=1).columns.tolist()
# Some
FEATS = ['Platelets', 'Copper', 'Alk_Phos', 'Diagnosis_Date', 'SGOT', 'Age', 'N_Days', 'Cholesterol',
'Tryglicerides', 'Albumin', 'Bilirubin', 'Prothrombin', 'Symptom_Score', 'Stage', 'Drug',
'Hepatomegaly', 'Spiders', 'Sex', 'Edema_N', 'Edema_S', 'Edema_Y']
# FEATS = FEATS + pca_c_names
# FEATS = sel_feats.tolist()
# 打印特征数量和使用的特征
print(f"Number of feats: {len(FEATS)}")
print(f"Features used: {FEATS}")
# 本地optuna优化测试(交叉验证得分:0.4129666179319006),使用21个特征
xgb_params = {'objective': 'multi_logloss', 'early_stopping_rounds': 50, 'max_depth': 9, 'min_child_weight': 8, 'learning_rate': 0.0337716365315986, 'n_estimators': 733, 'subsample': 0.6927955384688348, 'colsample_bytree': 0.1234702658812108, 'reg_alpha': 0.18561628377665318, 'reg_lambda': 0.5565488299127089, 'random_state': 42}
Number of feats: 21
Features used: ['Platelets', 'Copper', 'Alk_Phos', 'Diagnosis_Date', 'SGOT', 'Age', 'N_Days', 'Cholesterol', 'Tryglicerides', 'Albumin', 'Bilirubin', 'Prothrombin', 'Symptom_Score', 'Stage', 'Drug', 'Hepatomegaly', 'Spiders', 'Sex', 'Edema_N', 'Edema_S', 'Edema_Y']
# 只使用选定的特征
df_train_final = df_train_mod[FEATS + [LABEL]] # 将训练数据集中的选定特征和标签列组合成新的数据集
df_test_final = df_test_mod[FEATS] # 将测试数据集中的选定特征提取出来作为新的数据集
模型选择
def validate_models(models: list[dict], data: pd.DataFrame, label=LABEL, n_splits=5, n_repeats=1, seed=43):
"""运行模型并在验证集上测试它们。最佳参数应从之前的运行中检索,例如GridSearchCV等。"""
# TODO: 模型字典应包含FEATS(因为应使用不同的FEATS)
train_scores, val_scores = {}, {}
pbar = tqdm(models) # 进度条
for model in pbar:
# 模型需要是字典(在元组之前)因为我需要一个可变数据类型
# 在最后插入平均验证分数
model_str = model["name"] # 模型名称
model_est = model["model"] # 模型估计器
model_feats = model["feats"] # 模型特征
pbar.set_description(f"Processing {model_str}...") # 进度条描述
train_scores[model_str] = [] # 训练分数
val_scores[model_str] = [] # 验证分数
# 在混合模型时,我认为应该删除种子
# -> 它们将在不同的数据集上进行训练
skf = RepeatedStratifiedKFold(n_splits=n_splits, n_repeats=n_repeats, random_state=seed) # 分层重复k折交叉验证
for i, (train_idx, val_idx) in enumerate(skf.split(data[model_feats], data[label])):
pbar.set_postfix_str(f"Fold {i+1}/{n_splits}") # 进度条后缀
X_train, y_train = data[model_feats].loc[train_idx], data[label].loc[train_idx] # 训练集
X_val, y_val = data[model_feats].loc[val_idx], data[label].loc[val_idx] # 验证集
if model_str in ["lgb_cl"]:
callbacks = [lgb.early_stopping(stopping_rounds=50), lgb.log_evaluation(period=0)]
model_est.fit(X_train, y_train, eval_set=[(X_val, y_val)], callbacks=callbacks) # 拟合模型
elif model_str in ["xgb_cl", "cat_cl"]:
model_est.fit(X_train, y_train, eval_set=[(X_val, y_val)], verbose=0) # 拟合模型
elif model_str in ["voting_clf"]:
pass # TODO: 找到解决方案
else:
model_est.fit(X_train, y_train) # 拟合模型
train_preds = model_est.predict_proba(X_train[model_feats]) # 训练集预测
valid_preds = model_est.predict_proba(X_val[model_feats]) # 验证集预测
train_score = log_loss(y_train, train_preds) # 训练集得分
val_score = log_loss(y_val, valid_preds) # 验证集得分
train_scores[model_str].append(train_score) # 训练集得分列表
val_scores[model_str].append(val_score) # 验证集得分列表
#print(f"{model_str} | Fold {i + 1} | " +
# f"Train log_loss: {round(train_score, 4)} | " +
# f"Valid log_loss: {round(val_score, 4)}")
model["avg_val_score"] = np.mean(val_scores[model_str]) # 平均验证分数
return models, pd.DataFrame(train_scores), pd.DataFrame(val_scores) # 返回模型、训练集得分和验证集得分的数据框
让我们在我们的“validate_models”函数中运行几个模型。
%%time
# 创建XGBoost分类器对象,并传入参数
xgb_cl = xgb.XGBClassifier(**xgb_params)
# 创建一个包含模型名称、模型对象和特征的字典列表
models = [
{"name": "xgb_cl", "model": xgb_cl, "feats": FEATS},
]
# 调用validate_models函数进行模型验证,并获取模型、训练分数和验证分数
models, train_scores, val_scores = validate_models(models=models,
data=df_train_final,
n_splits=10,
n_repeats=1)
Processing xgb_cl...: 100%|██████████| 1/1 [00:36<00:00, 36.19s/it, Fold 10/10]
CPU times: user 2min 19s, sys: 1.66 s, total: 2min 21s
Wall time: 36.2 s
让我们检查一下训练集和验证集上的表现。
# 创建一个大小为15x6的图形,并返回两个子图
fig, axes = plt.subplots(1, 2, figsize=(15, 6))
# 设置一些常量
eps = .05
hl = .39
# 计算最小和最大分数
min_score = train_scores.min().min()-eps
max_score = val_scores.max().max()+eps
# 计算刻度
def calculate_ticks(min_score, max_score, num_ticks=10):
return np.linspace(min_score, max_score, num_ticks)
ticks = calculate_ticks(min_score, max_score)
# 在第一个子图上绘制箱线图
_ = sns.boxplot(train_scores, ax=axes[0])
_ = axes[0].set_title('Train Scores')
_ = axes[0].set_ylim(min_score, max_score)
_ = axes[0].set_yticks(ticks)
_ = axes[0].yaxis.grid(True)
_ = axes[0].axhline(y=hl, color='r', linestyle='--', lw=.7)
_ = axes[0].text(-1, hl, f"{hl}", c="red")
# 在第二个子图上绘制箱线图
_ = sns.boxplot(val_scores, ax=axes[1])
_ = axes[1].set_title('Validation Scores')
_ = axes[1].set_ylim(min_score, max_score)
_ = axes[1].set_yticks(ticks)
_ = axes[1].yaxis.grid(True)
_ = axes[1].axhline(y=hl, color='r', linestyle='--', lw=.7)
_ = axes[1].text(-1, hl, f"{hl}", c="red")

# 将结果打印成DataFrame
model_res = pd.concat([train_scores.describe(), val_scores.describe()], axis=1)
model_res.columns = ['Train', 'Validation']
print(model_res)
# 将结果绘制成折线图
_ = sns.lineplot(pd.concat([train_scores, val_scores], keys=["Train Score", "Validation Score"], axis=1), markers=True, dashes=False)
plt.axhline(y=0.39, color='r', linestyle='--', lw=.5)
plt.title('训练集与验证集得分比较')
plt.xlabel('索引')
plt.ylabel('得分')
plt.show()
Train Validation
count 10.000000 10.000000
mean 0.255969 0.410636
std 0.019424 0.018420
min 0.225219 0.386856
25% 0.237610 0.394152
50% 0.265264 0.414089
75% 0.269775 0.422885
max 0.274766 0.442835

过采样,CL案例的训练和验证分数有所改善…然而,这是一种假象,因为我现在有更多的观察结果可以被正确分类…训练的验证集应该反映原始标签的分布!
从这里的讨论和Ravi的友好回应中,我遵循他的建议,不再对整个训练数据集重新拟合模型。
集成方法(不再使用)!
| 集成方法 | 描述 | 优点 | 缺点 | Python库 |
|---|---|---|---|---|
| 投票分类器 | 结合多个模型的预测结果。硬投票使用预测结果的众数,软投票使用平均概率。 | - 实现简单 - 减少方差,提高性能 - 有效地结合具有不同优势的模型 | - 受限于最佳个体模型 - 硬投票可能忽略概率估计 - 需要仔细选择要结合的模型 | scikit-learn (VotingClassifier) |
| 装袋 | 在数据的不同子集上训练相同的算法,然后对预测结果进行平均(例如随机森林)。 | - 减少方差;避免过拟合 - 对于决策树等不稳定模型有效 - 提高模型准确性而不增加复杂性 | - 模型可能变得非常相似,限制了效益 - 如果个体模型存在偏差,则效果较差 - 对于大型数据集而言,计算密集型 | scikit-learn (BaggingClassifier) |
| 堆叠 | 训练一个新模型来结合多个基模型的预测结果。请参见这里的示例。 | - 充分利用每个模型的优势 - 可以胜过单个模型 - 在选择基模型和元模型方面具有灵活性 | - 实现复杂;存在过拟合的风险 - 训练可能耗时 - 模型选择和调优可能具有挑战性 | scikit-learn (StackingClassifier) |
| 混合 | 使用加权平均或其他简单方法结合预测结果。 | - 比堆叠更简单;过拟合的风险较小 - 实现和运行快速 - 当模型具有类似性能时有效 | - 可能无法像堆叠那样捕捉复杂模式 - 在处理不同数据类型方面比堆叠或提升弱 - 需要良好的模型组合以实现有效混合 | 使用numpy,pandas手动实现 |
| 自定义集成方法 | 为特定问题开发定制的集成方法。 | - 定制化可以在特定任务上提高性能 - 灵活性以应对数据的独特方面 - 有可能胜过标准方法 | - 需要对问题和模型有深入的理解 - 开发和测试可能耗时 - 如果设计不当,存在过拟合的风险 | numpy,pandas,scikit-learn,tensorflow等 |
class MyAvgVoting(BaseEstimator, ClassifierMixin):
"""一个基本的投票方法,它只是对所有估计器的预测结果进行平均,并预测得票最高的类别。"""
def __init__(self, estimators, weighted=False):
self.estimators = estimators
# 是否根据验证分数进行加权平均
self.weighted = weighted
def fit(self, X, y):
for _, est in self.estimators:
est["model"].fit(X, y)
return self
def create_avg_prob_predictions(self, X):
predictions = np.array([est["model"].predict_proba(X) for est in self.estimators])
if self.weighted:
# 注意:我们需要验证分数的倒数,因为较低的值更好
weights = [{"name": est["name"], "value": 1/est["avg_val_score"]} for est in self.estimators]
print(f"Weights are:\n{pd.DataFrame(weights)}")
return np.average(predictions, axis=0, weights=[w["value"] for w in weights])
return np.average(predictions, axis=0)
def predict(self, X):
avg_predictions = self.create_avg_prob_predictions(X)
return np.argmax(avg_predictions, axis=1)
def predict_proba(self, X):
avg_predictions = self.create_avg_prob_predictions(X)
return avg_predictions
def score(self, X, y):
pass
voting_ests = models
voting_clf = MyAvgVoting(voting_ests, weighted=False)
# 不需要拟合
# 定义一个变量model_fin,用于存储models列表中第一个元素的'model'键对应的值
model_fin = models[0]['model']
模型分析
我们检查对训练数据的预测,并检查我们分类“状态”的准确性。
待定:进一步阐述…
# 预测模型
y_hat = model_fin.predict(df_train_final[FEATS])
# 生成混淆矩阵并显示
ConfusionMatrixDisplay.from_predictions(df_train_final[LABEL], y_hat, normalize='true', display_labels=label_encoder.classes_)
plt.show()

让我们分析来自XGBClassifier和LGBMClassifier的特征重要性。
# 导入必要的库
import pandas as pd
import matplotlib.pyplot as plt
# 创建Pandas Series来存储特征重要性
xgb_feat_importances = pd.Series(xgb_cl.feature_importances_, index=df_train_final[FEATS].columns)
# 在子图中绘制两个特征重要性
fig, axes = plt.subplots(1, 1, figsize=(15, 6))
# 绘制XGB特征重要性的水平条形图
xgb_feat_importances.nlargest(20).plot(kind='barh', title='XGB特征重要性')
# 调整子图布局
plt.tight_layout()
# 显示图形
plt.show()
# 打印排序后的XGBClassifier特征重要性的索引列表
print(f"总共排序后的XGBClassifier特征重要性: {xgb_feat_importances.nlargest(99).index.tolist()}")

Total sorted XGBClassifier importances: ['Edema_N', 'Edema_Y', 'Hepatomegaly', 'Symptom_Score', 'Spiders', 'Bilirubin', 'Stage', 'Edema_S', 'Prothrombin', 'N_Days', 'Sex', 'SGOT', 'Copper', 'Diagnosis_Date', 'Alk_Phos', 'Platelets', 'Cholesterol', 'Age', 'Albumin', 'Tryglicerides', 'Drug']
似乎当我们想要利用或结合这两种方法时,我们应该为每个模型使用不同的特征。
提交
# 预测测试集的标签概率
y_test_hat = model_fin.predict_proba(df_test_final[FEATS])
# 确认预测结果的形状为 (测试集样本数, 3)
assert y_test_hat.shape == (df_test.shape[0], 3)
# 导入必要的库
import pandas as pd
# 定义标签列表
submission_labels = ["Status_C", "Status_CL", "Status_D"]
# 创建一个DataFrame对象,包含id和预测结果
sub = pd.DataFrame(
{"id": test_IDs, **dict(zip(submission_labels, y_test_hat.T))}
)
# 打印前几行数据
sub.head()
| id | Status_C | Status_CL | Status_D | |
|---|---|---|---|---|
| 0 | 7905 | 0.323975 | 0.019388 | 0.656637 |
| 1 | 7906 | 0.407523 | 0.237383 | 0.355094 |
| 2 | 7907 | 0.033225 | 0.007383 | 0.959392 |
| 3 | 7908 | 0.969763 | 0.005476 | 0.024761 |
| 4 | 7909 | 0.930430 | 0.036615 | 0.032955 |
提交集合
# 读取其他的DataFrame
sub_list = [
sub,
#pd.read_csv('/kaggle/input/ps3e26-cirrhosis-survial-prediction-multiclass/submission.csv'),
#pd.read_csv('/kaggle/input/multi-class-prediction-of-cirrhosis-outcomes/submission.csv'),
#pd.read_csv('/kaggle/input/ps3e25-cirrhosis-multi-class-solution/submission.csv')
pd.read_csv('/kaggle/input/other-notebooks/Multi-Class Prediction of Cirrhosis Outcomes_V16.csv'),
pd.read_csv('/kaggle/input/other-notebooks/PS3E25 - Cirrhosis Multi-Class Solution_V10.csv'),
pd.read_csv('/kaggle/input/other-notebooks/PS3E26 Cirrhosis Survial Prediction Multiclass_V8.csv')
]
# 计算所有DataFrame的平均值
sub_ensemble = pd.concat(sub_list).groupby('id')[submission_labels].mean()
# 将列进行归一化,使其总和为1
sub_ensemble[submission_labels] = sub_ensemble[submission_labels].div(sub_ensemble[submission_labels].sum(axis=1), axis=0).fillna(0)
sub_ensemble = sub_ensemble.reset_index()
# 保存并显示结果
sub_ensemble.to_csv('submission.csv', index=False)
sub_ensemble.head()
| id | Status_C | Status_CL | Status_D | |
|---|---|---|---|---|
| 0 | 7905 | 0.455518 | 0.031246 | 0.513236 |
| 1 | 7906 | 0.450132 | 0.257913 | 0.291955 |
| 2 | 7907 | 0.029679 | 0.012259 | 0.958062 |
| 3 | 7908 | 0.970744 | 0.004791 | 0.024465 |
| 4 | 7909 | 0.857698 | 0.050979 | 0.091323 |

























 737
737











 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










