一、原理
OpenCV实现的贝叶斯分类器不是我们所熟悉的朴素贝叶斯分类器(Naïve Bayes Classifier),而是正态贝叶斯分类器(Normal Bayes Classifier),两者虽然英文名称很相似,但它们是不同的贝叶斯分类器。前者在使用上有一个限制条件,那就是变量的特征之间要相互独立,而后者没有这个苛刻的条件,因此它的适用范围更广。为了保持理论的系统性和完整性,我们还是先介绍朴素贝叶斯分类器,然后再介绍正态贝叶斯分类器。
1、朴素贝叶斯分类器
朴素贝叶斯分类器是一种基于贝叶斯理论的简单的概率分类器,而朴素的含义是指输入变量的特征属性之间具有很强的独立性。尽管这种朴素的设计和假设过于简单,但朴素贝叶斯分类器在许多复杂的实际情况下具有很好的表现,并且在综合性能上,该分类器要优于提升树(boosted trees)和随机森林(random forests)。
在许多实际应用中,对于朴素贝叶斯模型的参数估计往往使用的是极大似然法,因此我们可以这么认为,在不接受贝叶斯概率或不使用任何贝叶斯方法的前提下,我们仍然可以应用朴素贝叶斯模型对事物进行分类。
朴素贝叶斯分类器特别适用于输入变量的维数很高的情况,并且它只需要极少量的训练数据就可以估计出分类所需的参数。
抽象地说,朴素贝叶斯是一种条件概率模型:我们要对一个个体进行分类,该个体用代表n个特征(相互独立的变量)的n维向量表示,即x = (x1,…,xn)T,则分配给该个体的概率为:
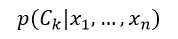 (1)
(1)
该式表示K个可能输出或分类中第k个分类的概率,Ck表示第k个响应输出,即分类结果。
如果个体的特征数量n很大,或者某个特征有大量的数值,则应用式1对个体进行分类是不可行。因此我们应用贝叶斯理论,把条件概率进行分解,使其更利于操作:
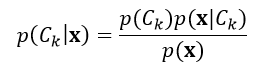 (2)
(2)
基于认识论的解释,概率是一种置信程度的度量。贝叶斯理论把某个事件在考虑证据之前和之后的置信程度关联了起来。回到式2,p(Ck)表示在不考虑个体x的情况下,第k个分类的概率,我们把它定义为先验概率,而p(Ck|x)表示在考虑个体x的情况下,第k个分类的概率,我们把它定义为后验概率,p(x| Ck)定义为似然度,p(x)定义为标准化常量。
在实际应用中,我们仅仅关心的是式2分式中的分子部分,这是因为分母部分不依赖于分类结果C,并且个体的特征属性Fi是给定的,所以分母是一个常数。
我们再来看式2中的分子部分,它是联合概率模型p(Ck,x1,…,xn)。基于链式法则,并重复应用条件概率的定义,这个联合概率模型可以重写为:
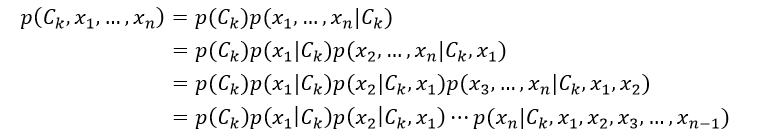 (3)
(3)
由于是“朴素”的贝叶斯,对于分类Ck来说,特征Fi是有条件的独立于特征Fj的,i≠j。因此,这意味着p(xi|Ck,xj) = p(xi|Ck),p(xi|Ck,xj,xq) =p(xi|Ck),p(xi|Ck,xj,xq,xl)= p(xi|Ck),以此类推,其中i≠j,q,l。则式2又可重写为:
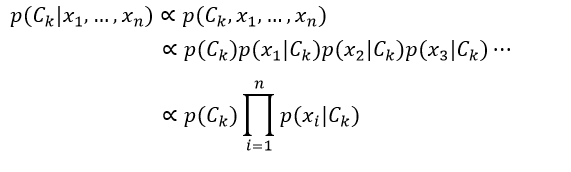 (4)
(4)
到目前为止我们得到了特征相互独立的朴素贝叶斯概率模型,我们利用该模型就可以得到具备决策规则的朴素贝叶斯分类器。应用得最普遍的决策规则是最大后验概率(MAP),即选择最可能的假设。则贝叶斯分类器所指定的分类结果为:
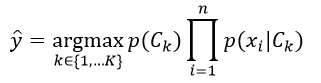 (5)
(5)
训练朴素贝叶斯分类器的任务是估计两组参数:先验概率p(Ck)和条件概率p(xi| Ck)。
我们先来计算条件概率p(xi | Ck),它分为两种情况:一种是样本数据都是离散的形式,即样本的特征是离散的形式;另一种是样本数据都是连续的数值形式,即样本的特征是数值的形式。
当样本的特征是离散的形式时,条件概率p(xi | Ck)的估计为
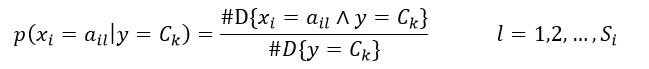 (6)
(6)
式中,ail表示第i个特征可能取的第l个值,Si表示第i个特征可能选取的所有值的数量,#D{X}表示在由N个训练样本构成的集合D中,满足条件X的样本的数量,因此分式中分母的含义是响应值为Ck的样本数,分子的含义是样本具有ail值并且响应值为Ck的数量。
式6给出了特征值为ail并且响应值为Ck的条件概率估计,该方法称为极大似然估计。但该方法可能会出现所要估计的概率值为0的情况,这时会影响到后验概率的计算结果,使分类产生偏差。采用平滑估计可以解决这个问题,即增加一个平滑系数λ,则条件概率为:
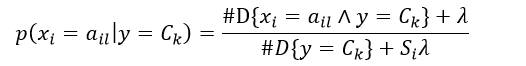 (7)
(7)
式中,λ≥ 0,显然λ = 0为极大似然估计,当λ = 1时,该平滑方法又称为拉普拉斯平滑。
当样本的特征是数值的形式时,条件概率的分布可以被认为是高斯分布,多项式分布或伯努利分布,则它们的朴素贝叶斯分别被称为高斯朴素贝叶斯,多项式朴素贝叶斯和伯努利朴素贝叶斯。在这里我们只介绍高斯朴素贝叶斯方法
高斯朴素贝叶斯方法是假设对每一个可能的响应值Ck,特征xi是满足高斯正态分布的,即
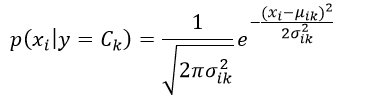 (8)
(8)
因此我们必须估计出该高斯分布的均值μik和方差σik2:
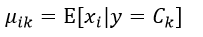 (9)
(9)
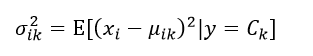 (10)
(10)
这里一共有2nK个参数,这些参数都需要独立的去估计。估计的方法仍然可以采用极大似然估计。均值μik的极大似然估计为:
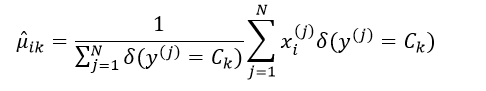 (11)
(11)
式中上标j表示全部N个训练样本中的第j个样本,函数δ(y = Ck)表示:
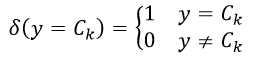 (12)
(12)
函数δ的作用就是选择那些响应值为Ck的训练样本。
方差σik2的极大似然估计为:
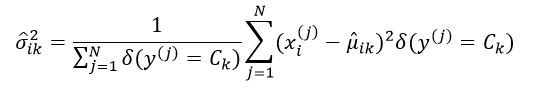 (13)
(13)
采用极大似然估计得到的方差是有偏估计,因此往往采用最小方差无偏估计(MVUE)来取代极大似然估计,则此时的σik2估计为:
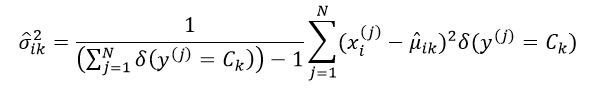 (14)
(14)
我们再来讨论先验概率。贝叶斯分类器只能处理分类问题,即分类结果C具有离散的K个值,因此先验概率p(Ck)的估计相对较简单。当我们已知所有的分类结果出现的概率都是相等的话(如投骰子),则先验概率p(Ck)为
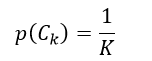 (15)
(15)
当我们仅考虑训练样本数据时,则先验概率p(Ck)为
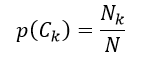 (16)
(16)
式中,N表示训练样本的数量,Nk表示分类结果为Ck的训练样本数量。式16这种极大似然估计仍然会有所要估计的概率值为0的情况,因此类似于式7改用平滑估计,得到先验概率为:
 (17)
(17)
如果我们应用高斯朴素贝叶斯分类器来预测样本,则先根据训练样本计算各个分类的先验概率,以及均值和方差,这样就得到了不同分类下的不同特征属性的高斯函数(式8),然后我们把预测样本数据带入这些不同的高斯函数中,得到不同分类的各个特征属性的似然度,最后把同一分类的先验概率和不同特征的似然度相乘(式5),哪个值大,该预测样本就属于该乘积所对应的分类。
2、正态贝叶斯分类器
下面我们来介绍正态贝叶斯分类器,该分类器只能处理特征值是连续数值的分类问题。
正态贝叶斯分类器认为每一个分类的所有特征属性(即特征向量)服从多变量正态高斯分布,即
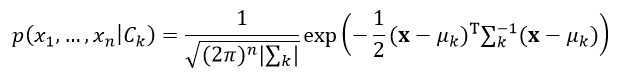 (18)
(18)
式中,μk表示第k个分类所对应的n维均值向量,|∑k|表示第k个分类所对应的n×n的协方差矩阵∑k的行列式的值。
因此该分类器认为特征属性之间不必是独立的,这要比朴素贝叶斯的适用条件要宽。则最终包括所有分类的整个分布函数是一个混合高斯分布,而每一个分类就是一个组件(component)。
由贝叶斯规则(式2)可知,后验概率正比于先验概率与似然度的乘积,但在有些情况下可以不考虑先验概率,如分类很少或维数较高等情况,即后验概率仅与似然度成正比,这样最大后验问题就变为了极大似然问题,这时我们只需要得到各个分类的似然度函数(式18),带入新的预测样本,哪个值大,该样本就属于该似然度函数对应的分类,即:
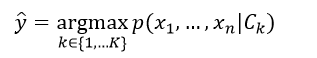 (19)
(19)
有时为了计算方法,我们可以把式18取对数,成为对数似然度函数,即
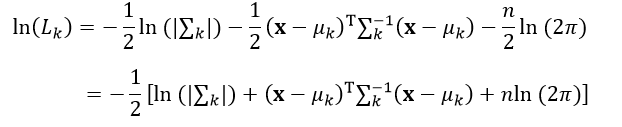 (20)
(20)
显然,求式20极大值问题可以转换为求式20中方括号内的极小值问题,其中最后一项nln(2π)是常数,可以不用计算。
为了计算式20,我们需要估计两组参数:均值向量μk和协方差矩阵∑k。
均值向量μk的第i个元素(即第i个特征)μki的极大似然估计为:
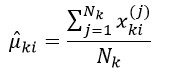 (21)
(21)
式中,xki(j)表示训练样本中属于分类k的第j个样本的第i个特征属性的值,则最终组成的n维(共有n个特征属性)均值向量μk的极大似然估计为:
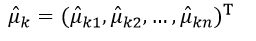 (22)
(22)
n×n的协方差矩阵∑k的无偏估计形式为:
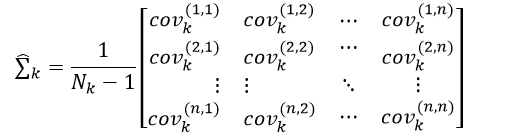 (23)
(23)
其中,第p行第q列元素covk(p,q)表示训练样本中第k个分类所组成的数据集合中,第p个特征与第q个特征的协方差,如果p等于q,则为方差,covk(p,q)为:
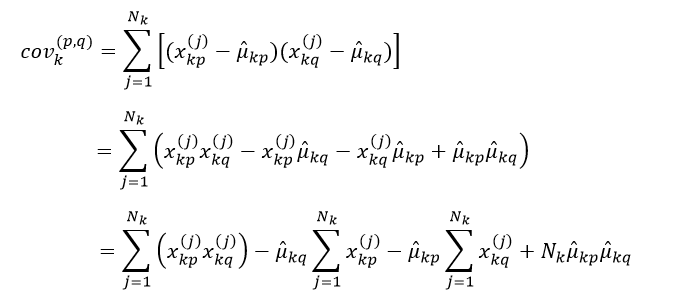 (24)
(24)
我们总结一下正态贝叶斯分类器的执行步骤。首先由训练样本数据估计每个分类的协方差矩阵(式23)和均值向量(式22),然后把这两组变量带入式20中,从而得到了每个分类的完整的对数似然函数。当需要预测样本时,把样本的特征属性值分别带入全部分类的对数似然函数中,最大对数似然函数对应的分类就是该样本的分类结果。
下面举一个例子,该例子是维基百科中英文条目Naive Bayes classifier所列举的例子。
下表是某国人体特征指标的一组统计资料:
| 序号 | 性别 | 身高(英尺) | 体重(磅) | 脚掌长(英寸) |
| 1 | 男 | 6 | 180 | 12 |
| 2 | 男 | 5.92 | 190 | 11 |
| 3 | 男 | 5.58 | 170 | 12 |
| 4 | 男 | 5.92 | 165 | 10 |
| 5 | 女 | 5 | 100 | 6 |
| 6 | 女 | 5.5 | 150 | 8 |
| 7 | 女 | 5.42 | 130 | 7 |
| 8 | 女 | 5.75 | 150 | 9 |
表中的样本一共有8个,分为男和女两类,各有样本数4个,即N男=4,N女=4。
由式21先计算男人的均值向量μ男:

则:
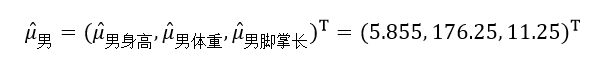
再计算女人的均值向量μ女:
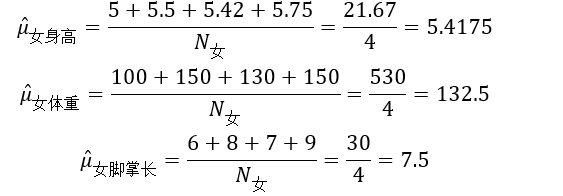
则:
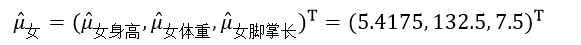
然后由式23和式24计算男人和女人的协方差矩阵,它们都是3×3的对称方阵。这里我们仅以男人的身高和体重为例,计算它们的协方差cov男(身高,体重):
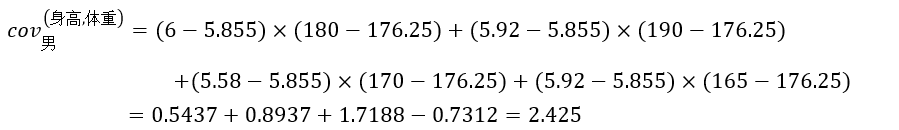
则最终的男人和女人的协方差矩阵分别为:
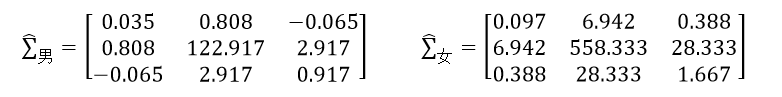
把得到的男人和女人的均值向量和协方差矩阵带入式20中,就得到两个分类的对数似然函数,由于该函数需要的是协方差矩阵的逆矩阵和行列式的值,所以还需要计算这两组值:
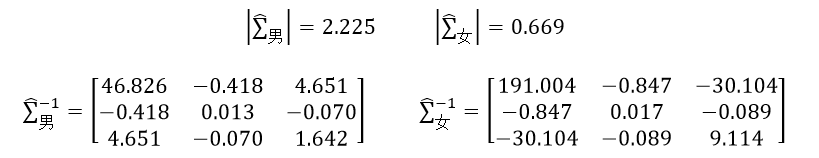
则对数似然函数为:
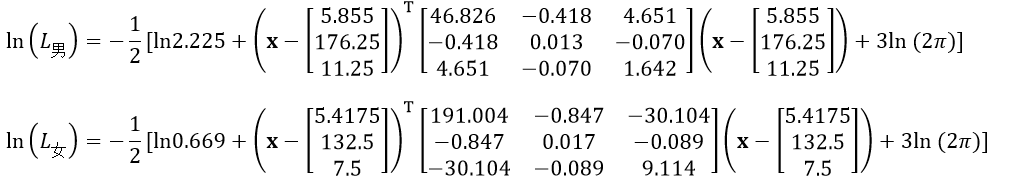
已知某人身高6英尺,体重130磅,脚掌长8英寸,我们利用前面的训练样本来预测该人是男还是女。预测样本向量x=(6, 130, 8)T,带入ln(L男)和ln(L女),则分别为-16.314和-28.730,显然ln(L男)大于ln(L女),所以这个人可能是男人。
这里要说明的是维基百科中应用的是高斯朴素贝叶斯分类器,即前面第一部分介绍的内容,它得到的结论是该人为女人。从中我们可以看出,高斯朴素贝叶斯分类器与正态贝叶斯分类器是不同的,正态贝叶斯分类器不在乎特征属性之间是否相互独立,个人认为正态贝叶斯分类器应该更准确一些。
在实际计算似然度函数时,应用奇异值分解会使程序更简洁。由于协方差矩阵∑k是对称矩阵,所以它的奇异值分解为
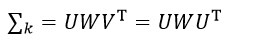 (25)
(25)
式中,W是由特征值组成的对角线矩阵,U是由特征向量组成的正交矩阵,具有U-1=UT的性质,则∑k的逆矩阵为
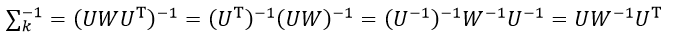 (26)
(26)
设D= x - μk,则(x-μk)T∑k-1(x-μk)为
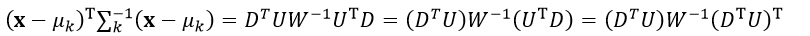 (27)
(27)
式中,DT是行向量,U是方阵,则DTU为行向量,设该行向量的元素为ai,(DTU)T则为列向量,因为W是对角线矩阵,设该矩阵对角线上的元素为wi,由矩阵的知识可知,W-1也是对角线矩阵,并且其对角线上的元素为1/wi,则式27改写为
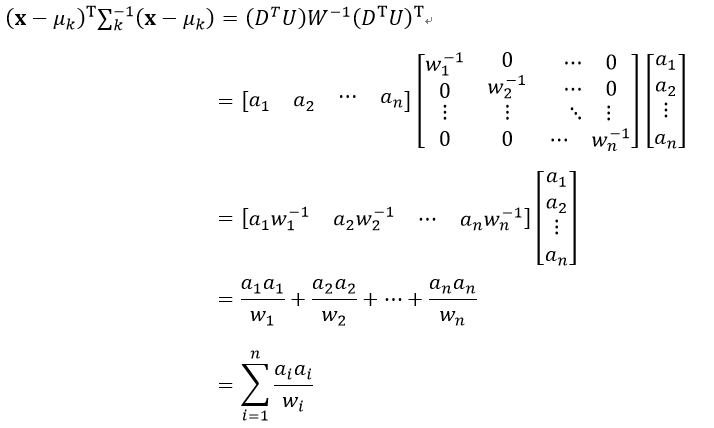 (28)
(28)
我们还注意到一个性质,那就是行列式的值等于该矩阵特征值的乘积,在前面我们已经得到了∑k的特征值为wi,则它的行列式值为:
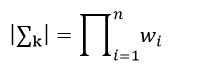 (29)
(29)
二、源码分析
下面我们就详细分析OpenCV中的贝叶斯分类器的源码。再次强调的是OpenCV实现的是正态贝叶斯分类器,不是朴素贝叶斯分类器。
CvNormalBayesClassifier类的缺省构造函数:
- CvNormalBayesClassifier::CvNormalBayesClassifier()
- {
- var_count = var_all = 0;
- var_idx = 0;
- cls_labels = 0;
- count = 0;
- sum = 0;
- productsum = 0;
- avg = 0;
- inv_eigen_values = 0;
- cov_rotate_mats = 0;
- c = 0;
- default_model_name = "my_nb";
- }
构建正态贝叶斯分类器的函数:
- bool CvNormalBayesClassifier::train( const CvMat* _train_data, const CvMat* _responses,
- const CvMat* _var_idx, const CvMat* _sample_idx, bool update )
- {
- const float min_variation = FLT_EPSILON;
- bool result = false;
- CvMat* responses = 0;
- const float** train_data = 0;
- CvMat* __cls_labels = 0;
- CvMat* __var_idx = 0;
- CvMat* cov = 0;
-
- CV_FUNCNAME( "CvNormalBayesClassifier::train" );
-
- __BEGIN__;
-
- int cls, nsamples = 0, _var_count = 0, _var_all = 0, nclasses = 0;
- int s, c1, c2;
- const int* responses_data;
-
- CV_CALL( cvPrepareTrainData( 0,
- _train_data, CV_ROW_SAMPLE, _responses, CV_VAR_CATEGORICAL,
- _var_idx, _sample_idx, false, &train_data,
- &nsamples, &_var_count, &_var_all, &responses,
- &__cls_labels, &__var_idx ));
-
- if( !update )
- {
- const size_t mat_size = sizeof(CvMat*);
- size_t data_size;
-
- clear();
-
- var_idx = __var_idx;
- cls_labels = __cls_labels;
- __var_idx = __cls_labels = 0;
- var_count = _var_count;
- var_all = _var_all;
-
- nclasses = cls_labels->cols;
- data_size = nclasses*6*mat_size;
-
- CV_CALL( count = (CvMat**)cvAlloc( data_size ));
- memset( count, 0, data_size );
-
-
-
- sum = count + nclasses;
-
- productsum = sum + nclasses;
-
- avg = productsum + nclasses;
-
- inv_eigen_values= avg + nclasses;
-
- cov_rotate_mats = inv_eigen_values + nclasses;
-
- CV_CALL( c = cvCreateMat( 1, nclasses, CV_64FC1 ));
-
- for( cls = 0; cls < nclasses; cls++ )
- {
-
- CV_CALL(count[cls] = cvCreateMat( 1, var_count, CV_32SC1 ));
-
- CV_CALL(sum[cls] = cvCreateMat( 1, var_count, CV_64FC1 ));
-
- CV_CALL(productsum[cls] = cvCreateMat( var_count, var_count, CV_64FC1 ));
-
- CV_CALL(avg[cls] = cvCreateMat( 1, var_count, CV_64FC1 ));
-
- CV_CALL(inv_eigen_values[cls] = cvCreateMat( 1, var_count, CV_64FC1 ));
-
- CV_CALL(cov_rotate_mats[cls] = cvCreateMat( var_count, var_count, CV_64FC1 ));
- CV_CALL(cvZero( count[cls] ));
- CV_CALL(cvZero( sum[cls] ));
- CV_CALL(cvZero( productsum[cls] ));
- CV_CALL(cvZero( avg[cls] ));
- CV_CALL(cvZero( inv_eigen_values[cls] ));
- CV_CALL(cvZero( cov_rotate_mats[cls] ));
- }
- }
- else
- {
-
- if( _var_count != var_count || _var_all != var_all || !((!_var_idx && !var_idx) ||
- (_var_idx && var_idx && cvNorm(_var_idx,var_idx,CV_C) < DBL_EPSILON)) )
- CV_ERROR( CV_StsBadArg,
- "The new training data is inconsistent with the original training data" );
-
- if( cls_labels->cols != __cls_labels->cols ||
- cvNorm(cls_labels, __cls_labels, CV_C) > DBL_EPSILON )
- CV_ERROR( CV_StsNotImplemented,
- "In the current implementation the new training data must have absolutely "
- "the same set of class labels as used in the original training data" );
-
- nclasses = cls_labels->cols;
- }
-
- responses_data = responses->data.i;
-
- CV_CALL( cov = cvCreateMat( _var_count, _var_count, CV_64FC1 ));
-
-
-
- for( s = 0; s < nsamples; s++ )
- {
- cls = responses_data[s];
-
- int* count_data = count[cls]->data.i;
- double* sum_data = sum[cls]->data.db;
- double* prod_data = productsum[cls]->data.db;
- const float* train_vec = train_data[s];
-
- for( c1 = 0; c1 < _var_count; c1++, prod_data += _var_count )
- {
-
- double val1 = train_vec[c1];
- sum_data[c1] += val1;
- count_data[c1]++;
-
- for( c2 = c1; c2 < _var_count; c2++ )
- prod_data[c2] += train_vec[c2]*val1;
- }
- }
- cvReleaseMat( &responses );
- responses = 0;
-
-
-
- for( cls = 0; cls < nclasses; cls++ )
- {
- double det = 1;
- int i, j;
- CvMat* w = inv_eigen_values[cls];
-
- int* count_data = count[cls]->data.i;
- double* avg_data = avg[cls]->data.db;
- double* sum1 = sum[cls]->data.db;
-
- cvCompleteSymm( productsum[cls], 0 );
-
- for( j = 0; j < _var_count; j++ )
- {
- int n = count_data[j];
- avg_data[j] = n ? sum1[j] / n : 0.;
- }
-
- count_data = count[cls]->data.i;
- avg_data = avg[cls]->data.db;
- sum1 = sum[cls]->data.db;
-
- for( i = 0; i < _var_count; i++ )
- {
- double* avg2_data = avg[cls]->data.db;
-
- double* sum2 = sum[cls]->data.db;
-
- double* prod_data = productsum[cls]->data.db + i*_var_count;
- double* cov_data = cov->data.db + i*_var_count;
- double s1val = sum1[i];
- double avg1 = avg_data[i];
- int _count = count_data[i];
-
- for( j = 0; j <= i; j++ )
- {
- double avg2 = avg2_data[j];
-
- double cov_val = prod_data[j] - avg1 * sum2[j] - avg2 * s1val + avg1 * avg2 * _count;
-
- cov_val = (_count > 1) ? cov_val / (_count - 1) : cov_val;
- cov_data[j] = cov_val;
- }
- }
-
- CV_CALL( cvCompleteSymm( cov, 1 ));
-
- CV_CALL( cvSVD( cov, w, cov_rotate_mats[cls], 0, CV_SVD_U_T ));
-
- CV_CALL( cvMaxS( w, min_variation, w ));
-
- for( j = 0; j < _var_count; j++ )
- det *= w->data.db[j];
-
- CV_CALL( cvDiv( NULL, w, w ));
-
- c->data.db[cls] = det > 0 ? log(det) : -700;
- }
-
- result = true;
-
- __END__;
-
- if( !result || cvGetErrStatus() < 0 )
- clear();
-
- cvReleaseMat( &cov );
- cvReleaseMat( &__cls_labels );
- cvReleaseMat( &__var_idx );
- cvFree( &train_data );
-
- return result;
- }
正态贝叶斯分类器的预测函数predict可以对一个样本进行预测,也可以同时对多个样本进行预测,但预测样本不能缺失任何一个特征属性。当只预测一个样本时,该函数的返回值就是预测分类结果。当预测多个样本是,样本以行的形式存储在参数矩阵samples中,而返回的预测结果存储在参数向量results中,因此这种情况results必须被定义。
- float CvNormalBayesClassifier::predict( const CvMat* samples, CvMat* results ) const
- {
- float value = 0;
-
- if( !CV_IS_MAT(samples) || CV_MAT_TYPE(samples->type) != CV_32FC1 || samples->cols != var_all )
- CV_Error( CV_StsBadArg,
- "The input samples must be 32f matrix with the number of columns = var_all" );
-
- if( samples->rows > 1 && !results )
- CV_Error( CV_StsNullPtr,
- "When the number of input samples is >1, the output vector of results must be passed" );
-
- if( results )
- {
- if( !CV_IS_MAT(results) || (CV_MAT_TYPE(results->type) != CV_32FC1 &&
- CV_MAT_TYPE(results->type) != CV_32SC1) ||
- (results->cols != 1 && results->rows != 1) ||
- results->cols + results->rows - 1 != samples->rows )
- CV_Error( CV_StsBadArg, "The output array must be integer or floating-point vector "
- "with the number of elements = number of rows in the input matrix" );
- }
-
- const int* vidx = var_idx ? var_idx->data.i : 0;
-
- cv::parallel_for_(cv::Range(0, samples->rows),
- predict_body(c, cov_rotate_mats, inv_eigen_values, avg, samples,
- vidx, cls_labels, results, &value, var_count));
-
- return value;
- }
预测样本的主体结构:
- struct predict_body : cv::ParallelLoopBody {
- predict_body(CvMat* _c, CvMat** _cov_rotate_mats, CvMat** _inv_eigen_values, CvMat** _avg,
- const CvMat* _samples, const int* _vidx, CvMat* _cls_labels,
- CvMat* _results, float* _value, int _var_count1
- )
- {
- c = _c;
- cov_rotate_mats = _cov_rotate_mats;
- inv_eigen_values = _inv_eigen_values;
- avg = _avg;
- samples = _samples;
- vidx = _vidx;
- cls_labels = _cls_labels;
- results = _results;
- value = _value;
- var_count1 = _var_count1;
- }
-
- CvMat* c;
- CvMat** cov_rotate_mats;
- CvMat** inv_eigen_values;
- CvMat** avg;
- const CvMat* samples;
- const int* vidx;
- CvMat* cls_labels;
-
- CvMat* results;
- float* value;
- int var_count1;
-
- void operator()( const cv::Range& range ) const
- {
-
- int cls = -1;
- int rtype = 0, rstep = 0;
- int nclasses = cls_labels->cols;
- int _var_count = avg[0]->cols;
-
- if (results)
- {
- rtype = CV_MAT_TYPE(results->type);
-
- rstep = CV_IS_MAT_CONT(results->type) ? 1 : results->step/CV_ELEM_SIZE(rtype);
- }
-
- cv::AutoBuffer<double> buffer(nclasses + var_count1);
-
- CvMat diff = cvMat( 1, var_count1, CV_64FC1, &buffer[0] );
-
- for(int k = range.start; k < range.end; k += 1 )
- {
- int ival;
- double opt = FLT_MAX;
-
- for(int i = 0; i < nclasses; i++ )
- {
-
- double cur = c->data.db[i];
- CvMat* u = cov_rotate_mats[i];
- CvMat* w = inv_eigen_values[i];
-
- const double* avg_data = avg[i]->data.db;
-
- const float* x = (const float*)(samples->data.ptr + samples->step*k);
-
-
-
- for(int j = 0; j < _var_count; j++ )
- diff.data.db[j] = avg_data[j] - x[vidx ? vidx[j] : j];
-
- cvGEMM( &diff, u, 1, 0, 0, &diff, CV_GEMM_B_T );
-
- for(int j = 0; j < _var_count; j++ )
- {
- double d = diff.data.db[j];
-
- cur += d*d*w->data.db[j];
- }
-
- if( cur < opt )
- {
- cls = i;
- opt = cur;
- }
-
- }
-
- ival = cls_labels->data.i[cls];
-
- if( results )
- {
- if( rtype == CV_32SC1 )
- results->data.i[k*rstep] = ival;
- else
- results->data.fl[k*rstep] = (float)ival;
- }
-
- if( k == 0 )
- *value = (float)ival;
- }
- }
- };
三、应用实例
我们还是以前面预测男人和女人为例,应用程序实现预测:
- #include "opencv2/core/core.hpp"
- #include "opencv2/highgui/highgui.hpp"
- #include "opencv2/imgproc/imgproc.hpp"
- #include "opencv2/ml/ml.hpp"
-
- #include <iostream>
- using namespace cv;
- using namespace std;
-
- int main( int argc, char** argv )
- {
- float trainingData[8][3] = { {6, 180, 12}, {5.92, 190, 11}, {5.58, 170, 12}, {5.92, 165, 10},
- {5, 100, 6}, {5.5, 150, 8},{5.42, 130, 7}, {5.75, 150, 9}};
- Mat trainingDataMat(8, 3, CV_32FC1, trainingData);
-
- float responses[8] = {'M', 'M', 'M', 'M', 'F', 'F', 'F', 'F'};
- Mat responsesMat(8, 1, CV_32FC1, responses);
-
- CvNormalBayesClassifier nbc;
- nbc.train(trainingDataMat, responsesMat);
-
- float myData[3] = {6, 130, 8};
- Mat myDataMat(1, 3, CV_32FC1, myData);
- float r = nbc.predict( myDataMat );
-
- cout<<endl<<"result: "<<(char)r<<endl;
-
- return 0;
-
- }
得到的结果为:
result: M
与我们前面的计算结果一致。
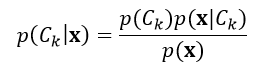
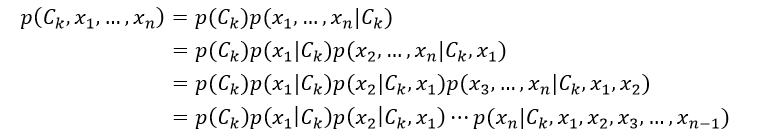
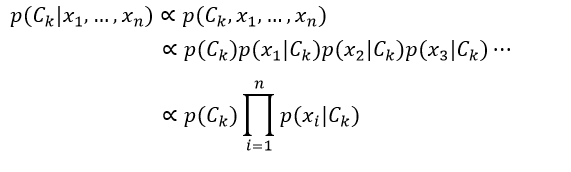
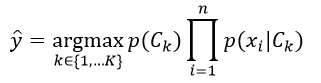
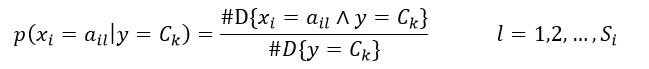
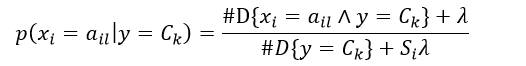
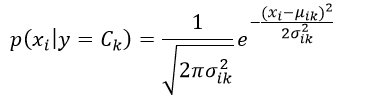
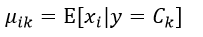
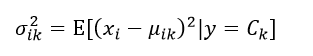
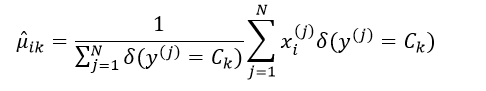
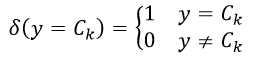
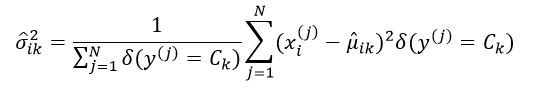
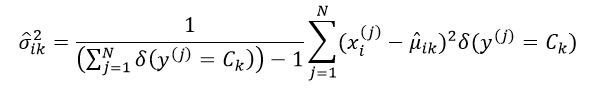
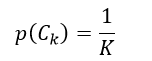
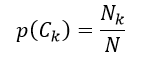

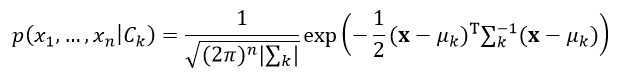
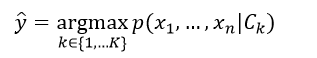
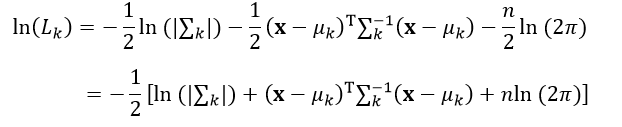
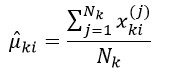
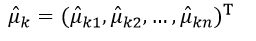
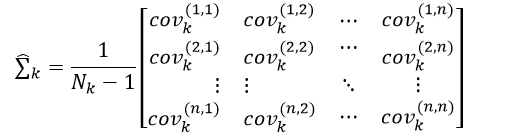
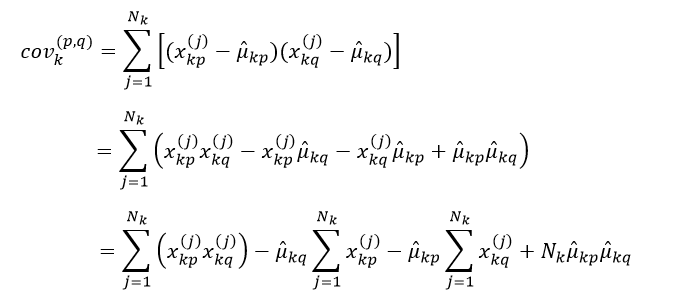
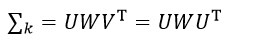
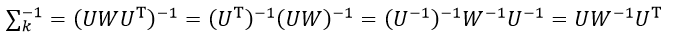
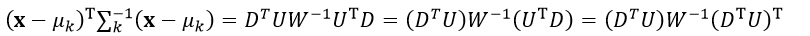
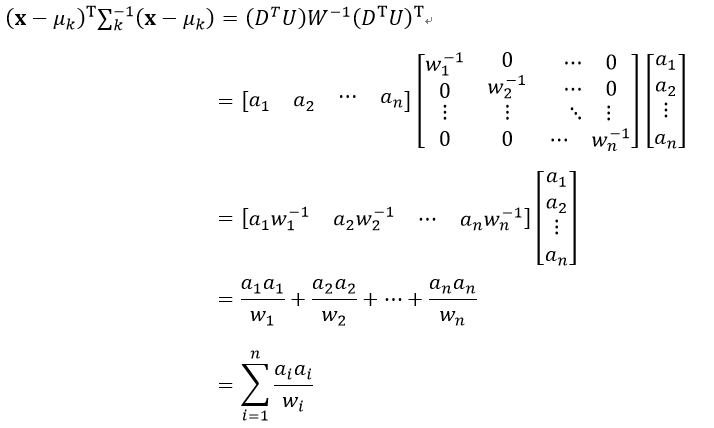
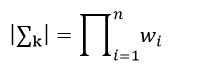









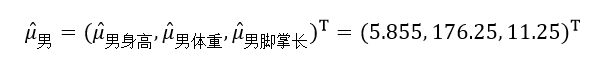
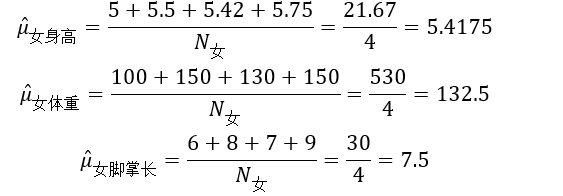
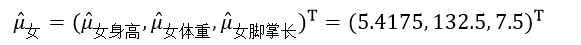
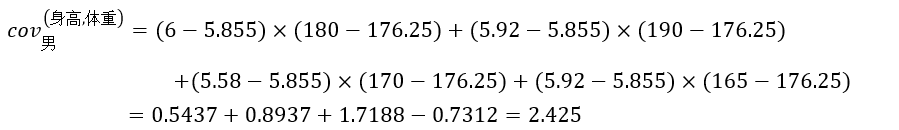
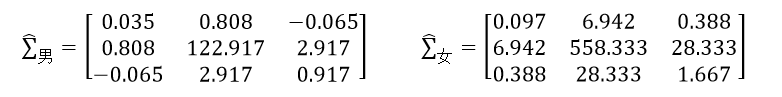
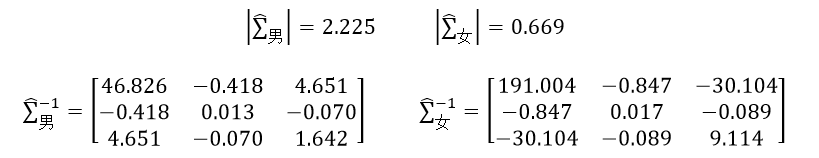















 957
957

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








