一、brat的安装
参考同伴的csdn创新实训-BRAT的安装-CSDN博客
在官网下载好了brat的安装包,在ubuntu中解压后利用解压包中的install脚本即可实现安装,在设定好自己的用户名密码及邮箱后,导入自己的数据,并导入与小组讨论得出的配置文件,便可实现brat的标注功能。现将配置文件放在此处。
注意:配置文件要和自己的数据在同一层目录下,如下图所示:
命名为:annotation.conf
该标注实体中的具体内涵参考创新实训-BRAT使用-CSDN博客,实体内容由小组讨论得出,并且更加适用于RCT随机对照试验。
[entities]
total-partcipants
intervention-participants
control-participants
age
eligibility
condition
location
ethinicity
intervention
control
outcome
outcome-Measure
iv-bin-abs
cv-bin-abs
iv-bin-percent
cv-bin-percent
iv-cont-mean
cv-cont-mean
iv-cont-median
cv-cont-median
iv-cont-sd
cv-cont-sd
此外,我们还需要在该目录的上一层中利用指令:find 文件夹名 -name '*.txt'|sed -e 's|\.txt|.ann|g'|xargs touch实现每个.ann文件的创建,在一切上述工作都完成后,即可进行brat的标注工作。
二、数据标注(集体分工合作)
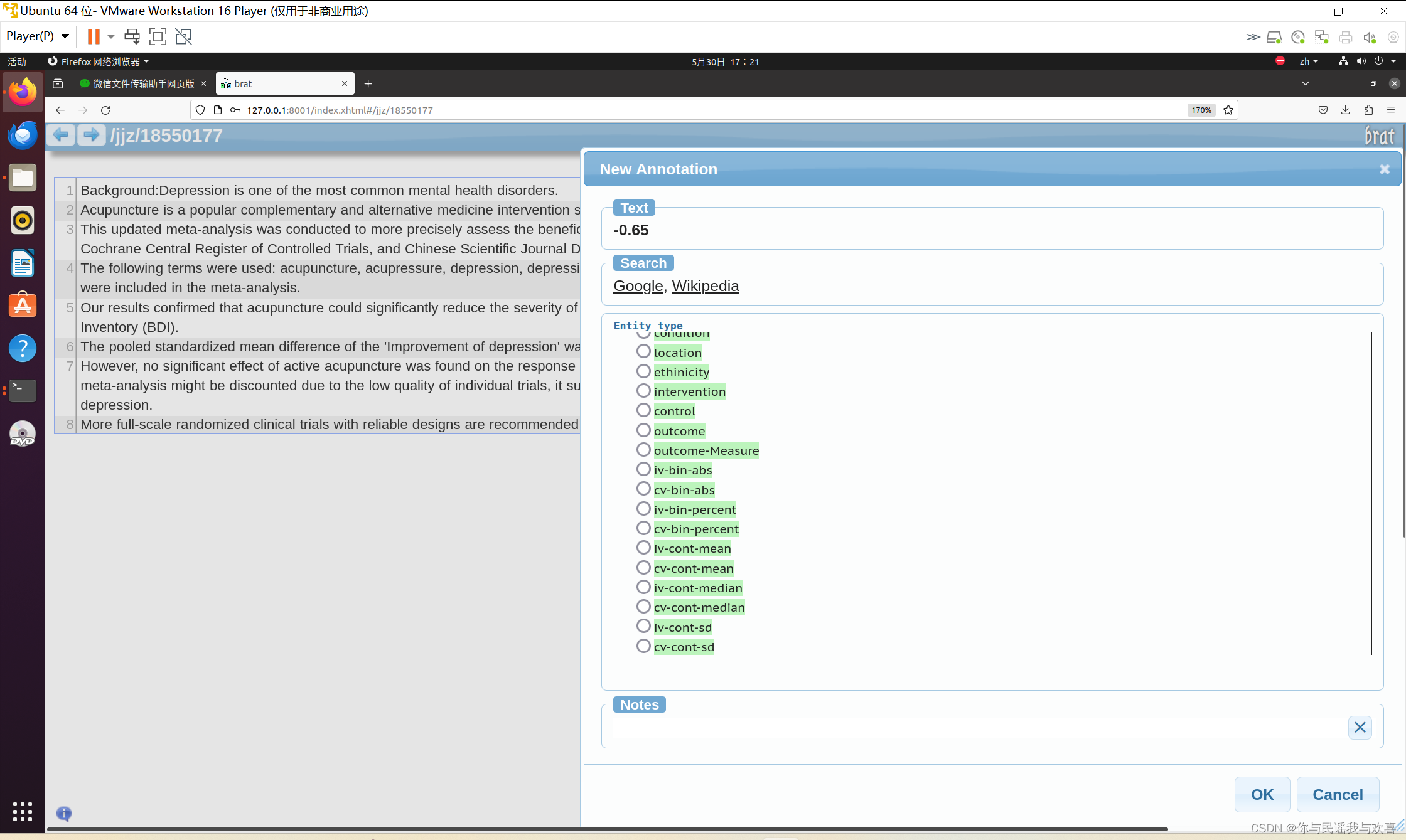
数据标注的具体页面如下图所示,利用鼠标在指定地方划出,便可以选择需要的实体类型了。本人负责标注了30篇,共标注了180篇。

图片展示:
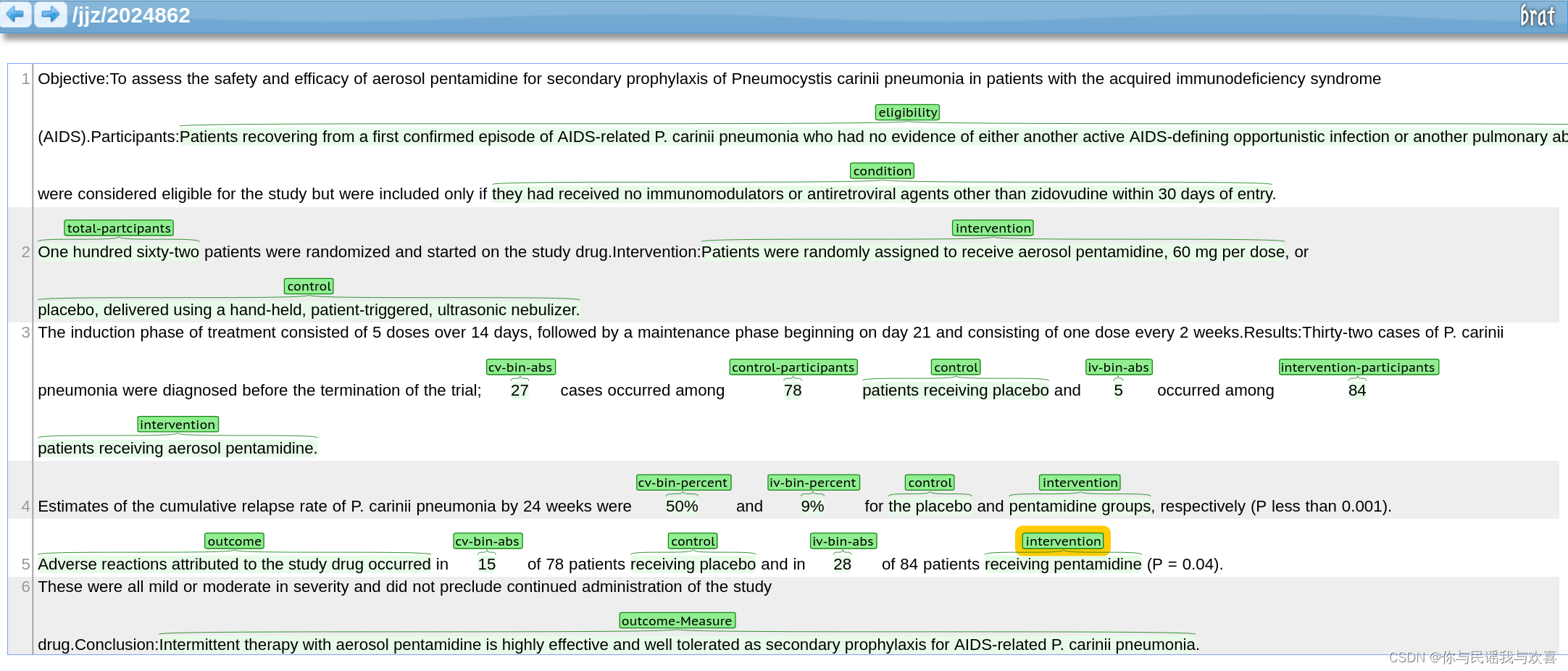
标注结果展示:
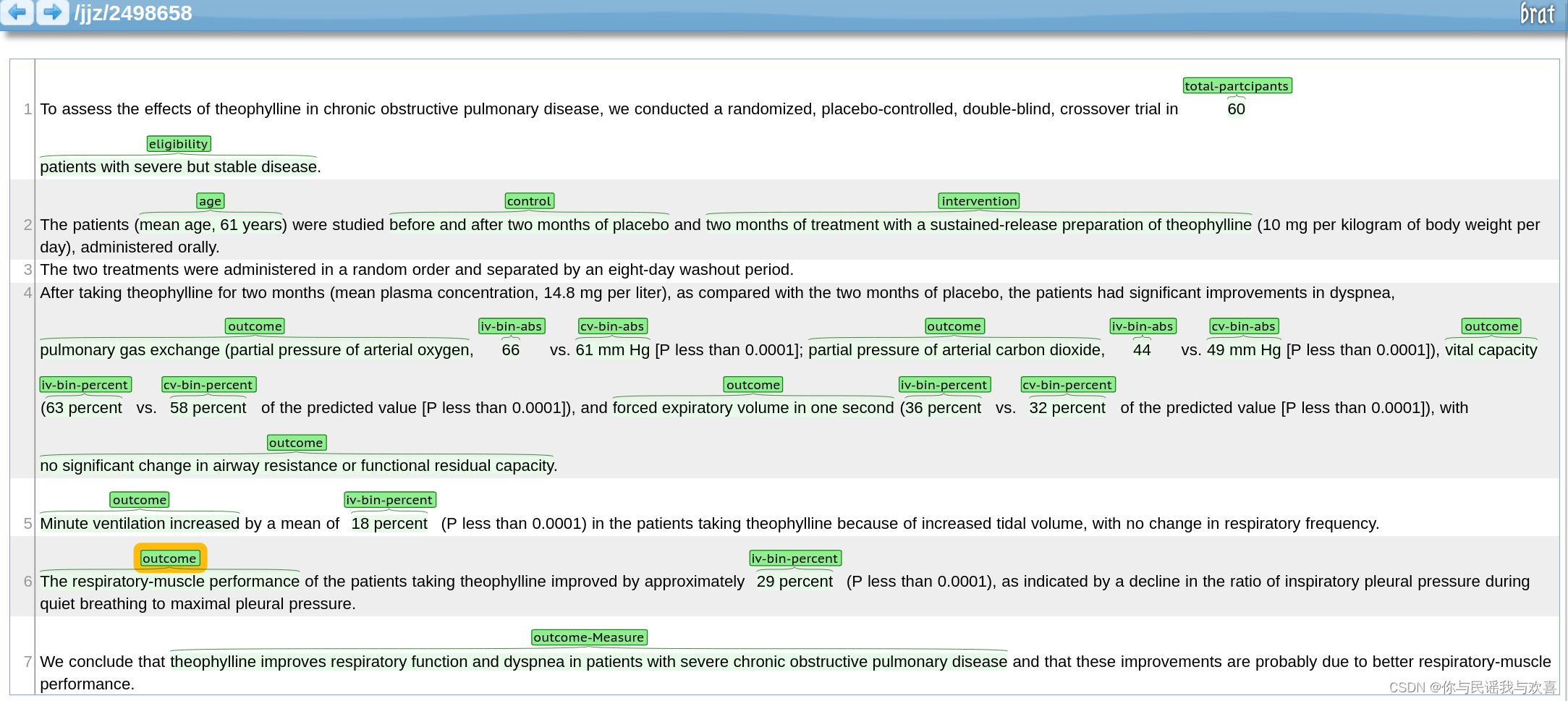
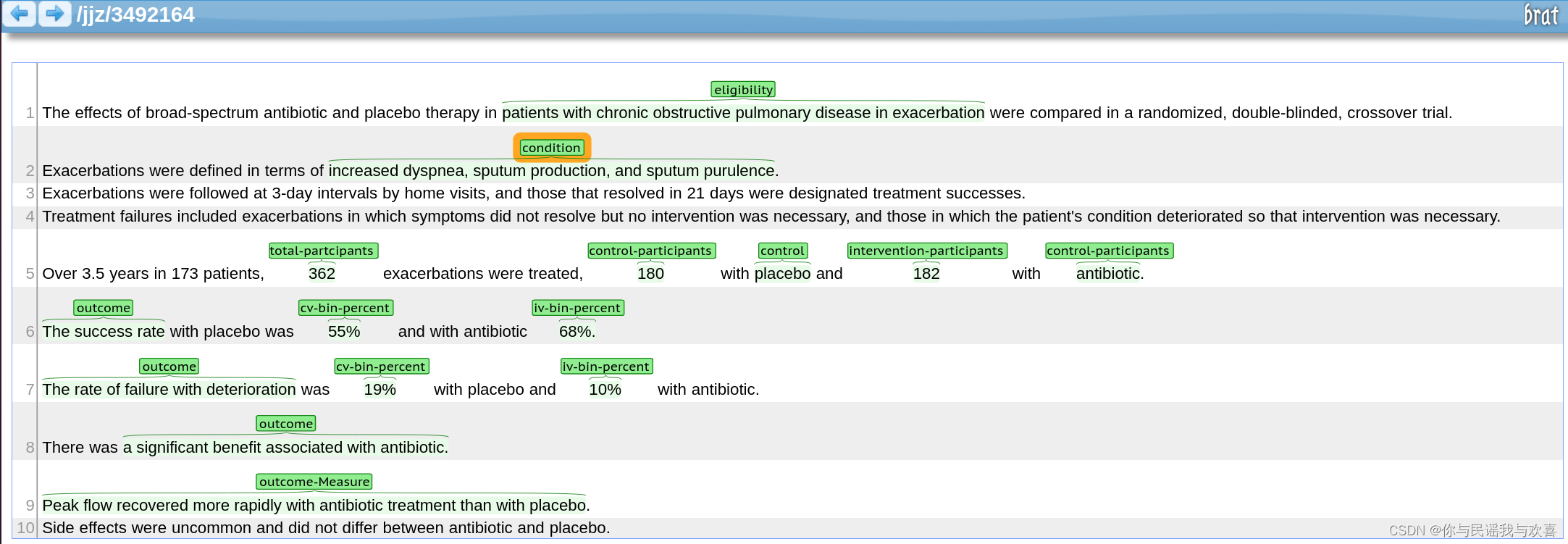
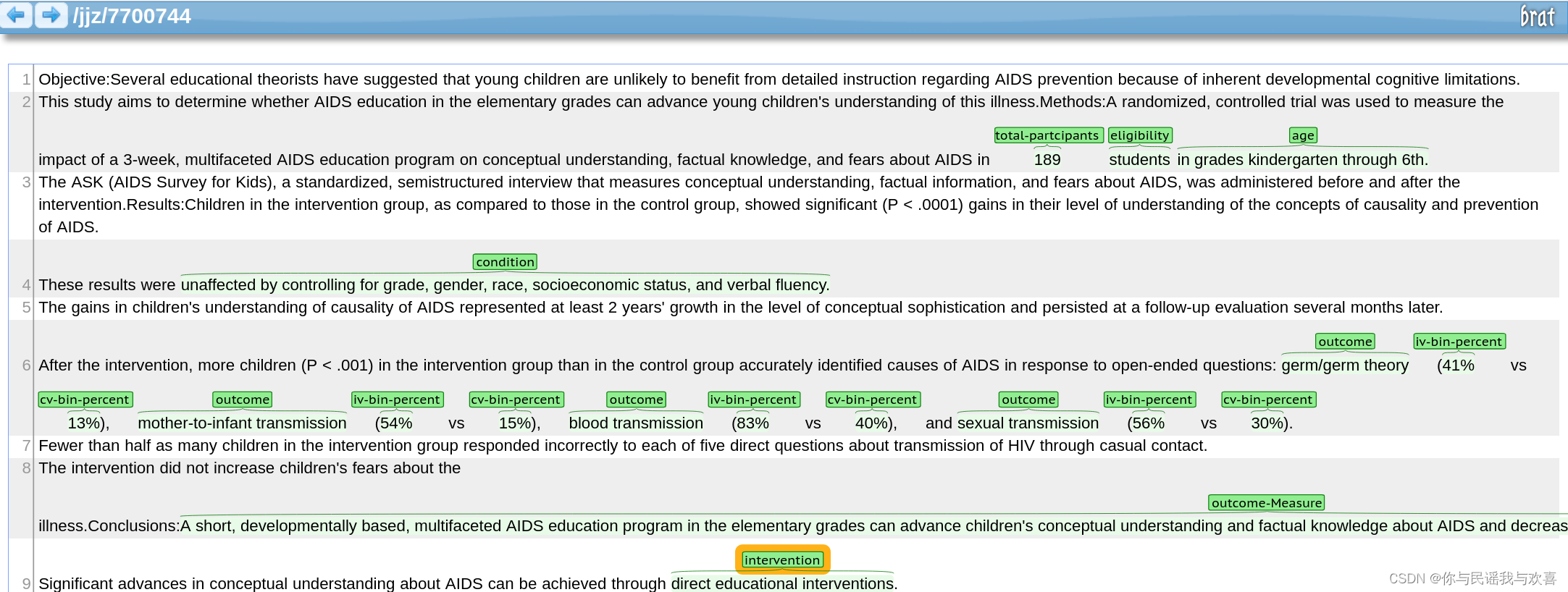
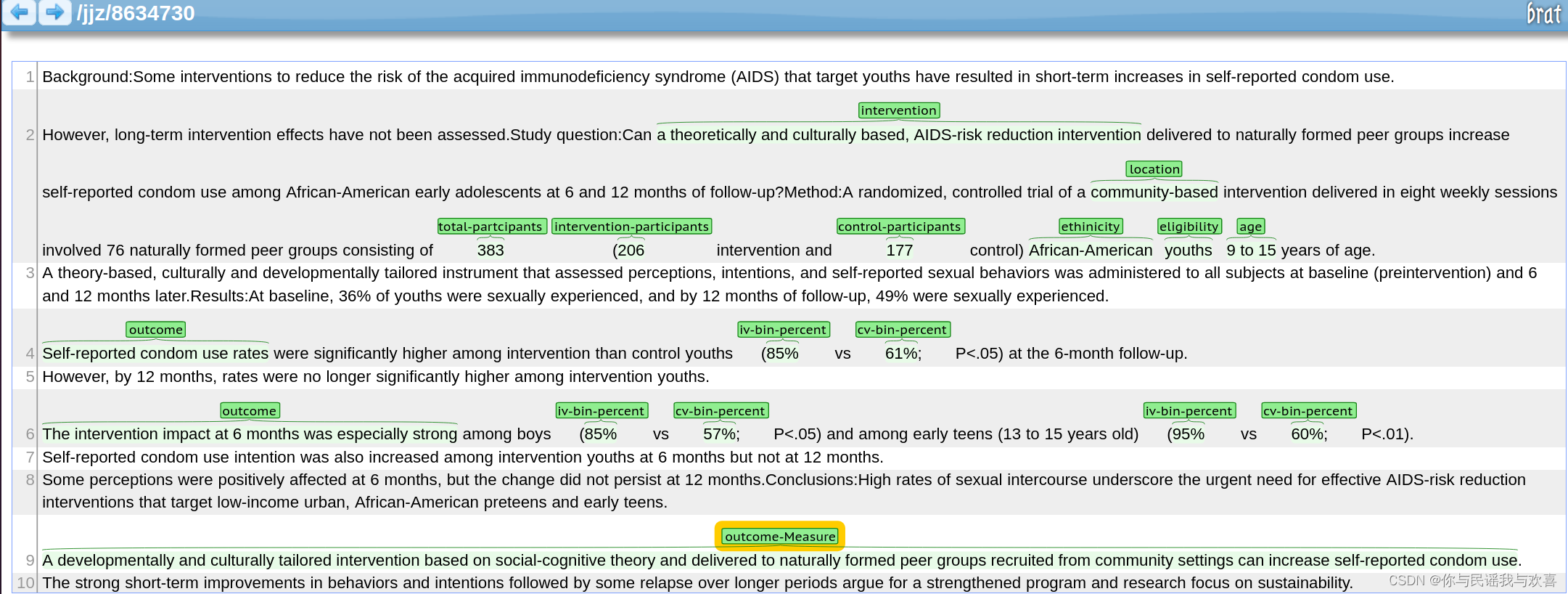
标注结果总览:
三、配置文件思考及后续优化
我们的配置文件仅设置了实体,对于信息提取这方面,能够方便模型的优化,但是当涉及到信息匹配及表格化输出时,还有待我们思考,后续会着重考虑与解决这个问题。





















 3592
3592

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








