
非小细胞肺癌(NSCLC)是全球肺癌发病率的80-85%,是最常见的恶性肿瘤类型。非小细胞肺癌最常见的组织学亚型是肺腺癌(LUAD),其次是肺鳞状细胞癌(LUSC),LUAD占非小细胞肺癌病例的40%以上。尽管使用了尖端技术、策略和治疗方法来治疗肺癌,但只有17%的肺癌患者能够存活五年。因此,作者迫切需要开发一种精确预测患者生存的方法
1. 胶原风险模型的构建
为了确定与LUAD预后相关的胶原家族蛋白,并找出影响LUAD发生和进展的关键因素,作者从TCGA数据库下载了598个LUAD样本的表达谱。本研究的标准是FDR < 0.01,| log (fold change) | > 1。共有44个胶原家族蛋白被纳入TCGA-LUAD转录组数据。根据筛选标准,在LUAD患者中有5542个基因显著差异表达,其中包括26个胶原家族成员。胶原家族26个差异表达基因的表达模式和特征在火山图和热图中呈现,其中5个胶原家族基因下调,21个胶原家族基因上调(图1A、B)。根据单变量Cox回归分析,胶原家族中的6个差异表达基因与OS显著相关(图1C)。通过使用LASSO-Cox回归分析,进一步研究了这些基因。通过Lasso分析确定了5个最重要的基因,分别是COL1A1、COL4A3、COL5A1、COL11A1和COL22A1(图1D、E)。根据多元Cox分析结果(P < 0.05)(图1F),COL4A3和COL22A1被证明是独立的预后风险变量。
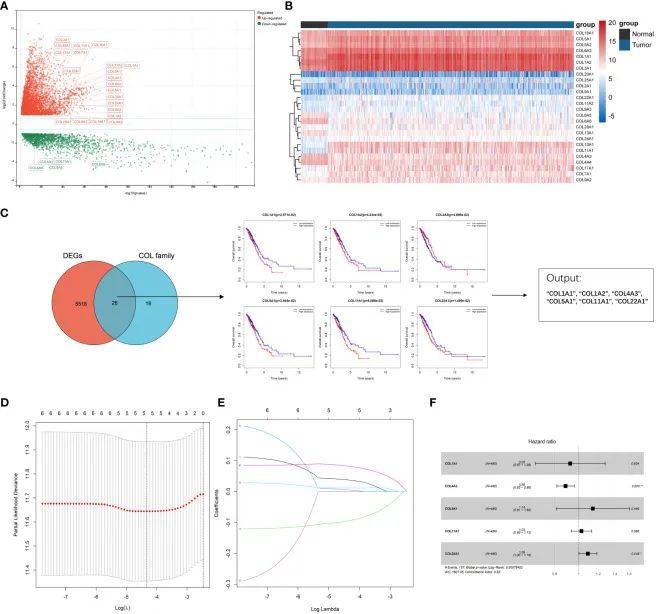
图1 在TCGA-LUAD中鉴定预后胶原家族基因并构建Collagen-Risk模型
2. 在TCGA-LUAD中鉴定预后胶原家族基因并构建Collagen-Risk模型
根据风险评分的中位数值,将所有LUAD患者分为高风险组和低风险组。散点图用于展示风险评分的分布以及风险评分与生存状态之间的关系,结果显示高风险组的死亡率较高(图2A)。在LUAD中,图2B比较了高风险组和低风险组中五个胶原家族基因的表达情况。尽管COL4A3在低风险组中的表达较高,但COL5A1、COL1A1、COL1A2和COL22A在高风险组中的表达上调。使用K-M生存分析比较了高风险组和低风险组的OS。根据研究结果,高风险组的预后较差(P=0.0019)(图2C)。为了更好地探索Collagen-Risk评分在LUAD不同病理分期中的预后意义,作者将LUAD分为早期和晚期,其中I期和II期为早期,III期和IV期为晚期。在早期阶段,作者发现胶原风险评分是OS的可靠指标,高风险组的患者预后明显较低风险组差。(p = 0.01) (图2D)。在另一个亚组分析中,高风险组的患者在晚期阶段的OS较低风险组更短,但差异在统计学上不显著(P=0.05) (图2E)。
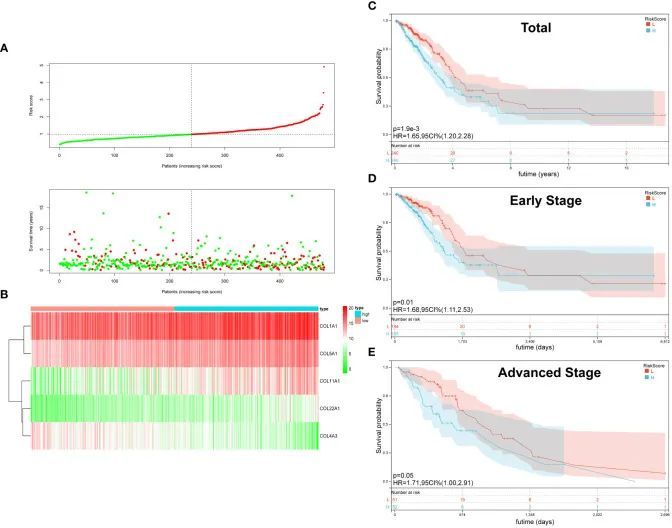
图2 胶原风险模型的特征和预测意义
3. 胶原风险模型的生存分析
首先,作者分析了不同临床特征的患者中胶原风险评分的分布情况,包括性别、病理分期和TNM分期。作者发现,N分期和病理分期较高的患者具有较高的风险评分(N 1 vs N 0,p < 0.05;N 2-3 vs N 0,p < 0.05;II期 vs I期,p < 0.01;III/IV期 vs I期,p < 0.01)。男性和女性患者之间的风险评分没有显著差异,M或T分期的患者之间也没有显著差异(P > 0.05)(图3A)。
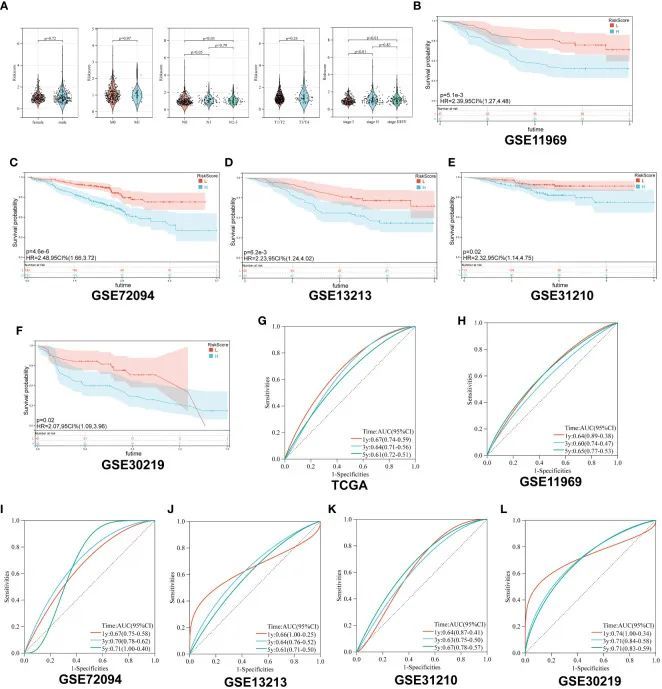
图3 胶原风险模型的生存分析
其次,根据Collagen-Risk评分的中位数,将五个GEO数据集中的患者分为高风险组和低风险组。作者使用五个GEO队列作为外部验证集评估了Collagen-Risk评分在预测OS方面的表现。与TCGA队列的生存分析一样,低风险组的患者在验证数据集中有更长的生存时间(P=0.0051,GSE11969;P<0.001,GSE72094;P=0.0062,GSE12313;P=0.02,GSE31210;P=0.02,GSE30219)(图3B-F)。高风险组的RFS明显降低,低风险组的RFS,这表明Collagen-Risk评分是LUAD患者RFS的优秀预测指标。接下来,为了进一步证明该模型在预测肺癌预后方面的优越性,作者使用ROC曲线评估了Collagen-Risk模型的能力,在六个数据集中,AUC范围从0.60到0.74(图3G-L)。总体而言,时间相关的AUC表明,在TCGA和GEO数据集中,Collagen-Risk评分在预测LUAD患者OS方面具有相当的价值。
4. LUAD中胶原风险模型的诊断价值
使用单变量和多变量Cox回归分析来研究胶原风险模型对预后的预测能力。作者在TCGA和Geo数据集中通过单变量和多变量Cox回归分析评估了胶原风险模型的预后意义。单变量Cox回归分析的结果显示,胶原风险分数与TCGA LUAD队列和5个GEO队列中的OS显著相关(TCGA,HR=2.115,95%CI=1.602-2.792,P<0.001;GSE11969,HR=2.432,95%CI=1.376-4.300,P=0.002;GSE13213,HR=2.514,95%CI=1.413-4.471,P=0.002;GSE30219,HR=2.541,95%CI=1.488-4.342,P<0.001;GSE31210,HR=2.174,95%CI=1.6922.901,P<0.001;GSE372094,HR=2.022,95%CI=1.536-2.663,P<0.001)(图4A)。多变量Cox回归分析证实,胶原风险分数是TCGA LUAD队列和四个GEO队列中OS的重要预测因子(P<0.001,HR=2.49,95%CI=1.618-3.822,TCGA;P=0.013,HR=2.134,95%CI=1.174-3.880,GSE11969;P=0.014,HR=2.339,95%CI=1.188-4.604,GSE13213;P=0.002,HR=2.479,95%CI=1.411-4.356,GSE30219;P<0.001,HR=2.347,95%CI=1.709-3.223,GSE72094)(图4A)。与此同时,作者通过单变量和多变量Cox回归分析,分析了不同数据集中包含的临床特征的预后价值。在TCGA数据集的测试集中,年龄、N分期和风险评分被发现是预后的独立因素。在验证数据集中,年龄和风险评分在单变量和多变量分析中一致被确定为预后的独立预测因子。此外,单变量和多变量Cox回归分析表明,胶原风险评分是RFS的独立预后因子。通过分析TCGA和5个GEO队列的预后结果,进行了一项荟萃分析,以确定胶原风险评分与LUAD患者的OS和RFS的关联和预后意义。通过总体风险比(HR),确认胶原风险评分是LUAD患者OS的危险因素(总体HR = 2.05,95% CI = 1.68-2.49,P < 0.001)(图4B)。同样,胶原风险评分是影响两个GEO队列中RFS的危险因素(总体HR = 3.10,95% CI = 1.96-4.91,P < 0.001)。
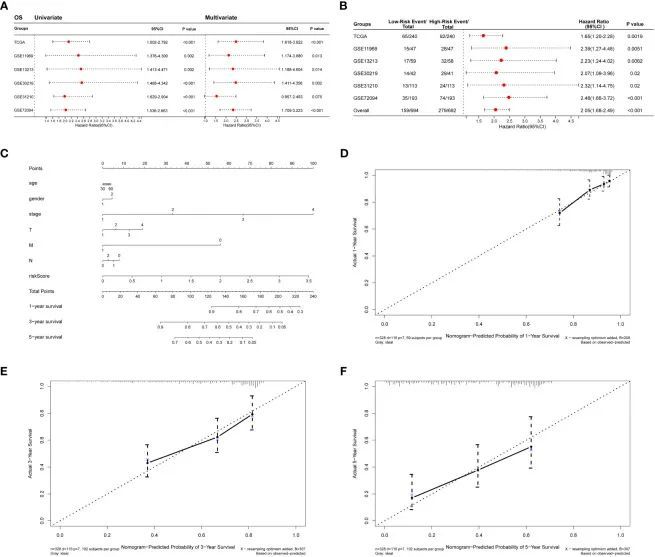
图4 胶原风险模型对LUAD的诊断价值
单变量和多变量Cox分析的结果显示,胶原风险评分与LUAD患者的总生存期(OS)和无复发生存期(RFS)密切相关。随后,作者构建了一个预测患者1年、3年和5年生存概率的诺模图,将胶原风险评分与年龄、性别、T分期、N分期、M分期和肿瘤分期等临床特征结合在一起(图4C)。诺模图的校准曲线显示,预测的生存率与实际1年、3年和5年的生存率密切相关(图4D-F)。
5. 与胶原风险模型相关的生物机制
作者已经证明了Collagen-Risk模型是LUAD患者重要的预后因素,并将进一步研究其对预后的影响机制。作者使用Pearson相关分析筛选出与胶原标志相关的基因(Pearson | R| > 0.5, p < 0.05)。结果表明,59个基因与Collagen-Risk模型显著相关,其中31个基因呈正相关,28个基因呈负相关(图5A)。然后,对这些胶原标志相关基因进行GO和KEGG通路富集分析。这些基因的GO-生物过程(BP)分析显示主要富集在细胞外基质组织、骨骼和心血管系统发育、心血管系统发育(图5B)。细胞组成(CC)的变化主要集中在胶原三聚体、细胞外区域和细胞外基质(图5C)。分子功能(MF)的变化主要集中在蛋白质结合、受体和蛋白酶活性以及细胞外基质结构(图5D)。KEGG信号通路分析显示,这些基因主要参与了细胞外基质受体相互作用、癌症通路、PI3K-Akts和AGE-RAGE信号通路(图5E)。通过GSEA分析,利用TCGA-LUAD患者的转录组数据确定了与胶原风险评分相关的信号转导通路。KEGG分析显示,细胞周期和p53信号通路在高风险组中富集(图5F)。标志物分析表明,上皮间质转化、未折叠蛋白应激、G2M检查点、有丝分裂纺锤体、糖酵解、MTORC1信号通路、E2F靶点、缺氧和血管生成主要富集在高风险组中(图5G)。

图5 与胶原风险模型相关的生物机制
6. 胶原蛋白标记与免疫标记之间的关系
在这项研究中,使用CIBERSORT算法计算了LUAD患者中22种浸润免疫细胞的丰度,并且所有样本中T细胞CD4幼稚型和T细胞Gamma Delta的含量均为0。同时,CIBERSORT结果中的P < 0.05表明样本的免疫浸润分析是可靠的,在淘汰不合格样本后,剩下了333个LUAD样本(图6A、B)。如图6C所示,低风险组和高风险组之间的B细胞记忆型、浆细胞、T细胞CD4记忆型静止型、T细胞CD4记忆型活化型、调节性T细胞(Tregs)、单核细胞、巨噬细胞(M0)、巨噬细胞(M1)、巨噬细胞(M2)、静止型树突状细胞和静止型肥大细胞之间存在显著差异(图6C)。此外,作者还使用ESTIMATE算法计算了每个样本的免疫评分、基质评分、估计评分和肿瘤纯度。作者发现基质评分、估计评分和胶原风险评分与Pearson系数分别为0.26和0.13之间存在正相关。然而,肿瘤纯度和胶原风险评分呈负相关,Pearson系数为-0.14(图6D、F、H、J)。高风险组的免疫评分和基质评分较高,而肿瘤纯度较低(图6E、G、I、K)。
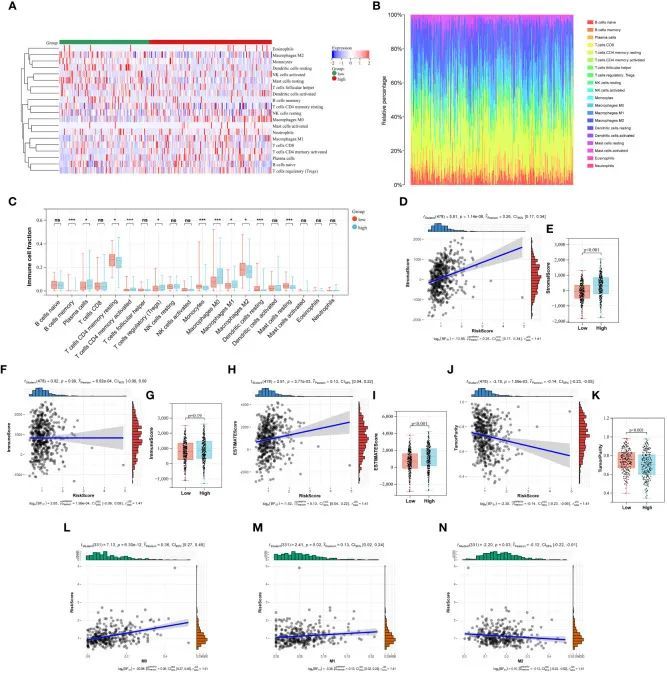
图6 胶原风险评分与免疫特征之间的关系
肿瘤微环境(TME)的概念在癌症研究领域是新的。ECM、与恶性肿瘤相关的成纤维细胞、血管上皮细胞以及浸润的免疫细胞构成了经典的TME。极化的巨噬细胞,包括M1和M2亚型,在最近几年被发现在肿瘤增殖、侵袭和转移中起着重要作用。巨噬细胞非常可塑,并在特定的TME中形成亚群,主要分为M1和M2类型,具有特定的分子和功能特性。M1对于促进肿瘤生长、侵袭、转移以及创造抑制性免疫环境至关重要,因为它能杀死肿瘤细胞并阻止病原体侵入。M2对肿瘤细胞的存活、生长、干细胞特性、侵袭、血管生成和免疫抑制有直接影响。作者深入研究了胶原蛋白标记与巨噬细胞浸润之间的相关性。高风险组和低风险组之间的巨噬细胞渗透水平在图6C中显示出明显差异。接下来,作者发现TCGA队列中TAM浸润与胶原风险评分之间存在联系。M0和M1巨噬细胞浸润与风险评分呈正相关,而M2巨噬细胞浸润呈负相关(图6L,M)。M1的生物标志物包括IL1A、Il1b、IL6、NOS2、TLR2、TLR4、CD80和CD86。M2的生物标志物包括CSF1R、MRC1、PPARG、ARG1、CD163、CLEC10A、RETNLB、PDCD1LG2和CLEC7A。然后,作者检查了胶原风险评分与M1和M2生物标志物之间的关联。M1标志物IL1A、IL6和NOS以及M2标志物RETNLB和PDCD1LG2都与胶原风险评分呈正相关,而TLR2与胶原风险评分呈负相关。因此,胶原特征可能对巨噬细胞浸润和类型具有预测价值。
癌相关成纤维细胞(CAFs)是肿瘤基质的主要组成部分。CAFs以多种方式影响肿瘤细胞,如异常分泌细胞外基质或重塑细胞外基质,分泌细胞因子引起代谢重编程,促进血管生成等。最近的研究发现,CAFs对肿瘤细胞的影响是多样的。生物标志物(FAP,POSTN,PDGFRα/β,FSP-1,CD90,Palladin,OPN,AEBP1,TNC,CD10和GPR77)代表了促癌的CAFs。这些细胞可以通过代谢效应促进肿瘤发展,从而促进血管生成和免疫抑制。然而,Meflin+ CAF和CD146+ CAF与患者的病理组织学特征和预后密切相关。因此,作者研究了这些生物标志物与胶原风险签名之间的关系。相关分析结果显示,风险评分与促癌和抑癌的CAFs生物标志物均呈正相关,表明高风险患者中CAFs的活性高于低风险患者,进一步证实了CAFs在肿瘤进展中的重要性。然而,需要进一步的基础研究来解释这种关系中涉及的机制。
7. 胶原蛋白标志物与免疫治疗相关生物标志物和肿瘤突变负荷之间的关系
近年来,肿瘤免疫疗法已成为癌症研究和治疗领域的热点,并取得了一些有希望和令人鼓舞的成就。对非小细胞肺癌免疫疗法的几项临床研究的初步结果表明,免疫疗法可能为非小细胞肺癌患者带来更好的生存益处。随后,作者研究了35个免疫检查点的表达与胶原风险评分之间的相关性。结果表明,胶原风险评分与CD70、CD200、PDCD1LG2、SIGLEC15、TNFRSF8、TNFRSF9和TNFSF4呈强正相关,与TNFRSF14和TNFSF15呈强负相关(图7A-I)。Wilcoxon检验证实了上述9个免疫检查点在低风险组和高风险组之间的显著差异(图7J)。
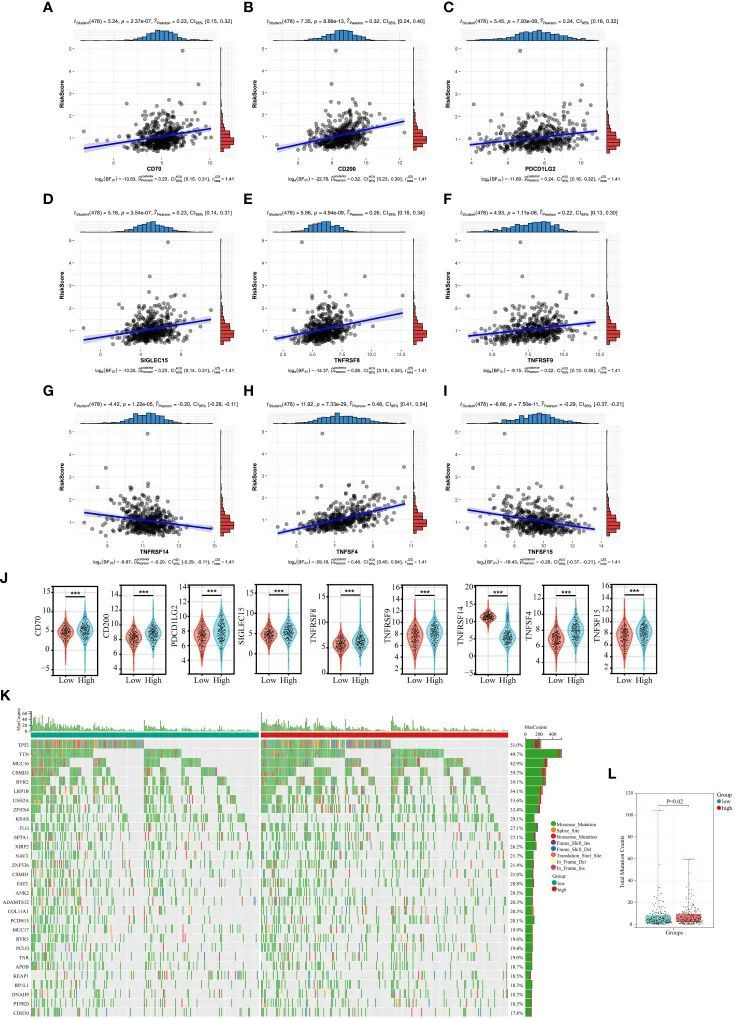
图7 胶原风险评分与免疫治疗相关生物标志物和TMB之间的关系
检查了每个LUAD患者的突变谱。在整个基因组中,前30个最显著改变的基因包括TP53、TTN、MUC16、CSMD3、RYR2、LRP1B、USH2A、ZFHX4、KRAS、FLG、SPTA1、XIRP2、NAV3、ZNF536、CSMD1、FAT3、ANK2、DAMTS12、COL11A1、PCDH15、MUC17、RYR3、PCLO、TNR、APOB、KEAP1、RR1L1、DNAH9、PTPRD、CDH10(图7K)。之后,作者计算了每个样本的TMB,并发现高风险评分组的TMB明显较高(图7L)。
8. 预测基因的实验数据验证
为了验证这些结果,作者检查了人类LUAD标本中五种胶原家族蛋白的转录水平。作者收集了30例正常组织和LUAD组织。结果显示,大多数肿瘤中五种胶原家族蛋白的表达水平比正常组织高得多(图8)。
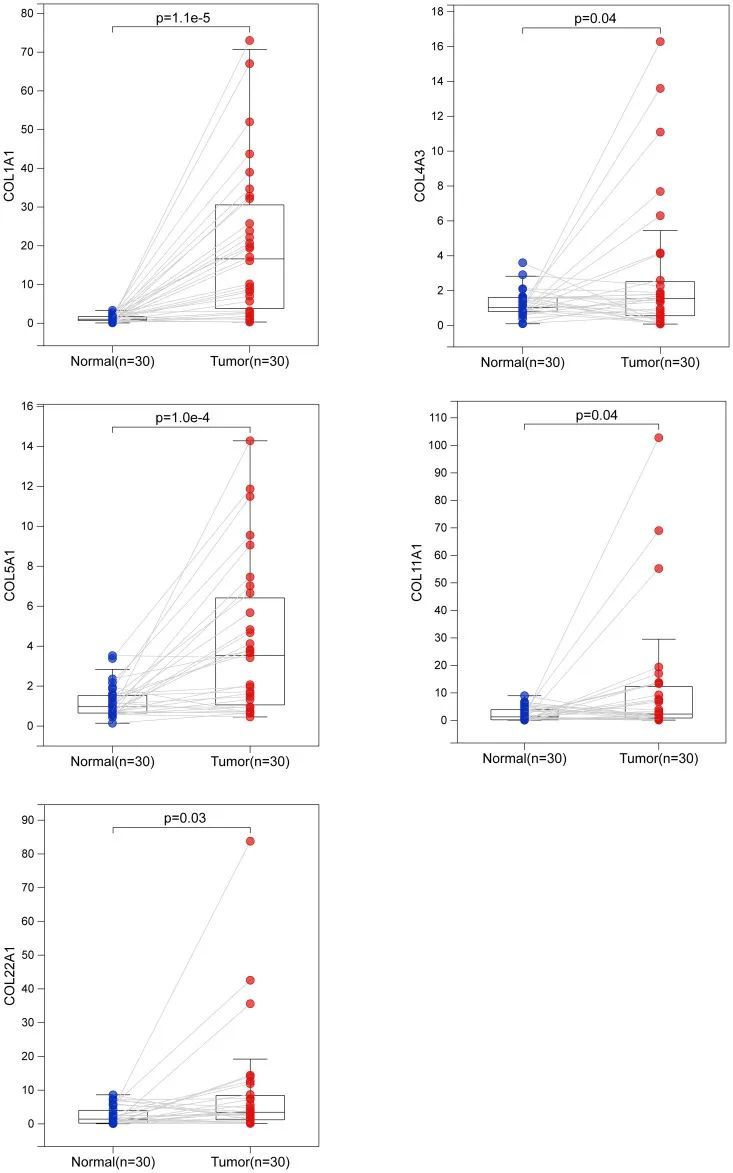
图8 通过定量实时聚合酶链反应对LUAD中5个胶原家族基因的表达进行生物学验证
总结
总结一下,作者在TCGA数据库中确定了一个可靠的预后胶原蛋白标志物。作者首次提出了基于胶原家族成员的预后模型,并详细描述了胶原蛋白标志物与肿瘤微环境之间的关系,这可能为LUAD患者的免疫治疗提供预后信息。这些重要的新发现将使临床医生能够更个体化地治疗LUAD患者。
























 849
849

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?










