一、简介
生存分析是分析生存时间的统计学方法,其因变量需要用生存时间和结局状态两个变量来刻画,可以将终点事件是否发生以及发生终点事件所经历的时间相结合起来。生存分析的主要内容有生存时间的分布描述、生存时间分布的组间比较以及生存时间分布的影响因子的效果评估。
大多数生存分析使用以下方法:
- Kaplan-Meier图可视化生存曲线。
- 对数秩检验以比较两组或更多组的生存曲线间是否存在差异。
- 用Cox比例风险回归描述变量对生存的影响。
二、R实现
1、绘制KM生存曲线
KM(Kaplan-Meier)生存曲线是生存概率与时间的关系图。在R中实现将使用两个R包:
- survival用于计算生存分析
- survminer用于总结和可视化生存分析结果
#安装软件包
install.packages(c("survival", "survminer"))
#加载软件包
library("survival")
library("survminer")
2、示例数据集
使用survival包中提供的肺癌数据:
data(lung) # 加载lung数据集
View(lung) # 查看数据集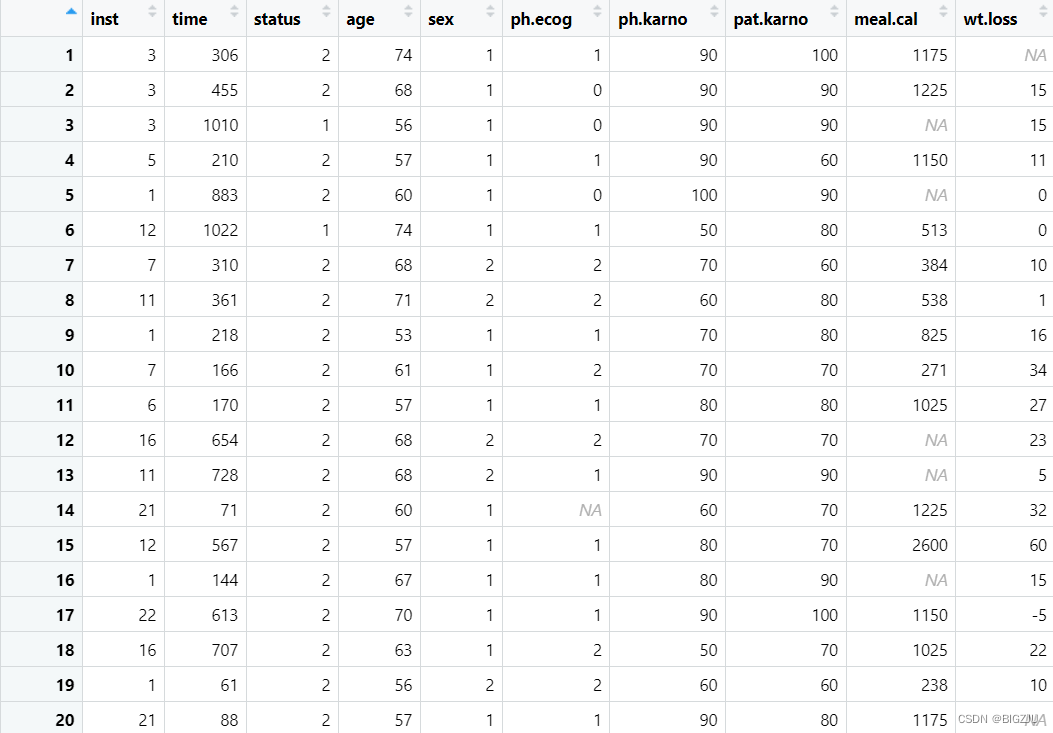 (截取了一部分数据显示)
(截取了一部分数据显示)
- inst:机构代码
- time:以天为单位的生存时间
- status:删失状态1 = 删失,2 = 出现失效事件
- age:岁
- sex:性别,男= 1女= 2
- ph.ecog:ECOG评分(0 =好,5 =死)
- ph.karno:医师进行的Karnofsky评分(0 = 差,100 = 好)
- pat.karno:患者自行进行的Karnofsky评分(0 = 差,100 = 好)
- meal.cal:用餐时消耗的卡路里
- wt.loss:最近六个月的体重减轻
3、计算生存曲线:survfit()
目标:按性别计算生存率。
函数survfit()[在survival包中]可用于计算kaplan-Meier生存估计。主要论包括:
- 使用Surv()函数创建的生存对象
- 数据集中包含了数据内容。
要计算生存曲线,需要输入R代码如下:
fit <- survfit(Surv(time, status) ~ sex, data = lung)
print(fit)
Call: survfit(formula = Surv(time, status) ~ sex, data = lung)
n events median 0.95LCL 0.95UCL
sex=1 138 112 270 212 310
sex=2 90 53 426 348 550默认情况下,函数print()显示生存曲线的简短统计内容。它输入了观察值,事件数,中位生存率和其置信区间。
还可以使用功能surv_summary()获取生存曲线的统计量。与默认的summary()函数相比,surv_summary()创建一个包含来自survfit结果的数据表。
如果要显示生存曲线的更完整统计内容,请键入以下内容:
#看整体的fit结果
res.sum <- surv_summary(fit)
View(res.sum) (截取了一部分数据显示)
(截取了一部分数据显示)
- n:每条曲线中的对象总数。
- time:曲线上的时间点。
- n.riks:在时间t处有风险的受试者人数
- n.event:在时间t发生的事件数
- n.censor:在时间t退出事件而不发生风险的删失者的数量
- lower,upper:曲线的置信度上限和下限。
- strata:表示曲线估计的分层。如果strata不为NULL,则结果中有多条曲线。strata的水平(一个因子)会是曲线的标签。
4、可视化生存曲线
使用功能ggsurvplot()生成两组受试者的生存曲线。它可以显示的内容有:
- 使用参数conf.int = TRUE,显示生存函数的95%置信区间。
- 当选择选项risk.table时,将会显示按时间划分的处于风险中的人的数量和/或百分比。risk.table的允许值包括:
- TRUE或FALSE,指定是否显示风险表。默认值为FALSE。
- “absolute”或“percentage”:分别显示按时间划分处于风险中的受试者的绝对数或百分比。使用“ abs_pct”同时显示绝对数字和百分比。
- 使用参数pval = TRUE,将显示对数秩检验(Log-Rank test)的p值。
- 使用参数surv.median.line,将显示中位生存时间点水平/垂直线。允许的值包括c(“ none”,“ hv”,“ h”,“ v”)之一。v:垂直,h:水平。
#按分层更改图形颜色,线型等
ggsurvplot(fit,
pval = TRUE, conf.int = TRUE,
risk.table = TRUE, # 添加风险表
risk.table.col = "strata", # 根据分层更改风险表颜色
linetype = "strata", # 根据分层更改线型
surv.median.line = "hv", # 同时显示垂直和水平参考线
ggtheme = theme_bw(), # 更改ggplot2的主题
palette = c("#E7B800", "#2E9FDF"))#定义颜色
横轴(x轴)表示以天为单位的时间,纵轴(y轴)表示生存的可能性或生存的人口比例。线代表两组/层的存活曲线。曲线中的垂直下降表示事件。曲线上的十字叉表示此时患者删失。
此外,可以使用参数xlim缩短生存曲线,如下所示:
ggsurvplot(fit,
conf.int = TRUE,
risk.table.col = "strata",
ggtheme = theme_bw(),
palette = c("#E7B800", "#2E9FDF"),
xlim = c(0, 600))若要绘制累积风险函数和累积风险,代码如下:
#绘制累积风险函数
ggsurvplot(fit,
conf.int = TRUE,
risk.table.col = "strata",
ggtheme = theme_bw(),
palette = c("#E7B800", "#2E9FDF"),
fun = "event")
#绘制累积风险
ggsurvplot(fit,
conf.int = TRUE,
risk.table.col = "strata",
ggtheme = theme_bw(),
palette = c("#E7B800", "#2E9FDF"),
fun = "cumhaz")
5、 生存分析的假设检验
对KM生存函数有三种常见检验方法:log-rank、breslow、tarone。这三种都属于卡方检验的方法。不同的是,不同的方法在计算统计量的时候,赋予了不同的权重。
比较生存曲线的对数秩(Log-Rank)检验:survdiff()
对数秩检验是比较两条或更多条生存曲线是否有差异的最常用方法。原假设是两组之间的生存率没有差异。对数秩检验是一种非参数检验,它不对生存分布做出任何假设。本质上,对数秩检验将每个组中观察到的事件数与原假设为真(即,生存曲线相同)下的预期事件进行比较。对数秩统计量近似作为卡方检验统计量分布。
函数survdiff()用于比较两个或多个生存曲线的对数秩检验:
surv_diff <- survdiff(Surv(time, status) ~ sex, data = lung)
surv_diff
Call:
survdiff(formula = Surv(time, status) ~ sex, data = lung)
N Observed Expected (O-E)^2/E (O-E)^2/V
sex=1 138 112 91.6 4.55 10.3
sex=2 90 53 73.4 5.68 10.3
Chisq= 10.3 on 1 degrees of freedom, p= 0.00131 - n:每组的受试者总人数。
- obs:每组中事件观测到的数量。
- exp:每个组中预期事件数。
- chisq:用于卡方检验统计量。
- strata:(可选)每个层中包含的主题数。
生存差异的对数秩检验得出p值为p = 0.0013,表明性别群体的生存差异显着。
6、基于Cox比例风险模型的生存分析
# 基于Cox比例风险模型从年龄和医学评分来预测男性的生存情况
MaleMod <- coxph(survobj~age+ph.ecog+ph.karno+pat.karno, data=lung, subset=sex==1)
MaleMod # 输出结果
Call:
coxph(formula = Surv(time, status) ~ age + ph.ecog + ph.karno +
pat.karno, data = lung, subset = sex == 1)
coef exp(coef) se(coef) z p
age 0.022465 1.022719 0.012216 1.839 0.0659
ph.ecog 0.665452 1.945370 0.225712 2.948 0.0032
ph.karno 0.025553 1.025883 0.011778 2.170 0.0300
pat.karno -0.011059 0.989002 0.008892 -1.244 0.2136
Likelihood ratio test=17.87 on 4 df, p=0.001311
n= 134, number of events= 108
(4 observations deleted due to missingness)从运行结果可以看出,ph.ecog和ph.karno对生存率的影响是显著的,而其他变量则并不显著。























 1282
1282

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








