-
一、引物设计中酶切位点的保护碱基总结
保护碱基的作用
1. 提高酶切效率
在分子克隆实验中,有时我们会在待扩增的目的基因片段两端加上特定的酶切位点,用于后续的酶切和连接反应。由于直接暴露在末端的酶切位点不容易直接被限制性核酸内切酶切开,因此在设计PCR引物时,人为地在酶切位点序列的5'端外侧添加额外的碱基序列,即保护碱基,用来提高将来酶切时的活性。
2. 避免相邻酶切位点的干扰
在分子克隆实验中选择载体的酶切位点时,相邻的两个酶切位点往往不能同时使用,因为一个位点切割后留下的碱基过少以至于影响旁边的酶切位点切割。保护碱基的添加可以避免这种情况的发生。
3. 增加酶切位点的稳定性
保护碱基可以增加酶切位点的稳定性,使其更容易被限制性核酸内切酶识别和切割。
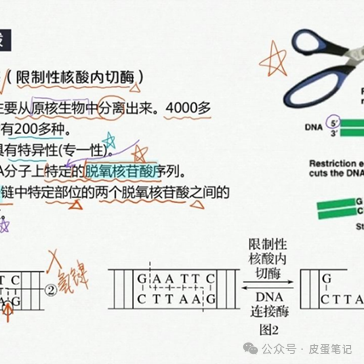
图1.限制性内切酶
4. 提高PCR扩增效率
在PCR扩增过程中,保护碱基可以帮助引物更好地结合到模板DNA上,从而提高扩增效率。这对于获得足够数量的目的基因片段非常重要。
-
二、添加保护碱基的基本原则
1.考虑酶切位点:如果引物设计中包含了酶切位点,那么在酶切位点的5'端外侧添加额外的碱基序列,即保护碱基123,如CGC。
2.保护碱基的数目:添加保护碱基时,最关心的应该是保护碱基的数目,而不是种类。什么样的酶切位点,添加几个保护碱基。
3.根据Tm值和GC含量调整:如果某条引物Tm值偏小,GC%较低,添加时多加G或C,反之亦反。
4.避免连续相同的碱基:特别是连续的G出现在引物中4。
5.引物长度:保护碱基引物的长度通常为15-25个碱基对6。
6.引物序列:引物应根据酶切位点的序列设计。为了确保引物的特异性,通常将酶切位点和其周围的碱基考虑在内。
7.引物组成:引物的核苷酸组成应考虑碱基的GC含量,以保持引物的稳定性。通常,GC含量高于50%的引物更稳定。
8.引物末端修饰:引物的末端修饰可以提高引物与目标DNA的亲和性,并增加引物的稳定性。常用的末端修饰包括磷酸化和胺基修饰等。
酶切位点的保护碱基总结表
没有列出的酶,则通常需在识别位点两端至少加上6个保护碱基
黑色:保护碱基,红色:内切酶序列
| 酶 | 寡核苷酸序列 | 酶切效率 | |
| 2 h | 20 h | ||
| Acc I | GGTCGACC CGGTCGACCG CCGGTCGACCGG | 0 0 0 | 0 0 0 |
| Afl III | CACATGTG CCACATGTGG CCCACATGTGGG | 0 >90 >90 | 0 >90 >90 |
| Asc I | GGCGCGCC AGGCGCGCCT TTGGCGCGCCAA | >90 >90 >90 | >90 >90 >90 |
| Ava I | CCCCGGGG CCCCCGGGGG TCCCCCGGGGGA | 50 >90 >90 | >90 >90 >90 |
| BamH I | CGGATCCG CGGGATCCCG CGCGGATCCGCG | 10 >90 >90 | 25 >90 >90 |
| Bgl II | CAGATCTG GAAGATCTTC GGAAGATCTTCC | 0 75 25 | 0 >90 >90 |
| BssH II | GGCGCGCC AGGCGCGCCT TTGGCGCGCCAA | 0 0 50 | 0 0 >90 |
| BstE II | GGGT(A/T)ACCC | 0 | 10 |
| BstX I | AACTGCAGAACCAATGCATTGG AAAACTGCAGCCAATGCATTGGAA CTGCAGAACCAATGCATTGGATGCAT | 0 25 25 | 0 50 >90 |
| Cla I | CATCGATG GATCGATC CCATCGATGG CCCATCGATGGG | 0 0 >90 50 | 0 0 >90 50 |
| EcoR I | GGAATTCC CGGAATTCCG CCGGAATTCCGG | >90 >90 >90 | >90 >90 >90 |
| Hae III | GGGGCCCC AGCGGCCGCT TTGCGGCCGCAA | >90 >90 >90 | >90 >90 >90 |
| Hind III | CAAGCTTG CCAAGCTTGG CCCAAGCTTGGG | 0 0 10 | 0 0 75 |
| Kpn I | GGGTACCC GGGGTACCCC CGGGGTACCCCG | 0 >90 >90 | 0 >90 >90 |
| Mlu I | GACGCGTC CGACGCGTCG | 0 25 | 0 50 |
| Nco I | CCCATGGG CATGCCATGGCATG | 0 50 | 0 75 |
| Nde I | CCATATGG CCCATATGGG CGCCATATGGCG GGGTTTCATATGAAACCC GGAATTCCATATGGAATTCC GGGAATTCCATATGGAATTCCC | 0 0 0 0 75 75 | 0 0 0 0 >90 >90 |
| Nhe I | GGCTAGCC CGGCTAGCCG CTAGCTAGCTAG | 0 10 10 | 0 25 50 |
| Not I | TTGCGGCCGCAA ATTTGCGGCCGCTTTA AAATATGCGGCCGCTATAAA ATAAGAATGCGGCCGCTAAACTAT AAGGAAAAAAGCGGCCGCAAAAGGAAAA | 0 10 10 25 25 | 0 10 10 90 >90 |
| Nsi I | TGCATGCATGCA CCAATGCATTGGTTCTGCAGTT | 10 >90 | >90 >90 |
| Pac I | TTAATTAA GTTAATTAAC CCTTAATTAAGG | 0 0 0 | 0 25 >90 |
| Pme I | GTTTAAAC GGTTTAAACC GGGTTTAAACCC AGCTTTGTTTAAACGGCGCGCCGG | 0 0 0 75 | 0 25 50 >90 |
| Pst I | GCTGCAGC TGCACTGCAGTGCA AACTGCAGAACCAATGCATTGG AAAACTGCAGCCAATGCATTGGAA CTGCAGAACCAATGCATTGGATGCAT | 0 10 >90 >90 0 | 0 10 >90 >90 0 |
| Pvu I | CCGATCGG ATCGATCGAT TCGCGATCGCGA | 0 10 0 | 0 25 10 |
| Sac I | CGAGCTCG | 10 | 10 |
| Sac II | GCCGCGGC TCCCCGCGGGGA | 0 50 | 0 >90 |
| Sal I | GTCGACGTCAAAAGGCCATAGCGGCCGC GCGTCGACGTCTTGGCCATAGCGGCCGCGG ACGCGTCGACGTCGGCCATAGCGGCCGCGGAA | 0 10 10 | 0 50 75 |
| Sca I | GAGTACTC AAAAGTACTTTT | 10 75 | 25 75 |
| Sma I | GACTAGTC GGACTAGTCC CGGACTAGTCCG CTAGACTAGTCTAG | 0 0 10 >90 | 10 10 50 >90 |
| Spe I | GACTAGTC GGACTAGTCC CGGACTAGTCCG CTAGACTAGTCTAG | 10 10 0 0 | >90 >90 50 50 |
| Sph I | GGCATGCC CATGCATGCATG ACATGCATGCATGT | 0 0 10 | 0 25 50 |
| Stu I | AAGGCCTT GAAGGCCTTC AAAAGGCCTTTT | >90 >90 >90 | >90 >90 >90 |
| Xba I | CTCTAGAG GCTCTAGAGC TGCTCTAGAGCA CTAGTCTAGACTAG | 0 >90 75 75 | 0 >90 >90 >90 |
| Xho I | CCTCGAGG CCCTCGAGGG CCGCTCGAGCGG | 0 10 10 | 0 25 75 |
| Xma I | CCCCGGGG CCCCCGGGGG CCCCCCGGGGGG TCCCCCCGGGGGGA | 0 25 50 >90 | 0 75 >90 >90 |
























 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








