质粒构建和引物设计是分子生物学中常见的实验操作,以下是详细的步骤和注意事项:
1. 质粒构建的基本流程**
(1)目标序列的获取
从基因库获取:如NCBI、ENSEMBL等。
人工设计:根据实验需求设计编码序列(如添加标签序列、突变位点等)。
(2)载体的选择
表达载体:常用于蛋白表达,如pET、pcDNA系列。
克隆载体:用于扩增,如pUC19。
选择时需考虑:
启动子类型(如CMV、T7)。
抗性标记(如Amp、Kan)。
多克隆位点(MCS)。
(3)构建策略
酶切连接:通过限制性内切酶切割目标序列和载体,随后连接。
-Gibson Assembly:多片段定向连接,适合无酶切位点或复杂构建。
-重组克隆:如使用Gateway、In-Fusion克隆试剂盒。
- PCR克隆:直接扩增目的片段并插入载体。
2. 引物设计的步骤**
引物设计是质粒构建中的关键环节,以下是详细步骤:
(1)基本要求
长度*:18-25 bp。
GC含量:40%-60%,避免过高或过低。
Tm值:55°C-65°C(上下游引物的Tm值差异小于2°C)。
特异性:避免形成二级结构(如发夹结构、自互补)。
引物末端:3'末端尽量以G或C结尾,提高扩增效率。
(2)添加特殊序列
酶切位点:引物5'端添加合适的酶切位点,同时加上3-6 bp的保护碱基。
标签序列:如FLAG、His-tag,或突变设计。
(3)工具和软件
在线工具:
- [Primer3](https://primer3.ut.ee/):经典引物设计工具。
- [SnapGene](https://www.snapgene.com/):便捷的克隆设计工具。
- [Benchling](https://www.benchling.com/):多功能分子生物学工具。
-分析软件:用OligoAnalyzer分析潜在的二级结构和自互补性。
以Nrf2为列对引物设计及质粒构建步骤进行阐述:
1. 打开NCBI官网:National Center for Biotechnology Information




2. 打开TaKaRa引物设计网址:Primer design and other tools




把整个质粒复制到里面。

选上1,然后在2部分搜酶切位点,选中3,把第一步的目的基因粘贴到4(我一般会在目的基因前面加个GCCACC(Kozak 序列),这样可以增加蛋白产量,如果目的基因后面连接了tag,就把最后三个终止密码子删掉,没有的话就不需要),然后点5。

下载下来,解压,分别为构建好的质粒跟引物。


打开输入特征中的第一个

打开骨架质粒

添加特征

选中你的目的基因,特征-添加特征-命名Nrf2
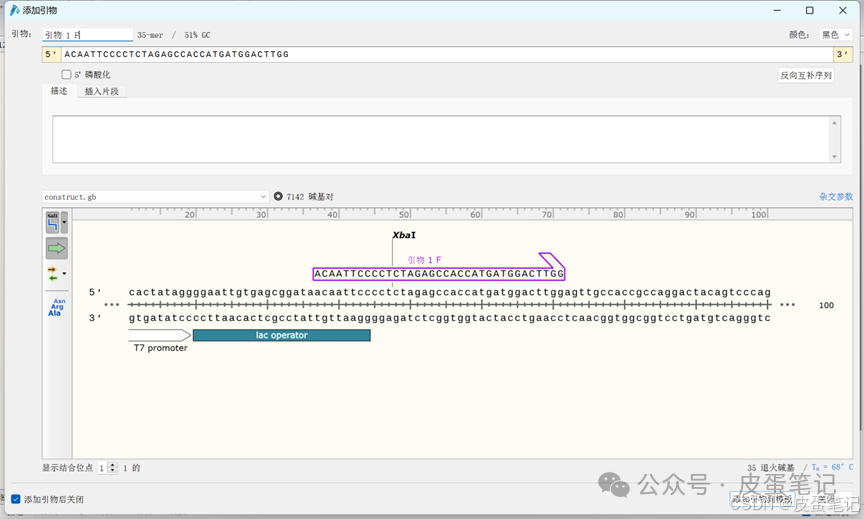
点引物-添加引物,复制表中的引物,分别添加F和R引物。
添加完后点最底下的引物,后点最上面的行动,选聚合酶链反应(PCR)


把F和R分别选到引物1和2,然后点右下角聚合酶链反应(PCR),会生成一个新的DNA文件,这个就是模拟PCR后的产物,保存。

点红框,然后点比对,把刚刚保存的PCR产物选上,就会出现下图结果。

滑动比较看是否有错误,没有错误(好像是红色小框)就可以订引物进行质粒构建了。最后可以自己改一下名字。

以上就是在质粒构建中,引物设计及质粒重构的操作教程,最后非常感谢北京大学吴博士(WSH)分享的质粒重构教程,祝吴博士科研顺利,CNS文章发发发!!!
文章转载自公众号:皮蛋笔记,欢迎关注,随时获取第一手文章内容。























 1万+
1万+

 被折叠的 条评论
为什么被折叠?
被折叠的 条评论
为什么被折叠?








